Encuentran mutación que determina la naturaleza de la respuesta del huésped a la infección por SARM
|
Por el equipo editorial de LabMedica en español Actualizado el 01 Oct 2019 |
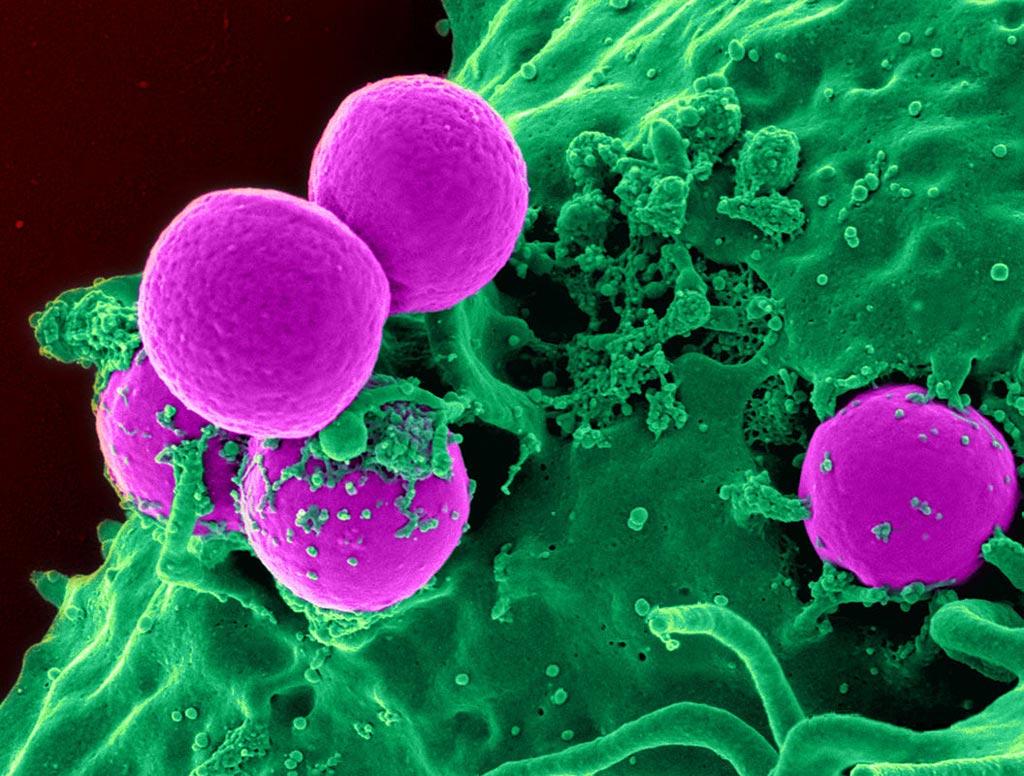
Imagen: Una microfotografía electrónica de barrido (SEM) de neutrófilos humanos ingiriendo un SARM (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los EUA).
Un artículo reciente describió los factores genéticos que determinan por qué algunas personas desarrollan infecciones crónicas por Staphylococcus aureus resistente a la meticilina (SARM), mientras que otras desarrollan infecciones que se resuelven relativamente rápido.
La gravedad y la duración de la infección por SARM varían ampliamente entre los individuos. Los factores del huésped que predisponen a la infección persistente por SARM son poco conocidos, aunque los estudios de asociación genética ya pudieron identificar variantes potencialmente influyentes.
Los investigadores de la Universidad de Duke (Durham, NC, EUA) buscaron dichos factores del huésped analizando dos conjuntos de pacientes que habían sido relacionados estrechamente por edad, género, condiciones de salud y otros factores de riesgo, para las infecciones del torrente sanguíneo por SARM. Sesenta y ocho pacientes fueron incluidos en el estudio; la mitad con una infección persistente por SARM y la otra mitad que pudo eliminar la infección del torrente sanguíneo. Se utilizó la secuenciación del exoma completo para identificar las diferencias genómicas entre los dos conjuntos de pacientes.
Los resultados revelaron que una mutación ubicada en el ADN de la región DNMT3A del cromosoma 2p se expresó en aproximadamente el 62% de los pacientes que eliminaron su infección por SARM, mientras que se expresó en solo el 9% en aquellos que tenían infecciones persistentes.
En otra serie de experimentos, los investigadores demostraron que las variantes de DNMT3A podían alterar la respuesta del huésped a la infección a través del aumento de la metilación de genes reguladores clave, lo que resultó en una producción reducida de interleuquina-10 y, a su vez, permitió una respuesta inmune más protectora que eliminó la infección.
“La creciente prevalencia de infecciones por estafilococos resistentes a los antibióticos ha creado una necesidad urgente de comprender mejor quién es más susceptible a estas infecciones por S. aureus difíciles de tratar y por qué”, dijo el autor principal, el Dr. Vance Fowler, profesor de medicina, genética molecular y microbiología en la Universidad de Duke. “Nuestro estudio identifica una mutación particular de DNMT3A que contribuye a una mayor capacidad para resolver las infecciones por SARM. El mecanismo para esto parece ser una mayor metilación de las regiones reguladoras de genes y niveles más bajos de la citoquina antiinflamatoria IL-10”.
El artículo sobre el SARM se publicó en la edición en línea del 16 de septiembre de 2019 de la revista Proceedings of the [U.S.] National Academy of Sciences.
Enlace relacionado:
Universidad de Duke
La gravedad y la duración de la infección por SARM varían ampliamente entre los individuos. Los factores del huésped que predisponen a la infección persistente por SARM son poco conocidos, aunque los estudios de asociación genética ya pudieron identificar variantes potencialmente influyentes.
Los investigadores de la Universidad de Duke (Durham, NC, EUA) buscaron dichos factores del huésped analizando dos conjuntos de pacientes que habían sido relacionados estrechamente por edad, género, condiciones de salud y otros factores de riesgo, para las infecciones del torrente sanguíneo por SARM. Sesenta y ocho pacientes fueron incluidos en el estudio; la mitad con una infección persistente por SARM y la otra mitad que pudo eliminar la infección del torrente sanguíneo. Se utilizó la secuenciación del exoma completo para identificar las diferencias genómicas entre los dos conjuntos de pacientes.
Los resultados revelaron que una mutación ubicada en el ADN de la región DNMT3A del cromosoma 2p se expresó en aproximadamente el 62% de los pacientes que eliminaron su infección por SARM, mientras que se expresó en solo el 9% en aquellos que tenían infecciones persistentes.
En otra serie de experimentos, los investigadores demostraron que las variantes de DNMT3A podían alterar la respuesta del huésped a la infección a través del aumento de la metilación de genes reguladores clave, lo que resultó en una producción reducida de interleuquina-10 y, a su vez, permitió una respuesta inmune más protectora que eliminó la infección.
“La creciente prevalencia de infecciones por estafilococos resistentes a los antibióticos ha creado una necesidad urgente de comprender mejor quién es más susceptible a estas infecciones por S. aureus difíciles de tratar y por qué”, dijo el autor principal, el Dr. Vance Fowler, profesor de medicina, genética molecular y microbiología en la Universidad de Duke. “Nuestro estudio identifica una mutación particular de DNMT3A que contribuye a una mayor capacidad para resolver las infecciones por SARM. El mecanismo para esto parece ser una mayor metilación de las regiones reguladoras de genes y niveles más bajos de la citoquina antiinflamatoria IL-10”.
El artículo sobre el SARM se publicó en la edición en línea del 16 de septiembre de 2019 de la revista Proceedings of the [U.S.] National Academy of Sciences.
Enlace relacionado:
Universidad de Duke
Últimas Microbiología noticias
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
- Prueba CRISPR-TB permite diagnóstico temprano de enfermedad y cribado de la población
- Panel sindrómico ofrece respuestas rápidas para diagnóstico ambulatorio de enfermedades gastrointestinales
Canales
Química Clínica
ver canal
Herramienta química a nanoescala 'brillantemente luminosa' mejora detección de enfermedades
Miles de moléculas brillantes disponibles comercialmente, conocidas como fluoróforos, se utilizan comúnmente en imágenes médicas, detección de enfermedades, marcado... Más
Prueba de detección portátil económica transforma detección de enfermedades renales
Millones de personas padecen enfermedad renal, que a menudo permanece sin diagnosticar hasta que alcanza una etapa crítica. Esta epidemia silenciosa no solo disminuye la calidad de vida de los afectados,... MásDiagnóstico Molecular
ver canal
Prueba de biomarcadores sanguíneos podría detectar predisposición genética al Alzheimer
Nuevos medicamentos para la enfermedad de Alzheimer, la forma más común de demencia, están ahora disponibles. Estos tratamientos, conocidos como "anticuerpos amiloides",... Más
Se descubre nuevo autoanticuerpo contra DAGLA en cerebelitis
Las ataxias cerebelosas autoinmunes son trastornos muy incapacitantes que se caracterizan por una disminución de la habilidad para coordinar el movimiento muscular. Los autoanticuerpos cerebelosos... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Nuevo método basado en aprendizaje automático detecta contaminación microbiana en cultivos celulares
La terapia celular tiene un gran potencial en el tratamiento de enfermedades como el cáncer, las enfermedades inflamatorias y los trastornos degenerativos crónicos mediante la manipulación o el reemplazo... Más
Nuevo método con corrección de errores detecta cáncer únicamente en muestras de sangre
La tecnología de biopsia líquida, que se basa en análisis de sangre para la detección temprana del cáncer y el seguimiento de la carga oncológica en los pacientes,... MásTecnología
ver canal
Tecnología de microchip desechable podría detectar selectivamente VIH en muestras de sangre completa
A finales de 2023, aproximadamente 40 millones de personas en todo el mundo vivían con VIH, y alrededor de 630.000 personas murieron por enfermedades relacionadas con el sida ese mismo año.... Más
Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
El dolor crónico es una afección generalizada que sigue siendo difícil de controlar, y los métodos clínicos existentes para su tratamiento se basan en gran medida en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más