Cambios en la carga bacteriana y los niveles de citoquinas en suero predicen la probabilidad de morir por sepsis
|
Por el equipo editorial de LabMedica en español Actualizado el 01 Jun 2020 |
Imagen: Representación en dibujos animados de la estructura molecular de la proteína blaTEM (beta-lactamasa) (Fotografía cortesía de Wikimedia Commons)
Se puede utilizar un método de análisis para la cuantificación sensible y rápida de las citoquinas séricas y la carga bacteriana para predecir la probabilidad de que un paciente muera por sepsis o shock séptico.
La sepsis es causada por una respuesta inmune inflamatoria desencadenada por una infección. Es una afección potencialmente mortal que surge cuando la respuesta del cuerpo a la infección causa daño a sus propios tejidos y órganos. Los signos y síntomas comunes incluyen fiebre, aumento de la frecuencia cardíaca, aumento de la frecuencia respiratoria y confusión. También puede haber síntomas relacionados con una infección específica, como tos en caso de neumonía o dolor al orinar cuando hay una infección renal. En los muy jóvenes, ancianos y aquellos con un sistema inmune debilitado, puede no haber síntomas de una infección específica y la temperatura corporal puede ser baja o normal, en lugar de alta.
Para facilitar el diagnóstico, el seguimiento y el tratamiento de enfermedades infecciosas como las responsables de la sepsis, los investigadores de la Universidad de Chicago (IL, EUA) desarrollaron un método de cuantificación sensible y rápido para la carga bacteriana y las citoquinas séricas a partir de muestras biológicas humanas.
El método utiliza ensayos digitales de ligadura de proximidad (dPLA) para cuantificar marcadores de ácidos nucleicos y proteínas. La lectura de la PCR digital por gotitas (ddPCR), en el protocolo PLA, permitió la medición simultánea de genes 16S rARN específicos de Gram negativos (GN) y Gram positivos (GP) (que reflejan la abundancia de todas las bacterias GN y GP en las muestras de los pacientes) y del gen blaTEM (Beta-lactamasa) (que induce resistencia a los antibióticos Beta-lactámicos) junto con los niveles de proteína IL-6 y TNF-alfa en la misma muestra de los pacientes.
Una ventaja importante de este método de amplificación digital es su capacidad para cuantificar cambios muy pequeños en la concentración de estas moléculas. La ddPCR tiene una resolución de una molécula de ADN único en muestras, y los investigadores pudieron lograr una resolución subfemtomolar para los objetivos de proteínas.
Para demostrar el potencial de este enfoque, los investigadores lo utilizaron primero para analizar muestras de líquido de lavado broncoalveolar (BALF) de pacientes con asma leve a grave, y encontraron que los pacientes con asma tenían niveles más altos de bacterias GN e IL-6 que los individuos de control sanos. Luego utilizaron los ensayos para caracterizar longitudinalmente muestras de plasma de pacientes con shock séptico, revelando varias características moleculares asociadas con la recuperación o la muerte. Los análisis mostraron que los cambios en el tiempo de varios biomarcadores, y no sus concentraciones absolutas, fueron predictores confiables de los resultados de los pacientes. La aplicación del análisis del árbol de decisión a los resultados obtenidos por este método permitió la predicción de la mortalidad del paciente/tasa de recuperación del shock séptico con más del 90% de exactitud.
“Nuestros hallazgos brindan un método nuevo para el diagnóstico de sepsis con el potencial de identificar temprano el patógeno causal”, dijo el autor colaborador, Dr. Gokhan Mutlu, profesor de medicina de la Universidad de Chicago. “Esto nos permitirá usar los antibióticos apropiados antes de que los resultados del cultivo estén disponibles y minimizar el uso de antibióticos necesarios para tratar la infección. Al combinar los datos relacionados con el patógeno y la respuesta del huésped, podemos predecir los resultados en los pacientes con sepsis”.
“Se necesita una prueba rápida como esta en muchas situaciones y realmente podría cambiar el juego para el tratamiento de la sepsis”, dijo el autor principal, el Dr. Savas Tay, profesor asociado de ingeniería molecular en la Universidad de Chicago. “Esta es una enfermedad que puede matar a todos, independientemente de su situación”.
La prueba rápida para la cuantificación digital de citoquinas y bacterias se describió en la edición en línea del 25 de mayo de 2020 de la revista Nature Communications.
Enlace relacionado:
Universidad de Chicago
La sepsis es causada por una respuesta inmune inflamatoria desencadenada por una infección. Es una afección potencialmente mortal que surge cuando la respuesta del cuerpo a la infección causa daño a sus propios tejidos y órganos. Los signos y síntomas comunes incluyen fiebre, aumento de la frecuencia cardíaca, aumento de la frecuencia respiratoria y confusión. También puede haber síntomas relacionados con una infección específica, como tos en caso de neumonía o dolor al orinar cuando hay una infección renal. En los muy jóvenes, ancianos y aquellos con un sistema inmune debilitado, puede no haber síntomas de una infección específica y la temperatura corporal puede ser baja o normal, en lugar de alta.
Para facilitar el diagnóstico, el seguimiento y el tratamiento de enfermedades infecciosas como las responsables de la sepsis, los investigadores de la Universidad de Chicago (IL, EUA) desarrollaron un método de cuantificación sensible y rápido para la carga bacteriana y las citoquinas séricas a partir de muestras biológicas humanas.
El método utiliza ensayos digitales de ligadura de proximidad (dPLA) para cuantificar marcadores de ácidos nucleicos y proteínas. La lectura de la PCR digital por gotitas (ddPCR), en el protocolo PLA, permitió la medición simultánea de genes 16S rARN específicos de Gram negativos (GN) y Gram positivos (GP) (que reflejan la abundancia de todas las bacterias GN y GP en las muestras de los pacientes) y del gen blaTEM (Beta-lactamasa) (que induce resistencia a los antibióticos Beta-lactámicos) junto con los niveles de proteína IL-6 y TNF-alfa en la misma muestra de los pacientes.
Una ventaja importante de este método de amplificación digital es su capacidad para cuantificar cambios muy pequeños en la concentración de estas moléculas. La ddPCR tiene una resolución de una molécula de ADN único en muestras, y los investigadores pudieron lograr una resolución subfemtomolar para los objetivos de proteínas.
Para demostrar el potencial de este enfoque, los investigadores lo utilizaron primero para analizar muestras de líquido de lavado broncoalveolar (BALF) de pacientes con asma leve a grave, y encontraron que los pacientes con asma tenían niveles más altos de bacterias GN e IL-6 que los individuos de control sanos. Luego utilizaron los ensayos para caracterizar longitudinalmente muestras de plasma de pacientes con shock séptico, revelando varias características moleculares asociadas con la recuperación o la muerte. Los análisis mostraron que los cambios en el tiempo de varios biomarcadores, y no sus concentraciones absolutas, fueron predictores confiables de los resultados de los pacientes. La aplicación del análisis del árbol de decisión a los resultados obtenidos por este método permitió la predicción de la mortalidad del paciente/tasa de recuperación del shock séptico con más del 90% de exactitud.
“Nuestros hallazgos brindan un método nuevo para el diagnóstico de sepsis con el potencial de identificar temprano el patógeno causal”, dijo el autor colaborador, Dr. Gokhan Mutlu, profesor de medicina de la Universidad de Chicago. “Esto nos permitirá usar los antibióticos apropiados antes de que los resultados del cultivo estén disponibles y minimizar el uso de antibióticos necesarios para tratar la infección. Al combinar los datos relacionados con el patógeno y la respuesta del huésped, podemos predecir los resultados en los pacientes con sepsis”.
“Se necesita una prueba rápida como esta en muchas situaciones y realmente podría cambiar el juego para el tratamiento de la sepsis”, dijo el autor principal, el Dr. Savas Tay, profesor asociado de ingeniería molecular en la Universidad de Chicago. “Esta es una enfermedad que puede matar a todos, independientemente de su situación”.
La prueba rápida para la cuantificación digital de citoquinas y bacterias se describió en la edición en línea del 25 de mayo de 2020 de la revista Nature Communications.
Enlace relacionado:
Universidad de Chicago
Últimas Microbiología noticias
- Nuevos ensayos de hepatitis con marcado CE permite la detección temprana de infecciones
- Prueba de PCR múltiplex identifica el 95 % de los patógenos que causan la sepsis en una hora
- Prueba de bacterias bucales podría predecir la progresión del cáncer de colon
- Firma metabólica unica podría permitir el diagnóstico de sepsis dentro de una hora de la extracción de sangre
- Innovadora plataforma de diagnóstico proporciona resultados de AST con velocidad sin precedentes
- Análisis de sangre predice sepsis e insuficiencia orgánica en niños
- Análisis de sangre para tuberculosis podría detectar millones de propagadores silenciosos
- Un análisis de sangre simple combinado con un modelo de riesgo personalizado mejora el diagnóstico de sepsis
- Nuevo análisis de sangre reduce tiempo de diagnóstico de infecciones por micobacterias no tuberculosas de meses a horas
- Nuevo análisis para tuberculosis podría ampliar acceso a pruebas en países de ingresos bajos y medios
- Prueba rápida diagnostica enfermedades tropicales en horas para tratamiento con antibióticos más rápido
- Pruebas moleculares rápidas permiten tratamiento antibiótico más rápido y específico para neumonía
- Plataforma rápida de PSA proporciona resultados terapéuticos específicos días antes que el estándar de atención actual
- Nuevo método de análisis detecta patógenos en sangre de forma más rápida y precisa al fundir ADN
- Prueba rápida de sepsis ofrece resultados dos días más rápidos
- Diagnóstico rápido portátil por PCR podría detectar gonorrea y susceptibilidad a antibióticos
Canales
Química Clínica
ver canal
Espectrómetro de masas impreso en 3D para el punto de atención supera a los modelos de última generación
La espectrometría de masas es una técnica precisa para identificar los componentes químicos de una muestra y tiene un potencial significativo para monitorear estados de salud de enfermedades... Más.jpg)
Prueba biomédica POC hace girar una gota de agua utilizando ondas sonoras para detección del cáncer
Los exosomas, pequeñas biopartículas celulares que transportan un conjunto específico de proteínas, lípidos y materiales genéticos, desempeñan un papel... Más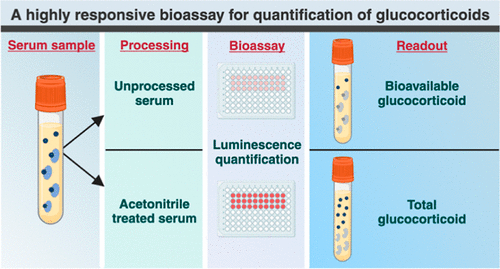
Prueba basada en células altamente confiable permite diagnóstico preciso de enfermedades endocrinas
Los métodos convencionales para medir el cortisol libre, la hormona del estrés del cuerpo, en la sangre o la saliva son bastante exigentes y requieren el procesamiento de muestras. Por lo tanto, el método... MásDiagnóstico Molecular
ver canal
Prueba de sangre predice con precisión el riesgo de cáncer de pulmón y reduce la necesidad de escaneos de TC
El cáncer de pulmón es extremadamente difícil de detectar tempranamente debido a las limitaciones de las tecnologías de detección actuales, que son costosas, a veces... Más
Firma única de autoanticuerpos ayuda a diagnosticar la esclerosis múltiple años antes de la aparición de síntomas
Se cree que las enfermedades autoinmunes como la esclerosis múltiple (EM) ocurren en parte debido a respuestas inmunes inusuales a infecciones comunes. Los primeros síntomas de la EM, incluidos... Más
Prueba de sangre podría detectar cánceres asociados al VPH 10 años antes del diagnóstico clínico
Se sabe que el virus del papiloma humano (VPH) causa varios cánceres, incluidos los de genitales, ano, boca, garganta y cuello uterino. El cáncer orofaríngeo asociado al VPH (VPH+COF)... Más
Un diagnóstico de bajo costo en el punto de atención ampliará el acceso a pruebas de enfermedades de transmisión sexual
La gonorrea es la segunda infección bacteriana de transmisión sexual (ITS) más comúnmente reportada, con alrededor de 82 millones de casos en todo el mundo en 2020. La infección puede tener graves consecuencias... MásHematología
ver canal
Primera prueba NAT 4 en 1 para el cribado de arbovirus podría reducir el riesgo de infecciones transmitidas por transfusiones
Los arbovirus representan una amenaza emergente para la salud mundial, exacerbada por el cambio climático y el aumento de la conectividad mundial que está facilitando su propagación a nuevas regiones.... Más
Instrumento de próxima generación detecta trastornos de la hemoglobina en recién nacidos
Las hemoglobinopatías, las enfermedades hereditarias más extendidas a nivel mundial, afectan a alrededor del 7 % de la población como portadores, y el 2,7 % de los recién nacidos nacen con estas enfermedades.... Más
Prueba de sangre POC por punción digital determina riesgo de sepsis neutropénica en pacientes sometidos a quimioterapia
La neutropenia, una disminución de los neutrófilos (un tipo de glóbulo blanco crucial para combatir las infecciones), es un efecto secundario frecuente de ciertos tratamientos contra... Más
Primera prueba rápida y asequible para beta talasemia demuestra precisión diagnóstica del 99 %
Los trastornos de la hemoglobina se encuentran entre las enfermedades monogénicas más prevalentes a nivel mundial. Entre los diversos trastornos de la hemoglobina, la beta talasemia, un trastorno sanguíneo... MásInmunología
ver canal
Análisis de sangre para diagnóstico de rechazo celular después de trasplante de órganos podría reemplazar las biopsias quirúrgicas
Los órganos trasplantados enfrentan constantemente el riesgo de ser rechazados por el sistema inmunológico del receptor, que los diferencia de los órganos no propios mediante... Más
Herramienta de IA ajusta con precisión los medicamentos contra el cáncer con los pacientes utilizando información de cada célula tumoral
Las estrategias actuales para emparejar a los pacientes con cáncer con tratamientos específicos a menudo dependen de la secuenciación masiva de ADN y ARN tumoral, que proporciona un perfil promedio de... Más
Pruebas genéticas combinadas con la detección de fármacos personalizadas en muestras de tumores podrían revolucionar el tratamiento del cáncer
El tratamiento del cáncer generalmente se adhiere a un estándar de atención: regímenes establecidos y validados estadísticamente que son efectivos para la mayoría de los pacientes. Sin embargo, la variabilidad... Más
Método de prueba podría ayudar a más pacientes recibir tratamiento adecuado contra el cáncer
El tratamiento del cáncer no siempre es una solución única, pero el campo de la investigación del cáncer está dando grandes pasos para encontrar a los pacientes los tratamientos más eficaces para sus afecciones... MásPatología
ver canalSistema de imágenes digitales impulsado por IA podría revolucionar el diagnóstico del cáncer
El proceso de biopsia es importante para confirmar la presencia de cáncer. En la técnica de histopatología convencional, el tejido se extirpa, se corta, se tiñe, se monta en... Más
Nuevo panel de mycobacterium tuberculosis respalda la vigilancia en tiempo real y combate la resistencia a los antimicrobianos
La tuberculosis (TB), la principal causa de muerte por enfermedad infecciosa a nivel mundial, es una infección bacteriana contagiosa que se propaga principalmente a través de la tos de pacientes con tuberculosis... Más
Análisis de tejido espacial identifica patrones asociados con la recaída del cáncer de ovario
El carcinoma de ovario seroso de alto grado es el tipo más letal de cáncer de ovario y plantea importantes desafíos de detección. Por lo general, los pacientes responden inicialmente... Más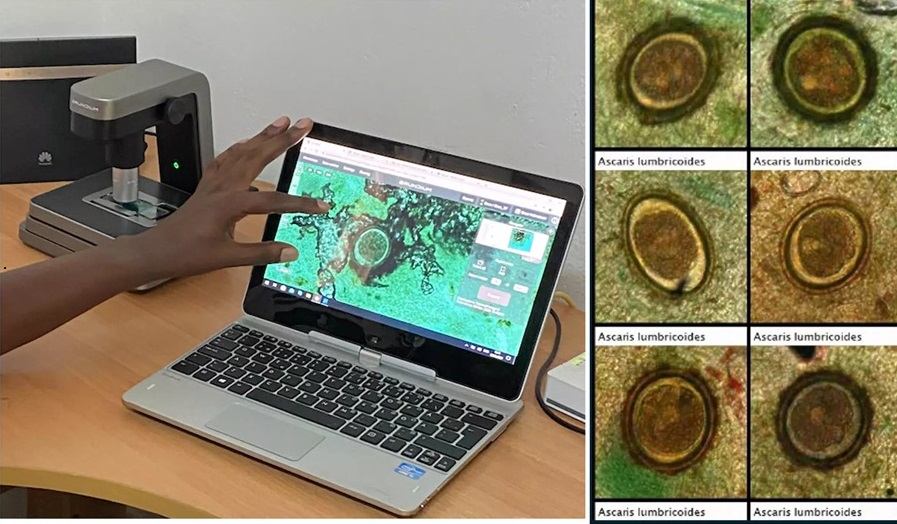
IA basada en imágenes se muestra prometedora para detección de parásitos en muestras de heces digitalizadas
Las infecciones por helmintos transmitidos por el suelo (STH), comúnmente conocidas como gusanos parásitos intestinales, se encuentran entre las enfermedades tropicales desatendidas más... MásTecnología
ver canal
Nuevo sistema de diagnóstico de laboratorio en un chip iguala la precisión de las pruebas de PCR
Si bien las pruebas de PCR son el estándar de oro en cuanto a precisión para las pruebas de virología, tienen limitaciones como la complejidad, la necesidad de operadores de laboratorio capacitados y tiempos... Más
Biosensor de ADN permite diagnóstico temprano del cáncer de cuello uterino
El disulfuro de molibdeno (MoS2), reconocido por su potencial para formar nanoláminas bidimensionales como el grafeno, es un material que llama cada vez más la atención de la comunidad... Más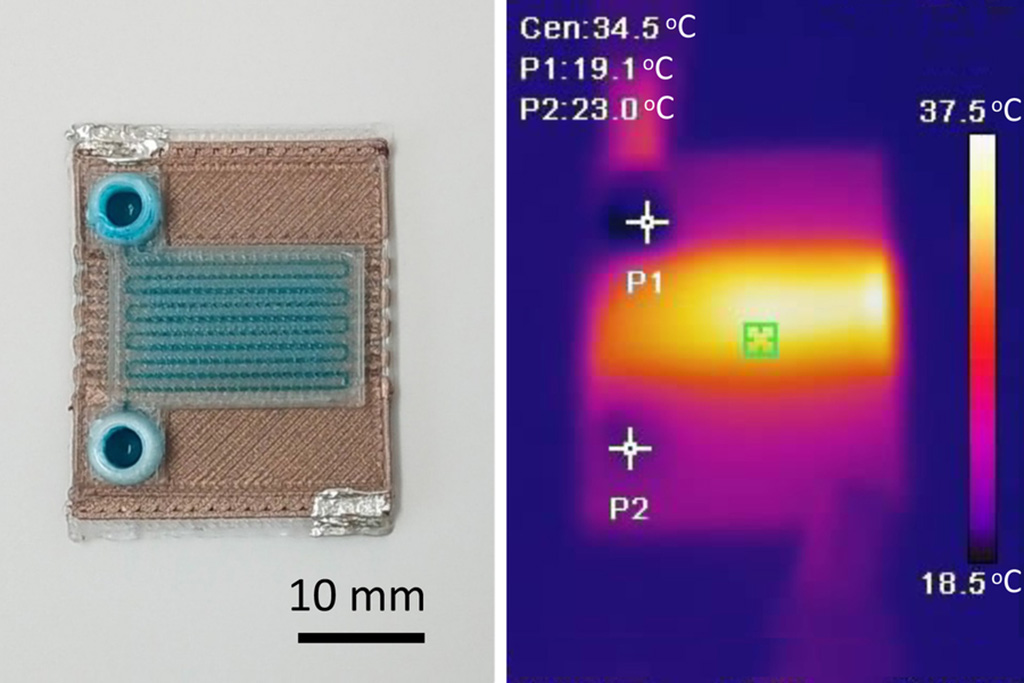
Dispositivos de microfluidos autocalentables pueden detectar enfermedades en pequeñas muestras de sangre o fluidos
Los microfluidos, que son dispositivos en miniatura que controlan el flujo de líquidos y facilitan reacciones químicas, desempeñan un papel clave en la detección de enfermedades... Más
Avance en tecnología de diagnóstico podría hacer que pruebas en el sitio sean ampliamente accesibles
Las pruebas caseras adquirieron una importancia significativa durante la pandemia de COVID-19, sin embargo, la disponibilidad de pruebas rápidas es limitada y la mayoría de ellas solo pueden conducir un... MásIndustria
ver canal
Congreso ECCMID cambia de nombre a ESCMID Global
En los últimos años, la Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas (ESCMID, Basilea, Suiza) ha evolucionado notablemente. La sociedad es ahora más... Más