Nuevo método de análisis detecta patógenos en sangre de forma más rápida y precisa al fundir ADN
Actualizado el 19 Mar 2024
A nivel mundial, una alarmante proporción de una de cada cinco muertes se atribuye a complicaciones relacionadas con la sepsis, y los niños representan el 41 % de estas muertes. La práctica común implica administrar antibióticos a pacientes con sepsis mientras se esperan los resultados de los hemocultivos, lo que puede contribuir a la resistencia a los antibióticos. El tratamiento ineficaz de la sepsis puede ser perjudicial, ya que hasta el 30 % de los pacientes reciben tratamientos incorrectos, lo que eleva aún más su riesgo de muerte. La naturaleza crítica del diagnóstico oportuno y preciso en los casos de sepsis se ve subrayada por el hecho de que el riesgo de mortalidad aumenta en un 4 % cada hora que la infección no se identifica o trata adecuadamente. Ahora, una nueva técnica de análisis ofrece una detección de patógenos más rápida y precisa en muestras de sangre en comparación con los hemocultivos tradicionales, que son el estándar en el diagnóstico de infecciones.
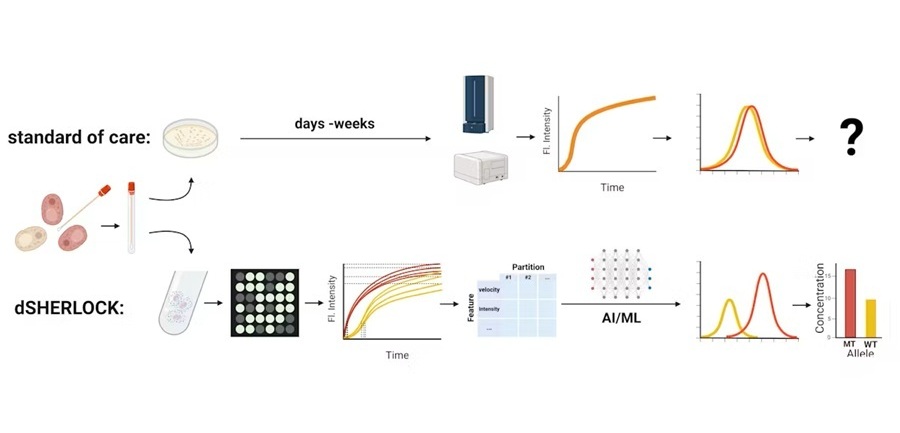
El nuevo método, llamado análisis digital de fusión de ADN, ha sido desarrollado por investigadores de la UC San Diego (La Jolla, CA, EUA) y es capaz de ofrecer resultados en menos de seis horas. Esto marca una mejora significativa con respecto a las típicas 15 horas a varios días que requieren los métodos de cultivo, dependiendo del patógeno involucrado. El proceso utiliza la fusión de ADN digital universal de alta resolución, lo que implica calentar el ADN hasta que se separa. Cada secuencia de ADN revela una firma única durante el proceso de fusión. Al obtener imágenes y analizar este proceso, los algoritmos de aprendizaje automático pueden discernir los tipos de ADN en las muestras e identificar patógenos. Este método no sólo supera a los hemocultivos en términos de velocidad, sino que también tiene un riesgo sustancialmente menor de generar falsos positivos en comparación con otras tecnologías emergentes de detección de ADN, como la secuenciación de próxima generación.

La investigación comenzó con un mililitro de sangre de cada uno de 17 pacientes en un estudio clínico preliminar. Estas muestras se recolectaron simultáneamente con las de hemocultivos de bebés y niños pequeños. Los investigadores perfeccionaron el proceso de aislamiento de ADN y las técnicas de aprendizaje automático para minimizar o eliminar la interferencia del ADN humano en contraste con el ADN patógeno en las muestras. Refinaron un algoritmo de aprendizaje automático para distinguir con precisión entre las curvas de fusión de los patógenos y el ruido de fondo. Este algoritmo correlaciona las curvas observadas con una base de datos de curvas de fusión de ADN conocidas. Además, puede identificar curvas producidas por organismos que no están en esta base de datos, lo que es particularmente útil para detectar patógenos raros o emergentes en una muestra.
Los resultados de este método no sólo fueron consistentes con los obtenidos de hemocultivos de las mismas muestras, sino que tampoco arrojaron ningún falso positivo. Esto contrasta con otras pruebas basadas en la amplificación de ácidos nucleicos y bases de datos de secuenciación de ADN de última generación, que tienden a amplificar todo el ADN presente, dando lugar a falsos positivos. La contaminación de diversas fuentes, como el medio ambiente, los tubos de ensayo, los reactivos y la piel, a menudo puede generar desafíos a la hora de interpretar los resultados de las pruebas. Este nuevo método detectó patógenos entre 7,5 horas y aproximadamente 3 días más rápido que los hemocultivos convencionales. Además, proporciona más que un simple resultado binario positivo o negativo; cuantifica la extensión de la presencia de patógenos en las muestras. Los planes futuros incluyen realizar un estudio clínico más amplio y extender la metodología a pacientes adultos.
“Esta es la primera vez que se prueba este método en sangre total de pacientes con sospecha de sepsis. Por lo tanto, este estudio es una vista previa más realista de cómo podría funcionar la tecnología en escenarios clínicos reales”, dijo Stephanie Fraley, profesora de la UC San Diego. "Queremos brindar a los médicos la capacidad de tratar a sus pacientes basándose no en datos agregados, sino con datos individuales precisos y exactos, permitiendo una medicina verdaderamente personalizada".
Enlaces relacionados:
UC San Diego