Una variante genética sin sentido se asocia con la enfermedad de Crohn
|
Por el equipo editorial de LabMedica en español Actualizado el 26 Feb 2019 |
Imagen: El kit de preparación de bibliotecas de ADN, Nextera (Fotografía cortesía de Illumina).
Las interacciones gen-microbioma son importantes en la etiología y patogénesis de la enfermedad inflamatoria intestinal, un trastorno inflamatorio crónico del tracto gastrointestinal que consiste en la enfermedad de Crohn y la colitis ulcerativa. Esta última condición causa inflamación de larga duración y llagas (úlceras) en el revestimiento interno del intestino grueso (colon) y el recto.
La enfermedad intestinal inflamatoria (EII) es un trastorno crónico frecuente del tracto gastrointestinal. Los pacientes con esta enfermedad experimentan períodos de inflamación alternados por períodos de remisión. Los subgrupos más comunes de EII son la enfermedad de Crohn (EC), la colitis ulcerosa (CU) y la enfermedad inflamatoria intestinal indeterminada (EIII). La inflamación causada por la enfermedad de Crohn a menudo se extiende profundamente en las capas del tejido intestinal afectado.
Científicos de la Universidad de Groningen (Groningen, Países Bajos) y sus colegas, recolectaron muestras de heces, sangre periférica y datos extensos de fenotipos de 291 pacientes con enfermedad inflamatoria intestinal que comprendían 171 pacientes con EC, 104 pacientes con CU y 16 Pacientes con EIII, además de 476 controles sanos. Para cada participante, hubo disponibilidad de muestras de sangre periférica almacenadas en tubos con EDTA.
La extracción de ADN se realizó utilizando el Autopure LS con química Puregene (Qiagen NV, Venlo, Países Bajos). La preparación de la muestra utilizó el kit de preparación Illumina Nextera (Illumina, San Diego, CA, EUA) y el enriquecimiento de las secuencias exónicas se realizó mediante captura híbrida con el Illumina rapid Capture Enrichment (objetivo de 37 mb). La composición microbiana intestinal de las muestras de heces se determinó mediante la secuenciación de etiquetas del gen 16S rARN. El ADN fecal se aisló haciendo alícuotas y para el aislamiento del ADN microbiano se utilizó el Mini Kit Qiagen AllPrep ADN/ARN. Se realizó una secuenciación Illumina MiSeq de extremo pareado de la región V4 del gen 16S rARN. Se infirió la asociación entre el alelo de riesgo SLC39A8 [Thr] 391 y la composición microbiana intestinal en pacientes con enfermedad inflamatoria intestinal y en los controles sanos.
El equipo informó que los pacientes con enfermedad de Crohn eran más a menudo portadores de la variante sin sentido (21/171, 12,3%) que los controles (30/476, 6,3%) (OR = 2,1). No pudieron identificar asociaciones entre el estado del portador y la composición general del microbioma intestinal y la riqueza microbiana en todos los grupos evaluados después de corregir para los posibles factores de confusión. Identificaron 37 unidades taxonómicas operativas diferentes que se asocian con el estado de portador entre los grupos evaluados. Dos de estos 37 fueron identificados antes en el estudio de descubrimiento.
Los autores concluyeron que en futuros estudios de genes y microbiomas se necesitan tamaños de muestras mucho más grandes, análisis estadísticos más estrictos, especialmente con respecto a los recuentos medios de unidades taxonómicas operativas (UTO) y la corrección de factores de confusión, la replicación en cohortes independientes y descripciones detalladas de los métodos con el fin de identificar asociaciones genoma-microbioma tanto en EII como en la EC. Pudieron identificar la variante genética que se asocia con la enfermedad de Crohn, aunque el impacto en la composición del microbioma se limitó a unas pocas UTO. El estudio fue publicado el 31 de enero de 2019 en la revista PLOS ONE.
Enlace relacionado:
Universidad de Groningen
Qiagen
Illumina
La enfermedad intestinal inflamatoria (EII) es un trastorno crónico frecuente del tracto gastrointestinal. Los pacientes con esta enfermedad experimentan períodos de inflamación alternados por períodos de remisión. Los subgrupos más comunes de EII son la enfermedad de Crohn (EC), la colitis ulcerosa (CU) y la enfermedad inflamatoria intestinal indeterminada (EIII). La inflamación causada por la enfermedad de Crohn a menudo se extiende profundamente en las capas del tejido intestinal afectado.
Científicos de la Universidad de Groningen (Groningen, Países Bajos) y sus colegas, recolectaron muestras de heces, sangre periférica y datos extensos de fenotipos de 291 pacientes con enfermedad inflamatoria intestinal que comprendían 171 pacientes con EC, 104 pacientes con CU y 16 Pacientes con EIII, además de 476 controles sanos. Para cada participante, hubo disponibilidad de muestras de sangre periférica almacenadas en tubos con EDTA.
La extracción de ADN se realizó utilizando el Autopure LS con química Puregene (Qiagen NV, Venlo, Países Bajos). La preparación de la muestra utilizó el kit de preparación Illumina Nextera (Illumina, San Diego, CA, EUA) y el enriquecimiento de las secuencias exónicas se realizó mediante captura híbrida con el Illumina rapid Capture Enrichment (objetivo de 37 mb). La composición microbiana intestinal de las muestras de heces se determinó mediante la secuenciación de etiquetas del gen 16S rARN. El ADN fecal se aisló haciendo alícuotas y para el aislamiento del ADN microbiano se utilizó el Mini Kit Qiagen AllPrep ADN/ARN. Se realizó una secuenciación Illumina MiSeq de extremo pareado de la región V4 del gen 16S rARN. Se infirió la asociación entre el alelo de riesgo SLC39A8 [Thr] 391 y la composición microbiana intestinal en pacientes con enfermedad inflamatoria intestinal y en los controles sanos.
El equipo informó que los pacientes con enfermedad de Crohn eran más a menudo portadores de la variante sin sentido (21/171, 12,3%) que los controles (30/476, 6,3%) (OR = 2,1). No pudieron identificar asociaciones entre el estado del portador y la composición general del microbioma intestinal y la riqueza microbiana en todos los grupos evaluados después de corregir para los posibles factores de confusión. Identificaron 37 unidades taxonómicas operativas diferentes que se asocian con el estado de portador entre los grupos evaluados. Dos de estos 37 fueron identificados antes en el estudio de descubrimiento.
Los autores concluyeron que en futuros estudios de genes y microbiomas se necesitan tamaños de muestras mucho más grandes, análisis estadísticos más estrictos, especialmente con respecto a los recuentos medios de unidades taxonómicas operativas (UTO) y la corrección de factores de confusión, la replicación en cohortes independientes y descripciones detalladas de los métodos con el fin de identificar asociaciones genoma-microbioma tanto en EII como en la EC. Pudieron identificar la variante genética que se asocia con la enfermedad de Crohn, aunque el impacto en la composición del microbioma se limitó a unas pocas UTO. El estudio fue publicado el 31 de enero de 2019 en la revista PLOS ONE.
Enlace relacionado:
Universidad de Groningen
Qiagen
Illumina
Últimas Diagnóstico Molecular noticias
- Primera prueba que utiliza microARN para predecir toxicidad de terapia contra el cáncer
- Ensayo basado en células proporciona detección sensible y específica de autoanticuerpos en desmielinización
- Novedosa tecnología en POC ofrece resultados precisos del VIH en minutos
- Análisis de sangre descarta riesgo futuro de demencia
- Prueba de dímero D puede identificar pacientes con mayor riesgo de embolia pulmonar
- Nuevos biomarcadores mejoran la detección temprana y seguimiento de la lesión renal
- Inmunoensayos de quimioluminiscencia respaldan diagnóstico de Alzheimer
- Análisis de sangre identifica múltiples biomarcadores para diagnóstico rápido de lesiones de médula espinal
- Análisis de sangre muy preciso diagnostica Alzheimer y mide progresión de demencia
- Prueba sencilla basada en PCR de ADN permite tratamiento personalizado de vaginosis bacteriana
- Prueba de diagnóstico detiene transmisión de hepatitis B de madre a hijo
- Simple prueba de orina podría ayudar a evitar exploraciones invasivas para cáncer de riñón
- Nueva prueba para cáncer de intestino mejorará detección temprana
- Prueba refinada mejora diagnóstico de enfermedad de Parkinson
- Nuevo método diagnostica rápidamente riesgo de ECV mediante análisis molecular de sangre
- Análisis de sangre muestra resultados prometedores para detección temprana de demencia
Canales
Química Clínica
ver canal
Nanotubos de carbono ayudan a construir sensores precisos para monitoreo continuo de la salud
Los sensores actuales pueden medir diversos indicadores de salud, como los niveles de glucosa en sangre. Sin embargo, es necesario desarrollar materiales para sensores más precisos y sensibles que... Más
Dispositivo basado en papel mejora la precisión de prueba del VIH
En las regiones donde el acceso a las clínicas para realizar análisis de sangre rutinarios presenta obstáculos financieros y logísticos, los pacientes con VIH pueden recolectar... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásMicrobiología
ver canal
Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
Las infecciones resistentes a los medicamentos, en particular las causadas por bacterias mortales como la tuberculosis y el estafilococo, se están convirtiendo rápidamente en una emergencia... Más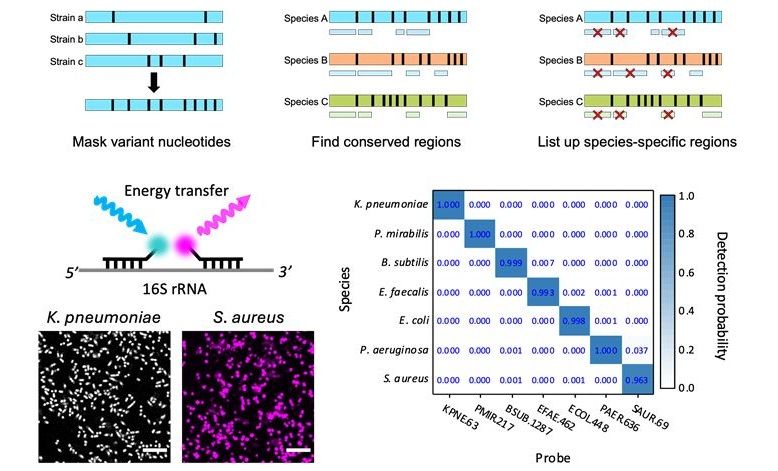
Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
La identificación rápida y precisa de microbios patógenos en muestras de pacientes es esencial para el tratamiento eficaz de enfermedades infecciosas agudas, como la sepsis.... MásPatología
ver canal
Modelo de IA predice eficazmente resultados de pacientes con cáncer de pulmón
El adenocarcinoma de pulmón, la forma más común de cáncer de pulmón de células no pequeñas (CPCNP), suele adoptar uno de seis patrones de crecimiento distintos,... Más
Modelo de IA predice respuesta al tratamiento del cáncer de vejiga
Cada año en Estados Unidos, se diagnostican alrededor de 81.000 nuevos casos de cáncer de vejiga, lo que provoca aproximadamente 17.000 muertes al año. El cáncer de vejiga ... MásTecnología
ver canal
Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
El dolor crónico es una afección generalizada que sigue siendo difícil de controlar, y los métodos clínicos existentes para su tratamiento se basan en gran medida en... Más
Innovador sensor fluorométrico sin etiquetas permite detección más sensible del ARN viral
Los virus representan un importante riesgo para la salud mundial, como lo demuestran las recientes pandemias, lo que hace que la detección e identificación tempranas sean esenciales para... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más