Secuenciación en células individuales revela diversidad clonal entre los pacientes con LMA
|
Por el equipo editorial de LabMedica en español Actualizado el 03 Nov 2020 |

Imagen: El sistema de secuenciación NovaSeq 6000 (Fotografía cortesía de Illumina).
Un cuerpo creciente de evidencia respalda el papel de la diversidad clonal en la resistencia terapéutica, la recurrencia y los malos resultados en el cáncer. La diversidad clonal también refleja la historia de la acumulación de mutaciones somáticas dentro de un tumor.
La capacidad de inferir heterogeneidad clonal y filogenia tumoral a partir de datos de secuenciación masiva es inherentemente limitada, porque las técnicas de secuenciación masiva no pueden inferir de manera confiable ocurrencias simultáneas de mutaciones y, por lo tanto, a menudo fallan en la reconstrucción exacta de la subestructura clonal. La secuenciación de ADN de una sola célula (scADN-seq) puede abordar algunos de estos desafíos.
Un gran equipo de científicos del Centro de Cáncer MD Anderson de la Universidad de Texas (Houston, TX, EUA), analizó 154 muestras (140 células mononucleares de médula ósea (BMMC) y 14 células mononucleares de sangre periférica) de 123 pacientes con leucemia mieloide aguda (LMA) que tenían al menos una mutación somática cubierta por el panel dirigido para scADN-seq. De los 123 pacientes, 108 pacientes fueron analizados antes del tratamiento (N = 98) o en el punto de tiempo de recaída/refractaria (N = 10). Entre 123 pacientes, 97 fueron analizados por scADN-seq, 23 fueron analizados por secuenciación simultánea de ADN de una sola célula y de las proteínas de superficie celular (scADN+protein-seq), y tres fueron analizados por scADN-seq y scADN+protein-seq.
La biblioteca agrupada se secuenció mediante una de las siguientes plataformas de secuenciación, MiSeq, HiSeq 4000 o NovaSeq 6000 (Illumina, San Diego, CA, EUA), con el procesamiento de pares de bases (pb) de 150 o 250 pares multiplexadas en el extremo. El equipo realizó una PCR digital por gotitas (ddPCR) utilizando el sistema digital QX200 Droplet (Bio-Rad Laboratories, Hércules, CA, EUA), para confirmar las variantes que fueron detectadas por scADN-seq, pero que no fueron detectadas por la secuenciación masiva. Se realizó un perfil simultáneo de la mutación del ADN y el inmunofenotipo de la superficie celular (scADN+protein-seq) utilizando el kit de panel de diseño personalizado y 10-15 anticuerpos oligoconjugados (Mission Bio, South San Francisco, CA, EUA). Los inmunofenotipos de las células de la médula ósea de pacientes con LMA se evaluaron mediante citometría de flujo de ocho colores en un instrumento FACSCanto II (BD Biosciences, San José, CA, EUA).
En total, los científicos secuenciaron más de 730.000 células para encontrar 543 mutaciones somáticas en 31 genes relacionados con el cáncer, el 98% de los cuales fueron validados ortogonalmente. Las mutaciones más comunes que detectaron fueron en NPM1, seguidas de las de DNMT3A y NRAS. Además, consiguieron que si bien se encontraron varias mutaciones que eran funcionalmente redundantes en los mismos pacientes, las alteraciones se encontraron a menudo en clones mutuamente excluyentes. Esto se extendió a las alteraciones que afectan a los genes de la vía de señalización del receptor tirosina quinasa (RTK)/Gas GTPasa (RAS)/MAP quinasa (MAPK), así como a las mutaciones IDH1 e IDH2 y a las mutaciones TET2 e IDH. Esto sugirió a los científicos que las células no necesitan dos mutaciones o que, cuando aparecen juntas, las mutaciones son tóxicas, lo que podría sugerir una posible vía de tratamiento para investigar.
Los investigadores también analizaron las correlaciones genotipo-fenotipo entre las células para encontrar, por ejemplo, que las células con mutaciones en NPM1 o IDH expresaban niveles más bajos de CD34 y HLA-DR, mientras que las células con una sola mutación en TP53 tenían un fenotipo CD34+ CD117+, pero que las mutaciones dobles en TP53 tenían un inmunofenotipo monocítico.
Koichi Takahashi, MD, PhD, autor principal del estudio, dijo: “Esta información también está disponible, de alguna manera, a partir de datos de secuenciación masiva longitudinal, pero creo que los datos de una sola célula proporcionan de manera única esta visión meticulosa de la dinámica clon por clon, lo que simplemente no es posible mediante la secuenciación masiva”. El estudio fue publicado el 21 de octubre de 2020 en la revista Nature Communications.
Enlace relacionado:
Centro de Cáncer MD Anderson de la Universidad de Texas
Bio-Rad Laboratories
Mission Bio
BD Biosciences
La capacidad de inferir heterogeneidad clonal y filogenia tumoral a partir de datos de secuenciación masiva es inherentemente limitada, porque las técnicas de secuenciación masiva no pueden inferir de manera confiable ocurrencias simultáneas de mutaciones y, por lo tanto, a menudo fallan en la reconstrucción exacta de la subestructura clonal. La secuenciación de ADN de una sola célula (scADN-seq) puede abordar algunos de estos desafíos.
Un gran equipo de científicos del Centro de Cáncer MD Anderson de la Universidad de Texas (Houston, TX, EUA), analizó 154 muestras (140 células mononucleares de médula ósea (BMMC) y 14 células mononucleares de sangre periférica) de 123 pacientes con leucemia mieloide aguda (LMA) que tenían al menos una mutación somática cubierta por el panel dirigido para scADN-seq. De los 123 pacientes, 108 pacientes fueron analizados antes del tratamiento (N = 98) o en el punto de tiempo de recaída/refractaria (N = 10). Entre 123 pacientes, 97 fueron analizados por scADN-seq, 23 fueron analizados por secuenciación simultánea de ADN de una sola célula y de las proteínas de superficie celular (scADN+protein-seq), y tres fueron analizados por scADN-seq y scADN+protein-seq.
La biblioteca agrupada se secuenció mediante una de las siguientes plataformas de secuenciación, MiSeq, HiSeq 4000 o NovaSeq 6000 (Illumina, San Diego, CA, EUA), con el procesamiento de pares de bases (pb) de 150 o 250 pares multiplexadas en el extremo. El equipo realizó una PCR digital por gotitas (ddPCR) utilizando el sistema digital QX200 Droplet (Bio-Rad Laboratories, Hércules, CA, EUA), para confirmar las variantes que fueron detectadas por scADN-seq, pero que no fueron detectadas por la secuenciación masiva. Se realizó un perfil simultáneo de la mutación del ADN y el inmunofenotipo de la superficie celular (scADN+protein-seq) utilizando el kit de panel de diseño personalizado y 10-15 anticuerpos oligoconjugados (Mission Bio, South San Francisco, CA, EUA). Los inmunofenotipos de las células de la médula ósea de pacientes con LMA se evaluaron mediante citometría de flujo de ocho colores en un instrumento FACSCanto II (BD Biosciences, San José, CA, EUA).
En total, los científicos secuenciaron más de 730.000 células para encontrar 543 mutaciones somáticas en 31 genes relacionados con el cáncer, el 98% de los cuales fueron validados ortogonalmente. Las mutaciones más comunes que detectaron fueron en NPM1, seguidas de las de DNMT3A y NRAS. Además, consiguieron que si bien se encontraron varias mutaciones que eran funcionalmente redundantes en los mismos pacientes, las alteraciones se encontraron a menudo en clones mutuamente excluyentes. Esto se extendió a las alteraciones que afectan a los genes de la vía de señalización del receptor tirosina quinasa (RTK)/Gas GTPasa (RAS)/MAP quinasa (MAPK), así como a las mutaciones IDH1 e IDH2 y a las mutaciones TET2 e IDH. Esto sugirió a los científicos que las células no necesitan dos mutaciones o que, cuando aparecen juntas, las mutaciones son tóxicas, lo que podría sugerir una posible vía de tratamiento para investigar.
Los investigadores también analizaron las correlaciones genotipo-fenotipo entre las células para encontrar, por ejemplo, que las células con mutaciones en NPM1 o IDH expresaban niveles más bajos de CD34 y HLA-DR, mientras que las células con una sola mutación en TP53 tenían un fenotipo CD34+ CD117+, pero que las mutaciones dobles en TP53 tenían un inmunofenotipo monocítico.
Koichi Takahashi, MD, PhD, autor principal del estudio, dijo: “Esta información también está disponible, de alguna manera, a partir de datos de secuenciación masiva longitudinal, pero creo que los datos de una sola célula proporcionan de manera única esta visión meticulosa de la dinámica clon por clon, lo que simplemente no es posible mediante la secuenciación masiva”. El estudio fue publicado el 21 de octubre de 2020 en la revista Nature Communications.
Enlace relacionado:
Centro de Cáncer MD Anderson de la Universidad de Texas
Bio-Rad Laboratories
Mission Bio
BD Biosciences
Últimas Diagnóstico Molecular noticias
- Prueba de sangre predice con precisión el riesgo de cáncer de pulmón y reduce la necesidad de escaneos de TC
- Firma única de autoanticuerpos ayuda a diagnosticar la esclerosis múltiple años antes de la aparición de síntomas
- Prueba de sangre podría detectar cánceres asociados al VPH 10 años antes del diagnóstico clínico
- Un diagnóstico de bajo costo en el punto de atención ampliará el acceso a pruebas de enfermedades de transmisión sexual
- Prueba de orina analiza 18 genes para identificar el cáncer de próstata de alto grado
- Prueba en orina detecta cáncer de cabeza y cuello
- Prueba de sangre detecta y monitorea cáncer pulmonar de células pequeñas agresivo
- Ensayo de aprendizaje automático basado en sangre detecta de forma no invasiva el cáncer de ovario
- Ensayo de PCR simple diferencia con precisión entre los subtipos de cáncer de pulmón de células pequeñas
- Enfoque revolucionario de análisis de células T permite detección temprana del cáncer
- Prueba genética única podría acelerar el diagnóstico de trastornos raros del desarrollo
- Analizador de pruebas sindrómicas actualizado permite acceso remoto a resultados de pruebas
- Prueba de PCR para infecciones respiratorias y de garganta detecta múltiples patógenos con síntomas coincidentes
- Técnica de enriquecimiento de ácido nucleico circulante en sangre permite diagnóstico no invasivo del cáncer de hígado
- Primera prueba molecular aprobada por la FDA para detectar malaria en donantes de sangre podría mejorar seguridad del paciente
- Prueba de biomarcadores líquidos detecta enfermedades neurodegenerativas antes de que aparezcan síntomas
Canales
Química Clínica
ver canal
Espectrómetro de masas impreso en 3D para el punto de atención supera a los modelos de última generación
La espectrometría de masas es una técnica precisa para identificar los componentes químicos de una muestra y tiene un potencial significativo para monitorear estados de salud de enfermedades... Más.jpg)
Prueba biomédica POC hace girar una gota de agua utilizando ondas sonoras para detección del cáncer
Los exosomas, pequeñas biopartículas celulares que transportan un conjunto específico de proteínas, lípidos y materiales genéticos, desempeñan un papel... Más
Prueba basada en células altamente confiable permite diagnóstico preciso de enfermedades endocrinas
Los métodos convencionales para medir el cortisol libre, la hormona del estrés del cuerpo, en la sangre o la saliva son bastante exigentes y requieren el procesamiento de muestras. Por lo tanto, el método... MásHematología
ver canal
Primera prueba NAT 4 en 1 para el cribado de arbovirus podría reducir el riesgo de infecciones transmitidas por transfusiones
Los arbovirus representan una amenaza emergente para la salud mundial, exacerbada por el cambio climático y el aumento de la conectividad mundial que está facilitando su propagación a nuevas regiones.... Más
Instrumento de próxima generación detecta trastornos de la hemoglobina en recién nacidos
Las hemoglobinopatías, las enfermedades hereditarias más extendidas a nivel mundial, afectan a alrededor del 7 % de la población como portadores, y el 2,7 % de los recién nacidos nacen con estas enfermedades.... Más
Prueba de sangre POC por punción digital determina riesgo de sepsis neutropénica en pacientes sometidos a quimioterapia
La neutropenia, una disminución de los neutrófilos (un tipo de glóbulo blanco crucial para combatir las infecciones), es un efecto secundario frecuente de ciertos tratamientos contra... Más
Primera prueba rápida y asequible para beta talasemia demuestra precisión diagnóstica del 99 %
Los trastornos de la hemoglobina se encuentran entre las enfermedades monogénicas más prevalentes a nivel mundial. Entre los diversos trastornos de la hemoglobina, la beta talasemia, un trastorno sanguíneo... MásInmunología
ver canal
Análisis de sangre para diagnóstico de rechazo celular después de trasplante de órganos podría reemplazar las biopsias quirúrgicas
Los órganos trasplantados enfrentan constantemente el riesgo de ser rechazados por el sistema inmunológico del receptor, que los diferencia de los órganos no propios mediante... Más
Herramienta de IA ajusta con precisión los medicamentos contra el cáncer con los pacientes utilizando información de cada célula tumoral
Las estrategias actuales para emparejar a los pacientes con cáncer con tratamientos específicos a menudo dependen de la secuenciación masiva de ADN y ARN tumoral, que proporciona un perfil promedio de... Más
Pruebas genéticas combinadas con la detección de fármacos personalizadas en muestras de tumores podrían revolucionar el tratamiento del cáncer
El tratamiento del cáncer generalmente se adhiere a un estándar de atención: regímenes establecidos y validados estadísticamente que son efectivos para la mayoría de los pacientes. Sin embargo, la variabilidad... Más
Método de prueba podría ayudar a más pacientes recibir tratamiento adecuado contra el cáncer
El tratamiento del cáncer no siempre es una solución única, pero el campo de la investigación del cáncer está dando grandes pasos para encontrar a los pacientes los tratamientos más eficaces para sus afecciones... MásMicrobiología
ver canal
Nuevos ensayos de hepatitis con marcado CE permite la detección temprana de infecciones
Según la Organización Mundial de la Salud (OMS), se estima que 354 millones de personas en todo el mundo padecen hepatitis B o C crónica. Estos virus son las principales causas de... Más
Prueba de PCR múltiplex identifica el 95 % de los patógenos que causan la sepsis en una hora
La sepsis contribuye a una de cada tres muertes hospitalarias en los Estados Unidos y, a nivel mundial, el shock séptico conlleva una tasa de mortalidad del 30 al 40 %. El diagnóstico temprano de la sepsis... Más
Prueba de bacterias bucales podría predecir la progresión del cáncer de colon
El cáncer de colon, una enfermedad relativamente común pero difícil de diagnosticar, requiere confirmación mediante una colonoscopia o cirugía. Recientemente, ha habido... Más.jpg)
Firma metabólica unica podría permitir el diagnóstico de sepsis dentro de una hora de la extracción de sangre
La sepsis es una afección potencialmente mortal provocada por una respuesta extrema del cuerpo a una infección. Requiere intervención médica inmediata para evitar una posible muerte o daños duraderos.... MásPatología
ver canalSistema de imágenes digitales impulsado por IA podría revolucionar el diagnóstico del cáncer
El proceso de biopsia es importante para confirmar la presencia de cáncer. En la técnica de histopatología convencional, el tejido se extirpa, se corta, se tiñe, se monta en... Más
Nuevo panel de mycobacterium tuberculosis respalda la vigilancia en tiempo real y combate la resistencia a los antimicrobianos
La tuberculosis (TB), la principal causa de muerte por enfermedad infecciosa a nivel mundial, es una infección bacteriana contagiosa que se propaga principalmente a través de la tos de pacientes con tuberculosis... Más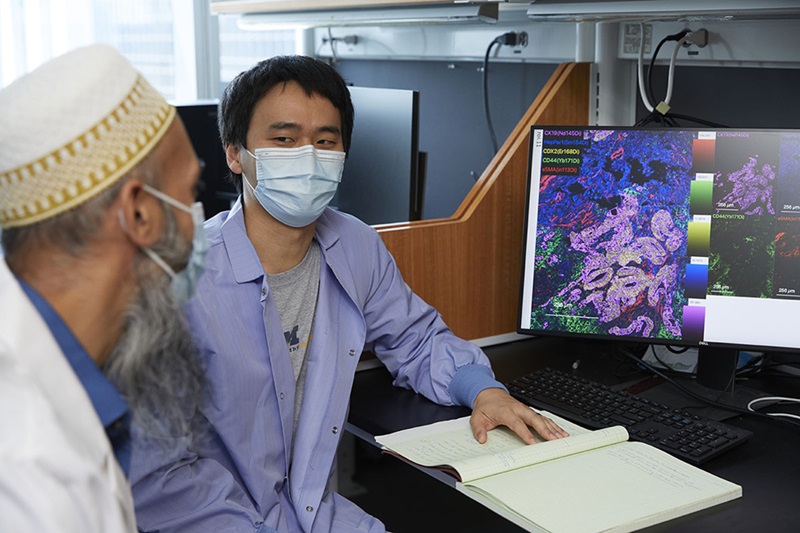
Análisis de tejido espacial identifica patrones asociados con la recaída del cáncer de ovario
El carcinoma de ovario seroso de alto grado es el tipo más letal de cáncer de ovario y plantea importantes desafíos de detección. Por lo general, los pacientes responden inicialmente... Más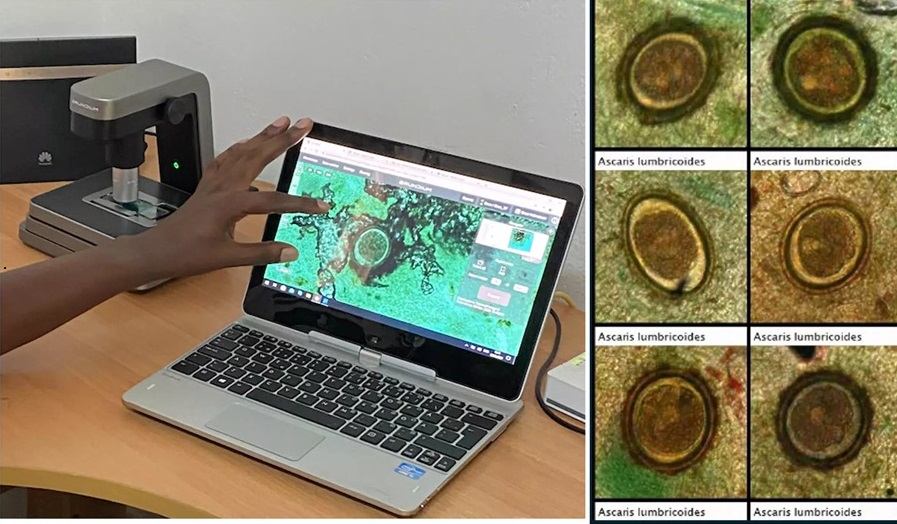
IA basada en imágenes se muestra prometedora para detección de parásitos en muestras de heces digitalizadas
Las infecciones por helmintos transmitidos por el suelo (STH), comúnmente conocidas como gusanos parásitos intestinales, se encuentran entre las enfermedades tropicales desatendidas más... MásTecnología
ver canal
Nuevo sistema de diagnóstico de laboratorio en un chip iguala la precisión de las pruebas de PCR
Si bien las pruebas de PCR son el estándar de oro en cuanto a precisión para las pruebas de virología, tienen limitaciones como la complejidad, la necesidad de operadores de laboratorio capacitados y tiempos... Más
Biosensor de ADN permite diagnóstico temprano del cáncer de cuello uterino
El disulfuro de molibdeno (MoS2), reconocido por su potencial para formar nanoláminas bidimensionales como el grafeno, es un material que llama cada vez más la atención de la comunidad... Más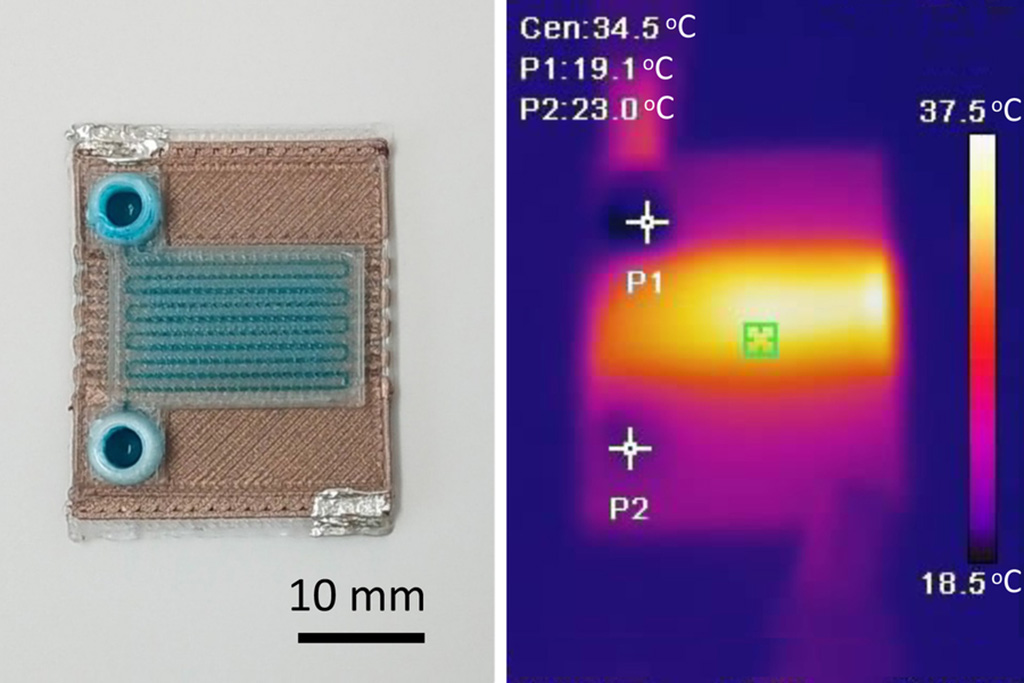
Dispositivos de microfluidos autocalentables pueden detectar enfermedades en pequeñas muestras de sangre o fluidos
Los microfluidos, que son dispositivos en miniatura que controlan el flujo de líquidos y facilitan reacciones químicas, desempeñan un papel clave en la detección de enfermedades... Más
Avance en tecnología de diagnóstico podría hacer que pruebas en el sitio sean ampliamente accesibles
Las pruebas caseras adquirieron una importancia significativa durante la pandemia de COVID-19, sin embargo, la disponibilidad de pruebas rápidas es limitada y la mayoría de ellas solo pueden conducir un... MásIndustria
ver canal
Congreso ECCMID cambia de nombre a ESCMID Global
En los últimos años, la Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas (ESCMID, Basilea, Suiza) ha evolucionado notablemente. La sociedad es ahora más... Más