Tecnología con ADN diagnostica tuberculosis rápidamente
|
Por el equipo editorial de LabMedica en español Actualizado el 06 Jan 2016 |

Imagen: El sistema de secuenciación de próxima generación, MiSeq (Fotografía cortesía de Illumina).
El diagnóstico prolongado y la obtención de resultados para las pruebas fenotípicas de susceptibilidad a los medicamentos (DST) del complejo Mycobacterium tuberculosis (MTBC), se deben al lento crecimiento del cultivo y contribuyen a retrasos reportados en la iniciación de tratamiento, entre 8 y 80 días.
Aunque los ensayos genotípicos pueden identificar, en menos de un día, las especies de micobacterias y las mutaciones que confieren resistencia a los medicamentos al MTBC, independientemente del cultivo, no detectan todas las mutaciones que confieren resistencia y se están usando, típicamente, después del cultivo microbiano.
Un equipo internacional de científicos dirigidos por los que están en el Hospital John Radcliffe (Universidad de Oxford; Oxford, Reino Unido), comparó la secuenciación de las micobacterias de todos los cultivos líquidos, recientemente positivos, con los flujos de trabajo diagnósticos de los laboratorio de rutina de ocho laboratorios en Europa y América del Norte, con respecto a la exactitud diagnóstica, los tiempos de procesamiento y el costo, entre el 6 de septiembre de 2013 y el 14 de abril de 2014. Los procedimientos de diagnóstico de rutina, en todos los centros, incluyeron la identificación de especie y el cultivo con isoniacida, rifampicina, etambutol y pirazinamida, con el fin de establecer la sensibilidad a los medicamentos.
Los equipos secuenciaron las muestras utilizando las plataformas locales MiSeq (Illumina, San Diego, CA, EUA) y procesaron los datos centralmente utilizando una tubería bioinformática semiautomática. Los equipos pudieron identificar especies o complejos utilizando la presencia o ausencia de genes, predijeron las susceptibilidades a los medicamentos, a partir de mutaciones que confieren resistencia, identificadas a partir de genomas MTBC de referencia asignados, y compararon la distancia genética con los aislados, previamente secuenciados, de MTBC en el Reino Unido, para detectar brotes.
Los científicos utilizaron la secuenciación de todo el genoma (WGS) y fueron capaces de detectar la presencia de la tuberculosis (TB) y si era resistente a los antibióticos de uso común, en un tiempo de una semana, que es hasta ocho veces más rápido que con la utilización de métodos de diagnóstico tradicionales. En comparación con los resultados de rutina, la WGS predijo especies con una exactitud del 93% en 322 de 345 muestras; en 356 muestras de micobacterias enviadas, y la sensibilidad a los medicamentos también mostró un 93% de exactitud en 628 de 672 muestras; 168 muestras de MTBC identificadas, con un intento de secuenciación. La tecnología novedosa también demostró ser más rentable, con un costo promedio de 481 libras esterlinas por cada caso positivo, en comparación con 517 libras estarlinas, por caso, utilizando las tecnologías actuales.
Louise J Pankhurst, PhD, la autora principal del estudio, dijo: “Este es un momento muy emocionante para estar trabajando en la investigación de las enfermedades infecciosas. El Reino Unido está a punto de convertirse en el primer país en el mundo en reemplazar el diagnóstico tradicional de la tuberculosis con la secuenciación todo el genoma. Nuestro estudio ha demostrado que esto acelerará dramáticamente el tiempo necesario para diagnosticar la TB, ayudando a los pacientes a recibir el tratamiento más efectivo tan pronto como sea posible y reducir el riesgo de transmisión de la enfermedad”. El estudio fue publicado el 4 de diciembre de 2015, en la revista Lancet Respiratory Medicine.
Enlaces relacionados:
The John Radcliffe Hospital
Illumina
Aunque los ensayos genotípicos pueden identificar, en menos de un día, las especies de micobacterias y las mutaciones que confieren resistencia a los medicamentos al MTBC, independientemente del cultivo, no detectan todas las mutaciones que confieren resistencia y se están usando, típicamente, después del cultivo microbiano.
Un equipo internacional de científicos dirigidos por los que están en el Hospital John Radcliffe (Universidad de Oxford; Oxford, Reino Unido), comparó la secuenciación de las micobacterias de todos los cultivos líquidos, recientemente positivos, con los flujos de trabajo diagnósticos de los laboratorio de rutina de ocho laboratorios en Europa y América del Norte, con respecto a la exactitud diagnóstica, los tiempos de procesamiento y el costo, entre el 6 de septiembre de 2013 y el 14 de abril de 2014. Los procedimientos de diagnóstico de rutina, en todos los centros, incluyeron la identificación de especie y el cultivo con isoniacida, rifampicina, etambutol y pirazinamida, con el fin de establecer la sensibilidad a los medicamentos.
Los equipos secuenciaron las muestras utilizando las plataformas locales MiSeq (Illumina, San Diego, CA, EUA) y procesaron los datos centralmente utilizando una tubería bioinformática semiautomática. Los equipos pudieron identificar especies o complejos utilizando la presencia o ausencia de genes, predijeron las susceptibilidades a los medicamentos, a partir de mutaciones que confieren resistencia, identificadas a partir de genomas MTBC de referencia asignados, y compararon la distancia genética con los aislados, previamente secuenciados, de MTBC en el Reino Unido, para detectar brotes.
Los científicos utilizaron la secuenciación de todo el genoma (WGS) y fueron capaces de detectar la presencia de la tuberculosis (TB) y si era resistente a los antibióticos de uso común, en un tiempo de una semana, que es hasta ocho veces más rápido que con la utilización de métodos de diagnóstico tradicionales. En comparación con los resultados de rutina, la WGS predijo especies con una exactitud del 93% en 322 de 345 muestras; en 356 muestras de micobacterias enviadas, y la sensibilidad a los medicamentos también mostró un 93% de exactitud en 628 de 672 muestras; 168 muestras de MTBC identificadas, con un intento de secuenciación. La tecnología novedosa también demostró ser más rentable, con un costo promedio de 481 libras esterlinas por cada caso positivo, en comparación con 517 libras estarlinas, por caso, utilizando las tecnologías actuales.
Louise J Pankhurst, PhD, la autora principal del estudio, dijo: “Este es un momento muy emocionante para estar trabajando en la investigación de las enfermedades infecciosas. El Reino Unido está a punto de convertirse en el primer país en el mundo en reemplazar el diagnóstico tradicional de la tuberculosis con la secuenciación todo el genoma. Nuestro estudio ha demostrado que esto acelerará dramáticamente el tiempo necesario para diagnosticar la TB, ayudando a los pacientes a recibir el tratamiento más efectivo tan pronto como sea posible y reducir el riesgo de transmisión de la enfermedad”. El estudio fue publicado el 4 de diciembre de 2015, en la revista Lancet Respiratory Medicine.
Enlaces relacionados:
The John Radcliffe Hospital
Illumina
Últimas Microbiología noticias
- Prueba de bacterias bucales podría predecir la progresión del cáncer de colon
- Firma metabólica unica podría permitir el diagnóstico de sepsis dentro de una hora de la extracción de sangre
- Innovadora plataforma de diagnóstico proporciona resultados de AST con velocidad sin precedentes
- Análisis de sangre predice sepsis e insuficiencia orgánica en niños
- Análisis de sangre para tuberculosis podría detectar millones de propagadores silenciosos
- Un análisis de sangre simple combinado con un modelo de riesgo personalizado mejora el diagnóstico de sepsis
- Nuevo análisis de sangre reduce tiempo de diagnóstico de infecciones por micobacterias no tuberculosas de meses a horas
- Nuevo análisis para tuberculosis podría ampliar acceso a pruebas en países de ingresos bajos y medios
- Prueba rápida diagnostica enfermedades tropicales en horas para tratamiento con antibióticos más rápido
- Pruebas moleculares rápidas permiten tratamiento antibiótico más rápido y específico para neumonía
- Plataforma rápida de PSA proporciona resultados terapéuticos específicos días antes que el estándar de atención actual
- Nuevo método de análisis detecta patógenos en sangre de forma más rápida y precisa al fundir ADN
- Prueba rápida de sepsis ofrece resultados dos días más rápidos
- Diagnóstico rápido portátil por PCR podría detectar gonorrea y susceptibilidad a antibióticos
- Prueba CRISPR diagnostica mpox más rápido que método de PCR de laboratorio
- Prueba de PCR multiplexada para detección de patógenos y resistencia a antibióticos ayuda a brindar un tratamiento rápido de ITU
Canales
Química Clínica
ver canal
Espectrómetro de masas impreso en 3D para el punto de atención supera a los modelos de última generación
La espectrometría de masas es una técnica precisa para identificar los componentes químicos de una muestra y tiene un potencial significativo para monitorear estados de salud de enfermedades... Más.jpg)
Prueba biomédica POC hace girar una gota de agua utilizando ondas sonoras para detección del cáncer
Los exosomas, pequeñas biopartículas celulares que transportan un conjunto específico de proteínas, lípidos y materiales genéticos, desempeñan un papel... Más
Prueba basada en células altamente confiable permite diagnóstico preciso de enfermedades endocrinas
Los métodos convencionales para medir el cortisol libre, la hormona del estrés del cuerpo, en la sangre o la saliva son bastante exigentes y requieren el procesamiento de muestras. Por lo tanto, el método... MásDiagnóstico Molecular
ver canal
Un diagnóstico de bajo costo en el punto de atención ampliará el acceso a pruebas de enfermedades de transmisión sexual
La gonorrea es la segunda infección bacteriana de transmisión sexual (ITS) más comúnmente reportada, con alrededor de 82 millones de casos en todo el mundo en 2020. La infección puede tener graves consecuencias... MásPrueba de orina de 18 genes para el cáncer de próstata ayuda a evitar biopsias innecesarias.
Uno de los principales desafíos en el tratamiento del cáncer de próstata es distinguir entre tumores de crecimiento lento, que tienen menos probabilidades de causar daño, y... MásHematología
ver canal
Primera prueba NAT 4 en 1 para el cribado de arbovirus podría reducir el riesgo de infecciones transmitidas por transfusiones
Los arbovirus representan una amenaza emergente para la salud mundial, exacerbada por el cambio climático y el aumento de la conectividad mundial que está facilitando su propagación a nuevas regiones.... Más
Instrumento de próxima generación detecta trastornos de la hemoglobina en recién nacidos
Las hemoglobinopatías, las enfermedades hereditarias más extendidas a nivel mundial, afectan a alrededor del 7 % de la población como portadores, y el 2,7 % de los recién nacidos nacen con estas enfermedades.... Más
Prueba de sangre POC por punción digital determina riesgo de sepsis neutropénica en pacientes sometidos a quimioterapia
La neutropenia, una disminución de los neutrófilos (un tipo de glóbulo blanco crucial para combatir las infecciones), es un efecto secundario frecuente de ciertos tratamientos contra... Más
Primera prueba rápida y asequible para beta talasemia demuestra precisión diagnóstica del 99 %
Los trastornos de la hemoglobina se encuentran entre las enfermedades monogénicas más prevalentes a nivel mundial. Entre los diversos trastornos de la hemoglobina, la beta talasemia, un trastorno sanguíneo... MásInmunología
ver canal
Análisis de sangre para diagnóstico de rechazo celular después de trasplante de órganos podría reemplazar las biopsias quirúrgicas
Los órganos trasplantados enfrentan constantemente el riesgo de ser rechazados por el sistema inmunológico del receptor, que los diferencia de los órganos no propios mediante... Más
Herramienta de IA ajusta con precisión los medicamentos contra el cáncer con los pacientes utilizando información de cada célula tumoral
Las estrategias actuales para emparejar a los pacientes con cáncer con tratamientos específicos a menudo dependen de la secuenciación masiva de ADN y ARN tumoral, que proporciona un perfil promedio de... Más
Pruebas genéticas combinadas con la detección de fármacos personalizadas en muestras de tumores podrían revolucionar el tratamiento del cáncer
El tratamiento del cáncer generalmente se adhiere a un estándar de atención: regímenes establecidos y validados estadísticamente que son efectivos para la mayoría de los pacientes. Sin embargo, la variabilidad... Más
Método de prueba podría ayudar a más pacientes recibir tratamiento adecuado contra el cáncer
El tratamiento del cáncer no siempre es una solución única, pero el campo de la investigación del cáncer está dando grandes pasos para encontrar a los pacientes los tratamientos más eficaces para sus afecciones... MásPatología
ver canal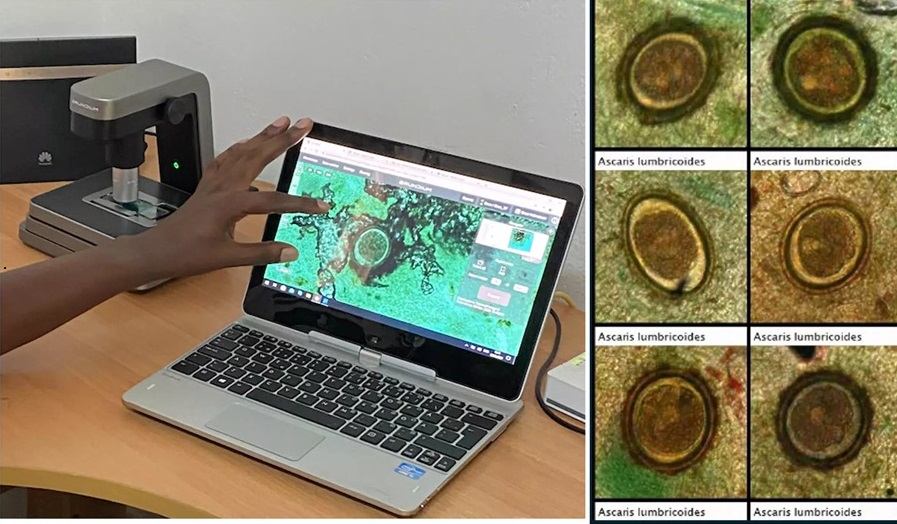
IA basada en imágenes se muestra prometedora para detección de parásitos en muestras de heces digitalizadas
Las infecciones por helmintos transmitidos por el suelo (STH), comúnmente conocidas como gusanos parásitos intestinales, se encuentran entre las enfermedades tropicales desatendidas más... Más
Algoritmos de inteligencia artificial potenciados por aprendizaje profundo mejoran la precisión en el diagnóstico de cáncer de piel.
Algoritmos de inteligencia artificial (IA) se utilizan cada vez más en diversos entornos clínicos, como la dermatología. Estos algoritmos se desarrollan entrenando una computadora... MásTecnología
ver canal
Nuevo sistema de diagnóstico de laboratorio en un chip iguala la precisión de las pruebas de PCR
Si bien las pruebas de PCR son el estándar de oro en cuanto a precisión para las pruebas de virología, tienen limitaciones como la complejidad, la necesidad de operadores de laboratorio capacitados y tiempos... Más
Biosensor de ADN permite diagnóstico temprano del cáncer de cuello uterino
El disulfuro de molibdeno (MoS2), reconocido por su potencial para formar nanoláminas bidimensionales como el grafeno, es un material que llama cada vez más la atención de la comunidad... Más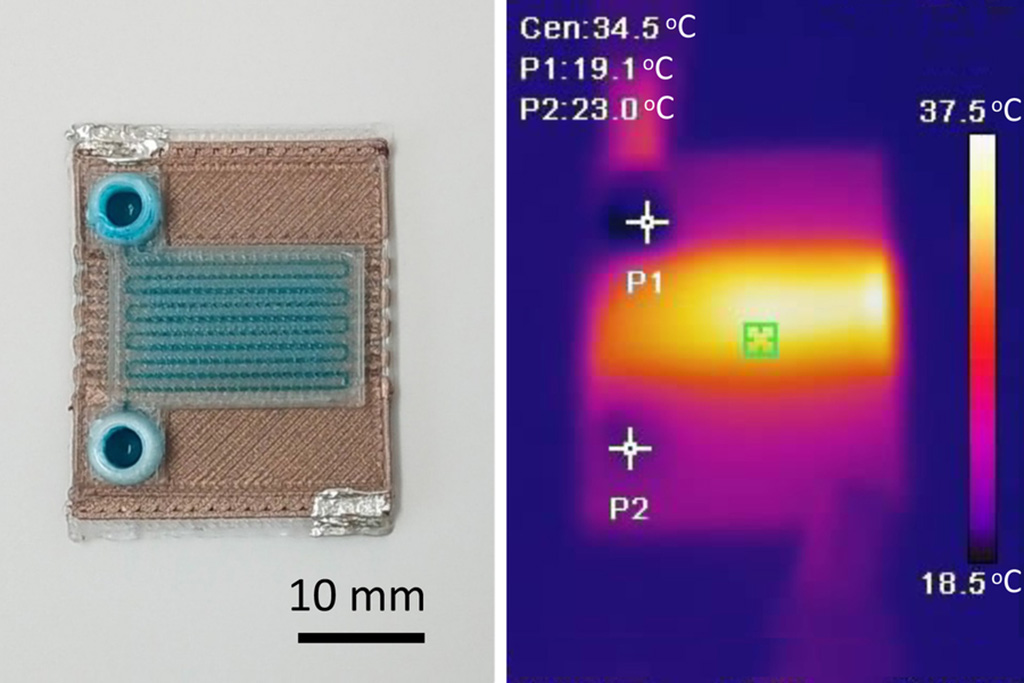
Dispositivos de microfluidos autocalentables pueden detectar enfermedades en pequeñas muestras de sangre o fluidos
Los microfluidos, que son dispositivos en miniatura que controlan el flujo de líquidos y facilitan reacciones químicas, desempeñan un papel clave en la detección de enfermedades... Más
Avance en tecnología de diagnóstico podría hacer que pruebas en el sitio sean ampliamente accesibles
Las pruebas caseras adquirieron una importancia significativa durante la pandemia de COVID-19, sin embargo, la disponibilidad de pruebas rápidas es limitada y la mayoría de ellas solo pueden conducir un... MásIndustria
ver canal
Congreso ECCMID cambia de nombre a ESCMID Global
En los últimos años, la Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas (ESCMID, Basilea, Suiza) ha evolucionado notablemente. La sociedad es ahora más... Más












.jpg)