Cambios en la carga bacteriana y los niveles de citoquinas en suero predicen la probabilidad de morir por sepsis
|
Por el equipo editorial de LabMedica en español Actualizado el 01 Jun 2020 |
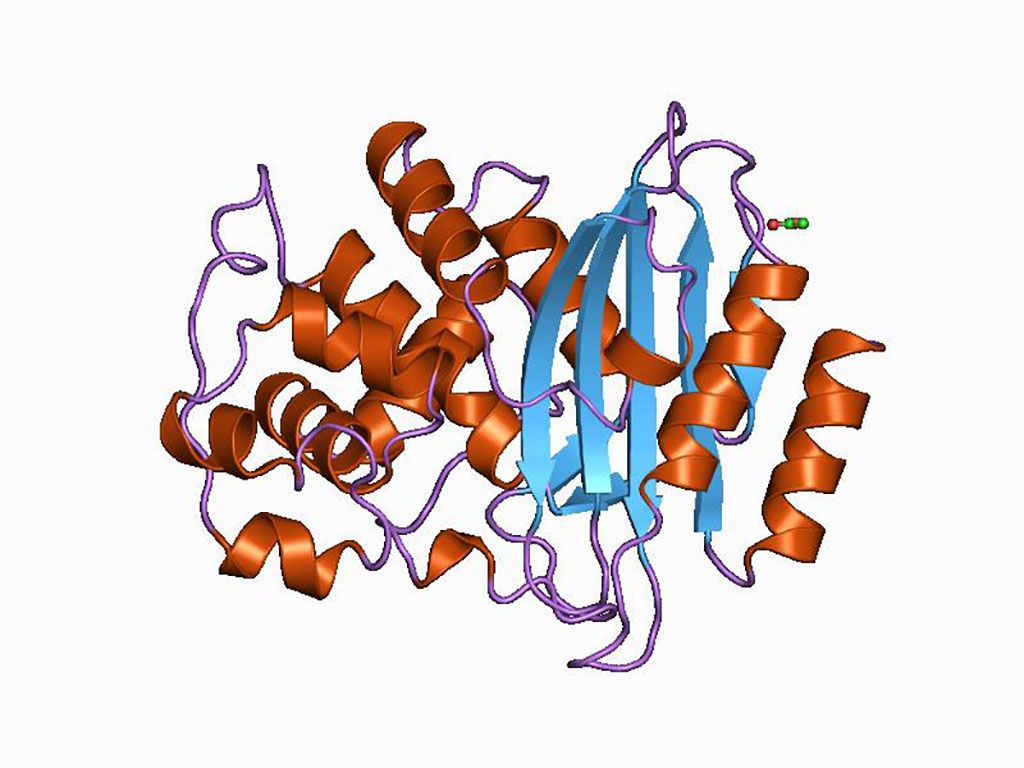
Imagen: Representación en dibujos animados de la estructura molecular de la proteína blaTEM (beta-lactamasa) (Fotografía cortesía de Wikimedia Commons)
Se puede utilizar un método de análisis para la cuantificación sensible y rápida de las citoquinas séricas y la carga bacteriana para predecir la probabilidad de que un paciente muera por sepsis o shock séptico.
La sepsis es causada por una respuesta inmune inflamatoria desencadenada por una infección. Es una afección potencialmente mortal que surge cuando la respuesta del cuerpo a la infección causa daño a sus propios tejidos y órganos. Los signos y síntomas comunes incluyen fiebre, aumento de la frecuencia cardíaca, aumento de la frecuencia respiratoria y confusión. También puede haber síntomas relacionados con una infección específica, como tos en caso de neumonía o dolor al orinar cuando hay una infección renal. En los muy jóvenes, ancianos y aquellos con un sistema inmune debilitado, puede no haber síntomas de una infección específica y la temperatura corporal puede ser baja o normal, en lugar de alta.
Para facilitar el diagnóstico, el seguimiento y el tratamiento de enfermedades infecciosas como las responsables de la sepsis, los investigadores de la Universidad de Chicago (IL, EUA) desarrollaron un método de cuantificación sensible y rápido para la carga bacteriana y las citoquinas séricas a partir de muestras biológicas humanas.
El método utiliza ensayos digitales de ligadura de proximidad (dPLA) para cuantificar marcadores de ácidos nucleicos y proteínas. La lectura de la PCR digital por gotitas (ddPCR), en el protocolo PLA, permitió la medición simultánea de genes 16S rARN específicos de Gram negativos (GN) y Gram positivos (GP) (que reflejan la abundancia de todas las bacterias GN y GP en las muestras de los pacientes) y del gen blaTEM (Beta-lactamasa) (que induce resistencia a los antibióticos Beta-lactámicos) junto con los niveles de proteína IL-6 y TNF-alfa en la misma muestra de los pacientes.
Una ventaja importante de este método de amplificación digital es su capacidad para cuantificar cambios muy pequeños en la concentración de estas moléculas. La ddPCR tiene una resolución de una molécula de ADN único en muestras, y los investigadores pudieron lograr una resolución subfemtomolar para los objetivos de proteínas.
Para demostrar el potencial de este enfoque, los investigadores lo utilizaron primero para analizar muestras de líquido de lavado broncoalveolar (BALF) de pacientes con asma leve a grave, y encontraron que los pacientes con asma tenían niveles más altos de bacterias GN e IL-6 que los individuos de control sanos. Luego utilizaron los ensayos para caracterizar longitudinalmente muestras de plasma de pacientes con shock séptico, revelando varias características moleculares asociadas con la recuperación o la muerte. Los análisis mostraron que los cambios en el tiempo de varios biomarcadores, y no sus concentraciones absolutas, fueron predictores confiables de los resultados de los pacientes. La aplicación del análisis del árbol de decisión a los resultados obtenidos por este método permitió la predicción de la mortalidad del paciente/tasa de recuperación del shock séptico con más del 90% de exactitud.
“Nuestros hallazgos brindan un método nuevo para el diagnóstico de sepsis con el potencial de identificar temprano el patógeno causal”, dijo el autor colaborador, Dr. Gokhan Mutlu, profesor de medicina de la Universidad de Chicago. “Esto nos permitirá usar los antibióticos apropiados antes de que los resultados del cultivo estén disponibles y minimizar el uso de antibióticos necesarios para tratar la infección. Al combinar los datos relacionados con el patógeno y la respuesta del huésped, podemos predecir los resultados en los pacientes con sepsis”.
“Se necesita una prueba rápida como esta en muchas situaciones y realmente podría cambiar el juego para el tratamiento de la sepsis”, dijo el autor principal, el Dr. Savas Tay, profesor asociado de ingeniería molecular en la Universidad de Chicago. “Esta es una enfermedad que puede matar a todos, independientemente de su situación”.
La prueba rápida para la cuantificación digital de citoquinas y bacterias se describió en la edición en línea del 25 de mayo de 2020 de la revista Nature Communications.
Enlace relacionado:
Universidad de Chicago
La sepsis es causada por una respuesta inmune inflamatoria desencadenada por una infección. Es una afección potencialmente mortal que surge cuando la respuesta del cuerpo a la infección causa daño a sus propios tejidos y órganos. Los signos y síntomas comunes incluyen fiebre, aumento de la frecuencia cardíaca, aumento de la frecuencia respiratoria y confusión. También puede haber síntomas relacionados con una infección específica, como tos en caso de neumonía o dolor al orinar cuando hay una infección renal. En los muy jóvenes, ancianos y aquellos con un sistema inmune debilitado, puede no haber síntomas de una infección específica y la temperatura corporal puede ser baja o normal, en lugar de alta.
Para facilitar el diagnóstico, el seguimiento y el tratamiento de enfermedades infecciosas como las responsables de la sepsis, los investigadores de la Universidad de Chicago (IL, EUA) desarrollaron un método de cuantificación sensible y rápido para la carga bacteriana y las citoquinas séricas a partir de muestras biológicas humanas.
El método utiliza ensayos digitales de ligadura de proximidad (dPLA) para cuantificar marcadores de ácidos nucleicos y proteínas. La lectura de la PCR digital por gotitas (ddPCR), en el protocolo PLA, permitió la medición simultánea de genes 16S rARN específicos de Gram negativos (GN) y Gram positivos (GP) (que reflejan la abundancia de todas las bacterias GN y GP en las muestras de los pacientes) y del gen blaTEM (Beta-lactamasa) (que induce resistencia a los antibióticos Beta-lactámicos) junto con los niveles de proteína IL-6 y TNF-alfa en la misma muestra de los pacientes.
Una ventaja importante de este método de amplificación digital es su capacidad para cuantificar cambios muy pequeños en la concentración de estas moléculas. La ddPCR tiene una resolución de una molécula de ADN único en muestras, y los investigadores pudieron lograr una resolución subfemtomolar para los objetivos de proteínas.
Para demostrar el potencial de este enfoque, los investigadores lo utilizaron primero para analizar muestras de líquido de lavado broncoalveolar (BALF) de pacientes con asma leve a grave, y encontraron que los pacientes con asma tenían niveles más altos de bacterias GN e IL-6 que los individuos de control sanos. Luego utilizaron los ensayos para caracterizar longitudinalmente muestras de plasma de pacientes con shock séptico, revelando varias características moleculares asociadas con la recuperación o la muerte. Los análisis mostraron que los cambios en el tiempo de varios biomarcadores, y no sus concentraciones absolutas, fueron predictores confiables de los resultados de los pacientes. La aplicación del análisis del árbol de decisión a los resultados obtenidos por este método permitió la predicción de la mortalidad del paciente/tasa de recuperación del shock séptico con más del 90% de exactitud.
“Nuestros hallazgos brindan un método nuevo para el diagnóstico de sepsis con el potencial de identificar temprano el patógeno causal”, dijo el autor colaborador, Dr. Gokhan Mutlu, profesor de medicina de la Universidad de Chicago. “Esto nos permitirá usar los antibióticos apropiados antes de que los resultados del cultivo estén disponibles y minimizar el uso de antibióticos necesarios para tratar la infección. Al combinar los datos relacionados con el patógeno y la respuesta del huésped, podemos predecir los resultados en los pacientes con sepsis”.
“Se necesita una prueba rápida como esta en muchas situaciones y realmente podría cambiar el juego para el tratamiento de la sepsis”, dijo el autor principal, el Dr. Savas Tay, profesor asociado de ingeniería molecular en la Universidad de Chicago. “Esta es una enfermedad que puede matar a todos, independientemente de su situación”.
La prueba rápida para la cuantificación digital de citoquinas y bacterias se describió en la edición en línea del 25 de mayo de 2020 de la revista Nature Communications.
Enlace relacionado:
Universidad de Chicago
Últimas Microbiología noticias
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
- Prueba CRISPR-TB permite diagnóstico temprano de enfermedad y cribado de la población
Canales
Química Clínica
ver canal
Monitorización con espectrometría de masas predice e identifica recaída temprana del mieloma
El mieloma, un tipo de cáncer que afecta la médula ósea, es actualmente incurable, aunque muchos pacientes pueden vivir más de 10 años tras el diagnóstico.... Más
Herramienta química a nanoescala 'brillantemente luminosa' mejora detección de enfermedades
Miles de moléculas brillantes disponibles comercialmente, conocidas como fluoróforos, se utilizan comúnmente en imágenes médicas, detección de enfermedades, marcado... MásDiagnóstico Molecular
ver canal
Herramienta genética predice supervivencia de pacientes con cáncer de páncreas
Un marcador tumoral es una sustancia presente en el organismo que puede indicar la presencia de cáncer. Estas sustancias, que pueden incluir proteínas, genes, moléculas u otros compuestos... Más
Prueba de orina diagnostica cáncer de próstata inicial
El cáncer de próstata es una de las principales causas de muerte en hombres a nivel mundial. Un desafío importante para diagnosticar la enfermedad es la ausencia de biomarcadores confiables... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Modelo de IA predice respuesta a terapia contra cáncer de riñón
Cada año, cerca de 435.000 personas son diagnosticadas con carcinoma renal de células claras (CRcc), lo que lo convierte en el subtipo más prevalente de cáncer de riñón.... Más
Kits de ensayo de enzima DUB sensibles y específicos requieren configuración mínima sin preparación del sustrato
La ubiquitinación y la desubiquitinación son dos procesos fisiológicos importantes en el sistema ubiquitina-proteasoma, responsable de la degradación de proteínas en... MásTecnología
ver canal
Algoritmo de firma ligera permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... Más
Tecnología de microchip desechable podría detectar selectivamente VIH en muestras de sangre completa
A finales de 2023, aproximadamente 40 millones de personas en todo el mundo vivían con VIH, y alrededor de 630.000 personas murieron por enfermedades relacionadas con el sida ese mismo año.... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más