Se utiliza la espectrometría de masas MALDI-TOF para identificar la pitiosis
|
Por el equipo editorial de LabMedica en español Actualizado el 01 Oct 2018 |

Imagen: El espectrómetro de masas de ionización-tiempo de vuelo de desorción láser asistida por matriz (Fotografía cortesía de Bruker Daltonics).
La pitiosis es una enfermedad infecciosa, potencialmente mortal, causada por el oomiceto Pythium insidiosum. La enfermedad se ha informado cada vez más en todo el mundo y a la mayoría de los pacientes con pitiosis es necesario realizar la extirpación quirúrgica de un órgano infectado.
En la última década, emergió la espectrometría de masas ionización-tiempo de vuelo de desorción láser asistida por matriz (MALDI-TOF MS), como una herramienta de diagnóstico, novedosa y poderosa, para facilitar la identificación clínica de muchos microorganismos patógenos, incluyendo las bacterias y los hongos.
Científicos de la Universidad Mahidol (Bangkok, Tailandia) aislaron un total de 13 cepas de P. insidiosum, de ocho humanos y de cinco animales con pitiosis, de diferentes ubicaciones geográficas y estas cepas se dividieron en dos grupos. Todos los organismos se mantuvieron en medios de agar dextrosa Sabouraud a 25°C. Varias porciones pequeñas de una colonia de cada organismo fueron transferidas a un matraz de 50 ml que contenía 10 mL de caldo dextrosa Sabouraud, y se incubaron a 37°C durante una semana, antes de recoger el material fúngico para realizar la extracción de proteínas.
La proteína se extrajo de los organismos y el sobrenadante que contenía la proteína extraída se colocó sobre una placa clara de acero pulido (Bruker Daltonics, Leipzig, Alemania) en 40 duplicados (con el fin de generar una base de datos MALDI-TOF MS para el P. insidiosum) o cinco repeticiones (para evaluar el desempeño de la MS MALDI-TOF para la identificación de P. insidiosum), secada al aire a temperatura ambiente, y luego superpuesta con 0,5 μL de la solución matriz. Se extrajeron moldes de ADN genómico (ADNg) de los organismos y se sometieron a una reacción en cadena de la polimerasa múltiple (PCR) basada en el polimorfismo de nucleótido único.
Los científicos informaron que la MALDI-TOF MS identificó con exactitud las 13 cepas de P. insidiosum analizadas, hasta el nivel de especie. Los espectros de masas de P. insidiosum no coincidieron con otros microorganismos, incluidos los hongos (es decir, especies de Aspergillus, especies de Fusarium y especies fúngicas de la clase Zigomicetos), que tienen morfologías microscópicas similares con este oomiceto. Los métodos de biotipificación basados en la secuencia MALDI-TOF MS y rADN clasificaron consistentemente a P. insidiosum en tres grupos: Clado-I (cepas americanas), II (cepas asiáticas y australianas) y III (principalmente cepas tailandesas).
Los autores concluyeron que la técnica de MALDI-TOF MS fue utilizada con éxito para la identificación y biotipificación de P. insidiosum. La base de datos de los espectros de masas obtenidos permite a los laboratorios de microbiología clínica, bien equipados con un espectrómetro de masas MALDI-TOF, identificar convenientemente a P. insidiosum, sin tener que requerir ningún reactivo específico de patógeno (es decir, antígeno, anticuerpo o cebadores). El estudio fue publicado el 6 de septiembre de 2018 en la revista International Journal of Infectious Diseases.
Enlace relacionado:
Universidad Mahidol
Bruker Daltonics
En la última década, emergió la espectrometría de masas ionización-tiempo de vuelo de desorción láser asistida por matriz (MALDI-TOF MS), como una herramienta de diagnóstico, novedosa y poderosa, para facilitar la identificación clínica de muchos microorganismos patógenos, incluyendo las bacterias y los hongos.
Científicos de la Universidad Mahidol (Bangkok, Tailandia) aislaron un total de 13 cepas de P. insidiosum, de ocho humanos y de cinco animales con pitiosis, de diferentes ubicaciones geográficas y estas cepas se dividieron en dos grupos. Todos los organismos se mantuvieron en medios de agar dextrosa Sabouraud a 25°C. Varias porciones pequeñas de una colonia de cada organismo fueron transferidas a un matraz de 50 ml que contenía 10 mL de caldo dextrosa Sabouraud, y se incubaron a 37°C durante una semana, antes de recoger el material fúngico para realizar la extracción de proteínas.
La proteína se extrajo de los organismos y el sobrenadante que contenía la proteína extraída se colocó sobre una placa clara de acero pulido (Bruker Daltonics, Leipzig, Alemania) en 40 duplicados (con el fin de generar una base de datos MALDI-TOF MS para el P. insidiosum) o cinco repeticiones (para evaluar el desempeño de la MS MALDI-TOF para la identificación de P. insidiosum), secada al aire a temperatura ambiente, y luego superpuesta con 0,5 μL de la solución matriz. Se extrajeron moldes de ADN genómico (ADNg) de los organismos y se sometieron a una reacción en cadena de la polimerasa múltiple (PCR) basada en el polimorfismo de nucleótido único.
Los científicos informaron que la MALDI-TOF MS identificó con exactitud las 13 cepas de P. insidiosum analizadas, hasta el nivel de especie. Los espectros de masas de P. insidiosum no coincidieron con otros microorganismos, incluidos los hongos (es decir, especies de Aspergillus, especies de Fusarium y especies fúngicas de la clase Zigomicetos), que tienen morfologías microscópicas similares con este oomiceto. Los métodos de biotipificación basados en la secuencia MALDI-TOF MS y rADN clasificaron consistentemente a P. insidiosum en tres grupos: Clado-I (cepas americanas), II (cepas asiáticas y australianas) y III (principalmente cepas tailandesas).
Los autores concluyeron que la técnica de MALDI-TOF MS fue utilizada con éxito para la identificación y biotipificación de P. insidiosum. La base de datos de los espectros de masas obtenidos permite a los laboratorios de microbiología clínica, bien equipados con un espectrómetro de masas MALDI-TOF, identificar convenientemente a P. insidiosum, sin tener que requerir ningún reactivo específico de patógeno (es decir, antígeno, anticuerpo o cebadores). El estudio fue publicado el 6 de septiembre de 2018 en la revista International Journal of Infectious Diseases.
Enlace relacionado:
Universidad Mahidol
Bruker Daltonics
Últimas Microbiología noticias
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
- Prueba CRISPR-TB permite diagnóstico temprano de enfermedad y cribado de la población
- Panel sindrómico ofrece respuestas rápidas para diagnóstico ambulatorio de enfermedades gastrointestinales
Canales
Química Clínica
ver canal
Nuevo método utiliza luz infrarroja pulsada para encontrar huellas del cáncer en plasma sanguíneo
Tradicionalmente, el diagnóstico de cáncer se ha basado en procedimientos invasivos o laboriosos, como las biopsias de tejido. Ahora, una nueva investigación publicada en ACS Central... Más
Nanotubos de carbono ayudan a construir sensores precisos para monitoreo continuo de la salud
Los sensores actuales pueden medir diversos indicadores de salud, como los niveles de glucosa en sangre. Sin embargo, es necesario desarrollar materiales para sensores más precisos y sensibles que... MásDiagnóstico Molecular
ver canal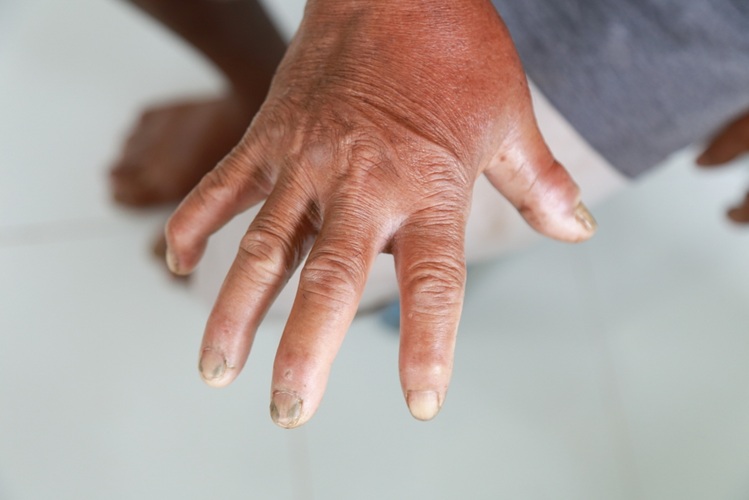
Análisis de sangre podría identificar a pacientes con riesgo de esclerodermia grave
La esclerosis sistémica, también conocida como esclerodermia, causa el endurecimiento de la piel y el tejido conectivo. En muchos casos, la enfermedad también puede dañar órganos... Más
Prueba de sangre basada en genes predice recurrencia del cáncer de piel avanzado
El melanoma, una forma agresiva de cáncer de piel, se vuelve extremadamente difícil de tratar una vez que se propaga a otras partes del cuerpo. En pacientes con tumores de melanoma metastásicos... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Nuevo método con corrección de errores detecta cáncer únicamente en muestras de sangre
La tecnología de biopsia líquida, que se basa en análisis de sangre para la detección temprana del cáncer y el seguimiento de la carga oncológica en los pacientes,... Más
Algoritmo "detector de metales" consigue tumores vulnerables
Científicos han desarrollado un algoritmo capaz de funcionar como un "detector de metales" para identificar tumores vulnerables, lo que supone un avance significativo en el tratamiento... MásTecnología
ver canal
Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
El dolor crónico es una afección generalizada que sigue siendo difícil de controlar, y los métodos clínicos existentes para su tratamiento se basan en gran medida en... Más
Innovador sensor fluorométrico sin etiquetas permite detección más sensible del ARN viral
Los virus representan un importante riesgo para la salud mundial, como lo demuestran las recientes pandemias, lo que hace que la detección e identificación tempranas sean esenciales para... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más