Técnica de secuenciación de ADN descubre la diversidad genética de los tumores cancerosos
|
Por el equipo editorial de LabMedica en español Actualizado el 08 Jul 2020 |

Imagen: Análisis del número de copias de una sola célula: la solución de cromo de una sola célula CNV, proporciona una solución integral y escalable para revelar la heterogeneidad del genoma y comprender la evolución clonal (Fotografía cortesía de 10X Genomics).
La capacidad de secuenciar el genoma de un tumor ha revolucionado el tratamiento del cáncer al identificar los promotores del cáncer a nivel molecular. Sin embargo, comprender la diversidad genética de las células individuales dentro de un tumor y cómo eso podría afectar la progresión de la enfermedad se mantiene como un desafío, debido a las limitaciones actuales de la secuenciación genómica.
El paradigma estándar es la secuenciación masiva de ADN genómico derivado de millones de células heterogéneas. En la secuenciación masiva, la capacidad de resolver la subclonalidad se confunde al depender de la inferencia indirecta, lo que, con frecuencia, resulta en una vista de conjunto dominada por el clon mayoritario. Los métodos para secuenciar el ADN de células individuales usando enfoques de secuenciación de próxima generación a menudo han sido laboriosos o limitados a multiplexar cientos de células o núcleos.
El biólogo molecular de la Facultad de Medicina Keck (Los Ángeles, CA, EUA) y sus colegas, realizaron una secuenciación superficial de una sola célula de ADN genómico a través de 1.475 células desde una línea celular, COLO829, hasta resolver la complejidad general y la clonalidad. Esta línea tumoral de melanoma se caracterizó previamente por múltiples tecnologías y es un punto de referencia para evaluar las alteraciones somáticas. Luego, las células se analizaron y se clasificaron en un citómetro de flujo usando excitación a 488 nm y activación de emisión verde en las células en la fase G1 del ciclo celular. Las células se contaron después de la clasificación para garantizar una concentración exacta.
Los científicos utilizaron una técnica emergente llamada “análisis del número de copias de células únicas”, desarrollado por 10X Genomics (Pleasanton, CA, EUA) con métodos novedosos de análisis que integraron estos resultados con los de los métodos históricos. La suspensión unicelular se procesó utilizando una solución de cromo CNV unicelular (10× Genomics). Las bibliotecas de secuenciación se cuantificaron mediante qPCR antes de la secuenciación en la plataforma Illumina (San Diego, CA, EUA) mediante la química NovaSeq S4 con 2 × 100 lecturas de pares. Las lecturas de pares se procesaron con la versión 1.0 de la línea de producción de ADN Cell Ranger de 10 × Genomics. Las células CNV unicelulares se extrajeron de un archivo BED generado por el Cell Ranger DNA Pipeline para 1.373 códigos de barras celulares.
El equipo informó que después de la secuenciación de células individuales poco profundas, primero identificaron al menos cuatro subclones principales mediante análisis discriminante de los componentes principales de los datos del número de copias de células individuales. Con base en el análisis de agrupamiento, el punto de ruptura y la pérdida de heterocigosidad de los datos agregados de los subclones, identificaron distintos eventos distintivos que se validaron dentro de la secuenciación masiva y el cariotipo espectral.
Enrique Velázquez-Villarreal, autor principal y profesor asistente de genómica traslacional, dijo: “En lugar de analizar el ADN del tejido que es el promedio de miles de células, analizamos el ADN individual de cerca de 1.500 células en un solo ensayo. Al estudiar el cáncer con esta resolución más alta, podemos descubrir información que la secuenciación masiva de baja resolución pasa por alto”. El estudio fue publicado el 25 de junio de 2020 en la revista Nature Communications Biology.
Enlace relacionado:
Facultad de Medicina Keck
10X Genomics
El paradigma estándar es la secuenciación masiva de ADN genómico derivado de millones de células heterogéneas. En la secuenciación masiva, la capacidad de resolver la subclonalidad se confunde al depender de la inferencia indirecta, lo que, con frecuencia, resulta en una vista de conjunto dominada por el clon mayoritario. Los métodos para secuenciar el ADN de células individuales usando enfoques de secuenciación de próxima generación a menudo han sido laboriosos o limitados a multiplexar cientos de células o núcleos.
El biólogo molecular de la Facultad de Medicina Keck (Los Ángeles, CA, EUA) y sus colegas, realizaron una secuenciación superficial de una sola célula de ADN genómico a través de 1.475 células desde una línea celular, COLO829, hasta resolver la complejidad general y la clonalidad. Esta línea tumoral de melanoma se caracterizó previamente por múltiples tecnologías y es un punto de referencia para evaluar las alteraciones somáticas. Luego, las células se analizaron y se clasificaron en un citómetro de flujo usando excitación a 488 nm y activación de emisión verde en las células en la fase G1 del ciclo celular. Las células se contaron después de la clasificación para garantizar una concentración exacta.
Los científicos utilizaron una técnica emergente llamada “análisis del número de copias de células únicas”, desarrollado por 10X Genomics (Pleasanton, CA, EUA) con métodos novedosos de análisis que integraron estos resultados con los de los métodos históricos. La suspensión unicelular se procesó utilizando una solución de cromo CNV unicelular (10× Genomics). Las bibliotecas de secuenciación se cuantificaron mediante qPCR antes de la secuenciación en la plataforma Illumina (San Diego, CA, EUA) mediante la química NovaSeq S4 con 2 × 100 lecturas de pares. Las lecturas de pares se procesaron con la versión 1.0 de la línea de producción de ADN Cell Ranger de 10 × Genomics. Las células CNV unicelulares se extrajeron de un archivo BED generado por el Cell Ranger DNA Pipeline para 1.373 códigos de barras celulares.
El equipo informó que después de la secuenciación de células individuales poco profundas, primero identificaron al menos cuatro subclones principales mediante análisis discriminante de los componentes principales de los datos del número de copias de células individuales. Con base en el análisis de agrupamiento, el punto de ruptura y la pérdida de heterocigosidad de los datos agregados de los subclones, identificaron distintos eventos distintivos que se validaron dentro de la secuenciación masiva y el cariotipo espectral.
Enrique Velázquez-Villarreal, autor principal y profesor asistente de genómica traslacional, dijo: “En lugar de analizar el ADN del tejido que es el promedio de miles de células, analizamos el ADN individual de cerca de 1.500 células en un solo ensayo. Al estudiar el cáncer con esta resolución más alta, podemos descubrir información que la secuenciación masiva de baja resolución pasa por alto”. El estudio fue publicado el 25 de junio de 2020 en la revista Nature Communications Biology.
Enlace relacionado:
Facultad de Medicina Keck
10X Genomics
Últimas Diagnóstico Molecular noticias
- Prueba de sangre predice con precisión el riesgo de cáncer de pulmón y reduce la necesidad de escaneos de TC
- Firma única de autoanticuerpos ayuda a diagnosticar la esclerosis múltiple años antes de la aparición de síntomas
- Prueba de sangre podría detectar cánceres asociados al VPH 10 años antes del diagnóstico clínico
- Un diagnóstico de bajo costo en el punto de atención ampliará el acceso a pruebas de enfermedades de transmisión sexual
- Prueba de orina analiza 18 genes para identificar el cáncer de próstata de alto grado
- Prueba en orina detecta cáncer de cabeza y cuello
- Prueba de sangre detecta y monitorea cáncer pulmonar de células pequeñas agresivo
- Ensayo de aprendizaje automático basado en sangre detecta de forma no invasiva el cáncer de ovario
- Ensayo de PCR simple diferencia con precisión entre los subtipos de cáncer de pulmón de células pequeñas
- Enfoque revolucionario de análisis de células T permite detección temprana del cáncer
- Prueba genética única podría acelerar el diagnóstico de trastornos raros del desarrollo
- Analizador de pruebas sindrómicas actualizado permite acceso remoto a resultados de pruebas
- Prueba de PCR para infecciones respiratorias y de garganta detecta múltiples patógenos con síntomas coincidentes
- Técnica de enriquecimiento de ácido nucleico circulante en sangre permite diagnóstico no invasivo del cáncer de hígado
- Primera prueba molecular aprobada por la FDA para detectar malaria en donantes de sangre podría mejorar seguridad del paciente
- Prueba de biomarcadores líquidos detecta enfermedades neurodegenerativas antes de que aparezcan síntomas
Canales
Química Clínica
ver canal
Espectrómetro de masas impreso en 3D para el punto de atención supera a los modelos de última generación
La espectrometría de masas es una técnica precisa para identificar los componentes químicos de una muestra y tiene un potencial significativo para monitorear estados de salud de enfermedades... Más.jpg)
Prueba biomédica POC hace girar una gota de agua utilizando ondas sonoras para detección del cáncer
Los exosomas, pequeñas biopartículas celulares que transportan un conjunto específico de proteínas, lípidos y materiales genéticos, desempeñan un papel... Más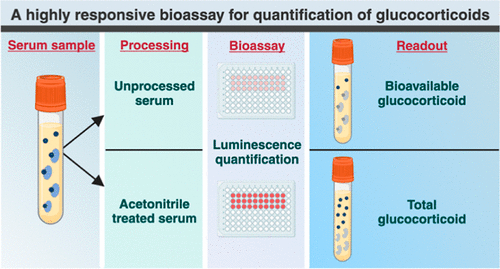
Prueba basada en células altamente confiable permite diagnóstico preciso de enfermedades endocrinas
Los métodos convencionales para medir el cortisol libre, la hormona del estrés del cuerpo, en la sangre o la saliva son bastante exigentes y requieren el procesamiento de muestras. Por lo tanto, el método... MásHematología
ver canal
Primera prueba NAT 4 en 1 para el cribado de arbovirus podría reducir el riesgo de infecciones transmitidas por transfusiones
Los arbovirus representan una amenaza emergente para la salud mundial, exacerbada por el cambio climático y el aumento de la conectividad mundial que está facilitando su propagación a nuevas regiones.... Más
Instrumento de próxima generación detecta trastornos de la hemoglobina en recién nacidos
Las hemoglobinopatías, las enfermedades hereditarias más extendidas a nivel mundial, afectan a alrededor del 7 % de la población como portadores, y el 2,7 % de los recién nacidos nacen con estas enfermedades.... Más
Prueba de sangre POC por punción digital determina riesgo de sepsis neutropénica en pacientes sometidos a quimioterapia
La neutropenia, una disminución de los neutrófilos (un tipo de glóbulo blanco crucial para combatir las infecciones), es un efecto secundario frecuente de ciertos tratamientos contra... Más
Primera prueba rápida y asequible para beta talasemia demuestra precisión diagnóstica del 99 %
Los trastornos de la hemoglobina se encuentran entre las enfermedades monogénicas más prevalentes a nivel mundial. Entre los diversos trastornos de la hemoglobina, la beta talasemia, un trastorno sanguíneo... MásInmunología
ver canal
Análisis de sangre para diagnóstico de rechazo celular después de trasplante de órganos podría reemplazar las biopsias quirúrgicas
Los órganos trasplantados enfrentan constantemente el riesgo de ser rechazados por el sistema inmunológico del receptor, que los diferencia de los órganos no propios mediante... Más
Herramienta de IA ajusta con precisión los medicamentos contra el cáncer con los pacientes utilizando información de cada célula tumoral
Las estrategias actuales para emparejar a los pacientes con cáncer con tratamientos específicos a menudo dependen de la secuenciación masiva de ADN y ARN tumoral, que proporciona un perfil promedio de... Más
Pruebas genéticas combinadas con la detección de fármacos personalizadas en muestras de tumores podrían revolucionar el tratamiento del cáncer
El tratamiento del cáncer generalmente se adhiere a un estándar de atención: regímenes establecidos y validados estadísticamente que son efectivos para la mayoría de los pacientes. Sin embargo, la variabilidad... Más
Método de prueba podría ayudar a más pacientes recibir tratamiento adecuado contra el cáncer
El tratamiento del cáncer no siempre es una solución única, pero el campo de la investigación del cáncer está dando grandes pasos para encontrar a los pacientes los tratamientos más eficaces para sus afecciones... MásMicrobiología
ver canal
Prueba de PCR múltiplex identifica el 95 % de los patógenos que causan la sepsis en una hora
La sepsis contribuye a una de cada tres muertes hospitalarias en los Estados Unidos y, a nivel mundial, el shock séptico conlleva una tasa de mortalidad del 30 al 40 %. El diagnóstico temprano... Más
Prueba de bacterias bucales podría predecir la progresión del cáncer de colon
El cáncer de colon, una enfermedad relativamente común pero difícil de diagnosticar, requiere confirmación mediante una colonoscopia o cirugía. Recientemente, ha habido... Más.jpg)
Firma metabólica unica podría permitir el diagnóstico de sepsis dentro de una hora de la extracción de sangre
La sepsis es una afección potencialmente mortal provocada por una respuesta extrema del cuerpo a una infección. Requiere intervención médica inmediata para evitar una posible muerte o daños duraderos.... Más
Innovadora plataforma de diagnóstico proporciona resultados de AST con velocidad sin precedentes
Una plataforma de diagnóstico innovadora que ofrece resultados de pruebas de susceptibilidad a los antibióticos (AST) con una velocidad sin precedentes puede convertirse en una herramienta importante para... MásPatología
ver canal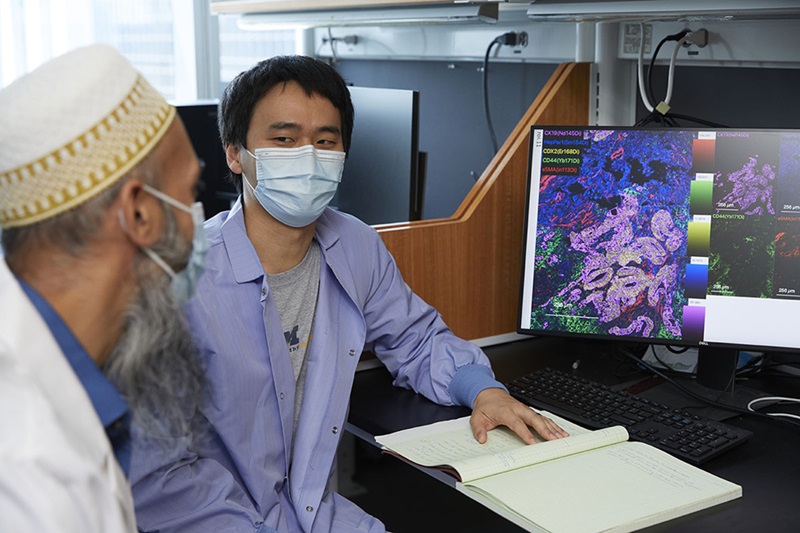
Análisis de tejido espacial identifica patrones asociados con la recaída del cáncer de ovario
El carcinoma de ovario seroso de alto grado es el tipo más letal de cáncer de ovario y plantea importantes desafíos de detección. Por lo general, los pacientes responden inicialmente... Más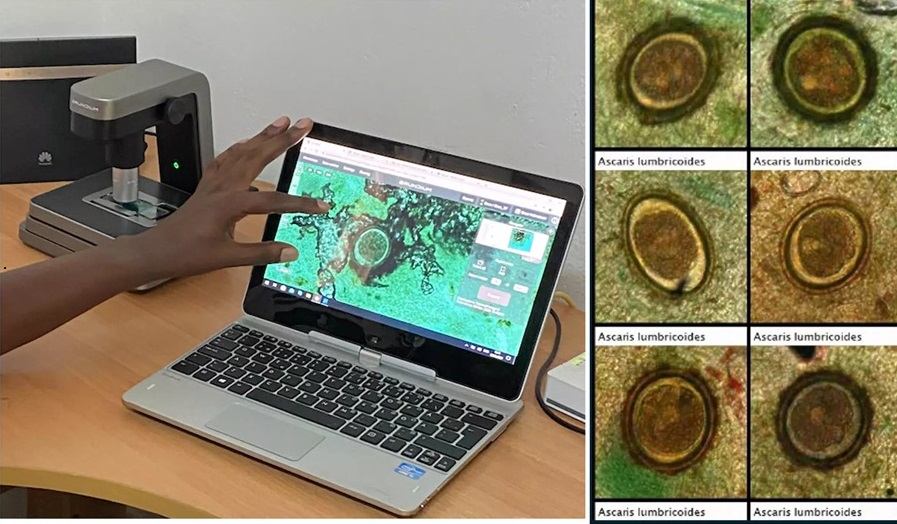
IA basada en imágenes se muestra prometedora para detección de parásitos en muestras de heces digitalizadas
Las infecciones por helmintos transmitidos por el suelo (STH), comúnmente conocidas como gusanos parásitos intestinales, se encuentran entre las enfermedades tropicales desatendidas más... Más
Algoritmos de inteligencia artificial potenciados por aprendizaje profundo mejoran la precisión en el diagnóstico de cáncer de piel.
Algoritmos de inteligencia artificial (IA) se utilizan cada vez más en diversos entornos clínicos, como la dermatología. Estos algoritmos se desarrollan entrenando una computadora... MásTecnología
ver canal
Nuevo sistema de diagnóstico de laboratorio en un chip iguala la precisión de las pruebas de PCR
Si bien las pruebas de PCR son el estándar de oro en cuanto a precisión para las pruebas de virología, tienen limitaciones como la complejidad, la necesidad de operadores de laboratorio capacitados y tiempos... Más
Biosensor de ADN permite diagnóstico temprano del cáncer de cuello uterino
El disulfuro de molibdeno (MoS2), reconocido por su potencial para formar nanoláminas bidimensionales como el grafeno, es un material que llama cada vez más la atención de la comunidad... Más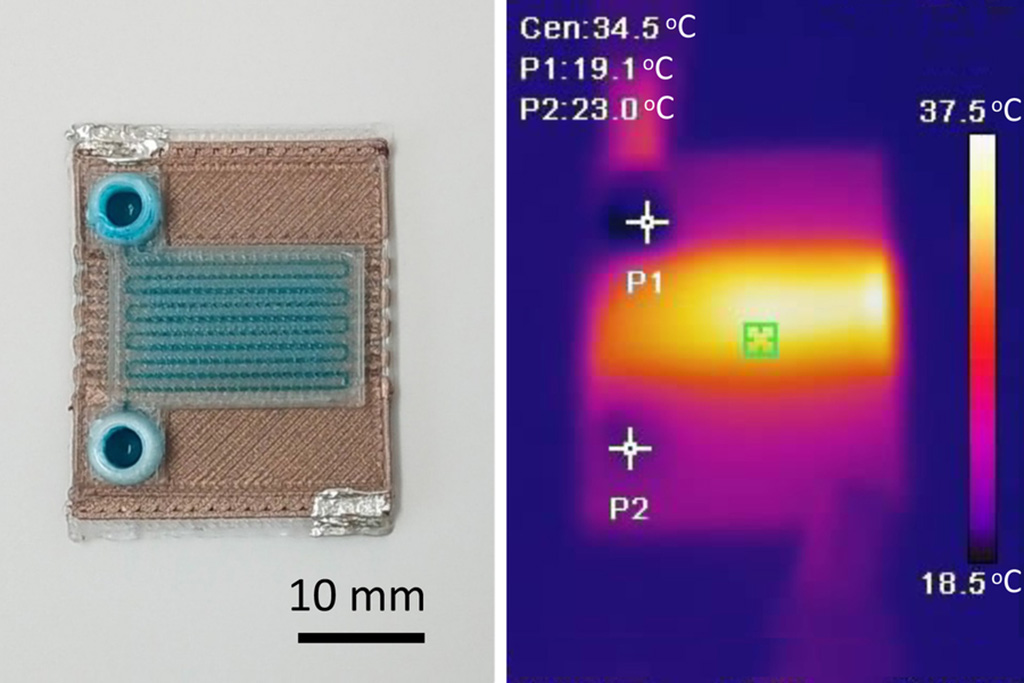
Dispositivos de microfluidos autocalentables pueden detectar enfermedades en pequeñas muestras de sangre o fluidos
Los microfluidos, que son dispositivos en miniatura que controlan el flujo de líquidos y facilitan reacciones químicas, desempeñan un papel clave en la detección de enfermedades... Más
Avance en tecnología de diagnóstico podría hacer que pruebas en el sitio sean ampliamente accesibles
Las pruebas caseras adquirieron una importancia significativa durante la pandemia de COVID-19, sin embargo, la disponibilidad de pruebas rápidas es limitada y la mayoría de ellas solo pueden conducir un... MásIndustria
ver canal
Congreso ECCMID cambia de nombre a ESCMID Global
En los últimos años, la Sociedad Europea de Microbiología Clínica y Enfermedades Infecciosas (ESCMID, Basilea, Suiza) ha evolucionado notablemente. La sociedad es ahora más... Más