Células T reconocen variantes recientes del SARS-CoV-2
|
Por el equipo editorial de LabMedica en español Actualizado el 06 Jun 2021 |
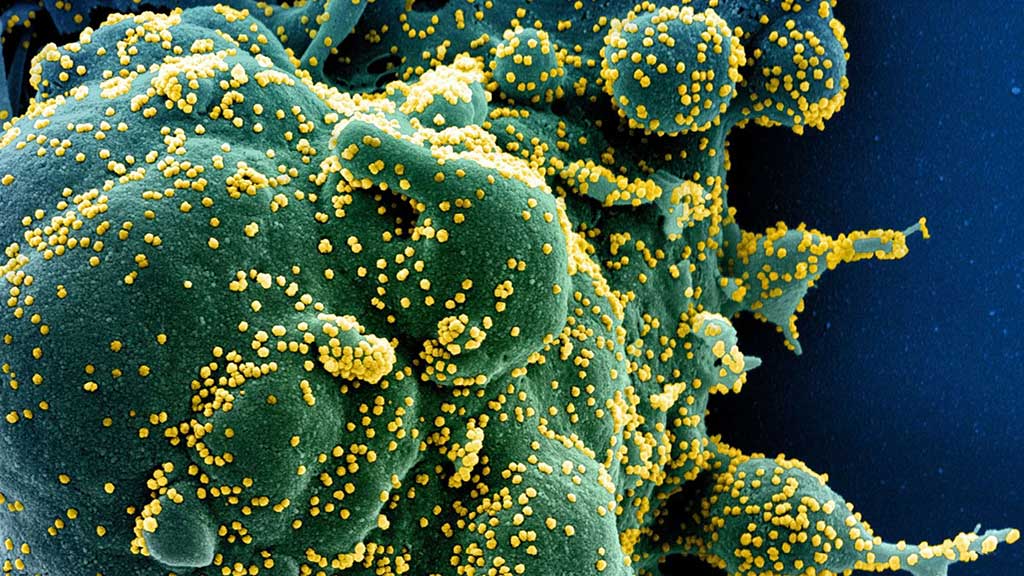
Imagen: los linfocitos de las células T inmunes pueden encontrar y destruir una célula (verde) infectada con SARS-CoV-2 (amarillo) y hacer copias (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los EUA)
Debido a la capacidad de corrección de la ARN polimerasa dependiente del ARN del coronavirus (CoV), la evolución de la población viral global del SARS-CoV-2 durante la pandemia actual ha sido relativamente limitada en comparación con otros virus ARN endémicos que no poseen esta capacidad.
A finales de 2020 se identificaron tres variantes distintas que poseían cada una cantidad significativamente mayor de polimorfismos de aminoácidos en asociación con las proteínas Spike en casos de COVID-19 en el Reino Unido (variante B.1.1.7), Sudáfrica (variante B.1.351) y Brasil (variante B.1.1.248). Todas estas variantes poseen la mutación N501Y en el dominio de unión al receptor (RBD) de la proteína Spike del SARS-CoV-2, un objetivo principal para la unión del anticuerpo neutralizante (NAb).
Un equipo colaborador de científicos dirigido por la Facultad de Medicina de la Universidad Johns Hopkins (Baltimore, MD, EUA), analizó muestras de células sanguíneas de 30 personas que habían contraído y se habían recuperado de la enfermedad del coronavirus 2019 (COVID-19) antes de la aparición de las variantes del virus. La mayoría (60%) de los individuos incluidos en el análisis eran hombres y las muestras se recolectaron una mediana de 42,5 días (rango intercuartílico, 37,5-48 días) desde el diagnóstico inicial. El equipo tenía como objetivo determinar si las células T CD8+ en la sangre aún podían reconocer las tres variantes principales del SARS-CoV-2. Se recolectaron y examinaron muestras de células mononucleares de sangre periférica (PBMC) de los pacientes en seis haplotipos de antígeno leucocitario humano (HLA) diferentes (HLAA*01:01, HLA-A*02:01, HLA-A03:01, HLA-A*11:01, HLA-A*24:02 y HLA-B*07:02).
Un método de coloración de tetrámero de péptido-MHC multiplexado permitió el cribado de 408 epítopos candidatos potenciales de SARS-CoV-2 para el reconocimiento de células T CD8+. Las células T también se evaluaron usando un panel fenotípico de 28 marcadores (Immunoscape Pte. Ltd, Singapur). Se encontraron un total de 52 respuestas de epítopos únicos y se dirigieron contra varias proteínas virales estructurales y no estructurales. Para los controles, se sondaron las células T CD8 + para determinar la reactividad de hasta 20 péptidos de control no relacionados con SARS-CoV-2 diferentes por HLA (epítopos derivados de Adenovirus, CMV, EBV, Influenza y MART-1).
El equipo informó que solo una mutación encontrada en la proteína Spike de B.1.35 se superpuso con un epítopo previamente identificado (1/52), sugiriendo que prácticamente todas las respuestas de células T anti-SARS-CoV-2 CD8+ deberían reconocer estas variantes recién descritas. Esta mutación es la mutación D80A en la proteína Spike y ocurre en el tercer residuo del epítopo RFDN VLPF. Este es un epítopo restringido a HLA*A24: 02 para el que se detectó una respuesta de linfocitos T CD8+ en 1/5 individuos HLA*A24:02+, y con una frecuencia baja (0,005 del total de linfocitos T CD8 +), lo que indica que no es un epítopo de alta prevalencia.
Los autores concluyeron que sus datos resaltaban el papel significativo potencial de una respuesta de células T multiepítopo en la limitación del escape viral y en parte medían la protección de la enfermedad causada por las variantes del SARS-CoV-2. Es importante que las vacunas utilizadas para campañas generalizadas generen fuertes respuestas de células T multivalentes además de anticuerpos neutralizantes (Nab) y otras respuestas humorales para optimizar la eficacia contra el SARS-CoV-2 actual y las cepas emergentes. Será importante continuar monitoreando la amplitud, magnitud y durabilidad de las respuestas de células T anti-SARS-CoV-2 en individuos recuperados y vacunados como parte de cualquier evaluación para determinar si se necesitan vacunas de refuerzo. El estudio fue publicado el 30 de marzo de 2021 en la revista Open Forum Infectious Diseases.
Enlace relacionado:
Facultad de Medicina de la Universidad Johns Hopkins
Immunoscape Pte. Ltd
A finales de 2020 se identificaron tres variantes distintas que poseían cada una cantidad significativamente mayor de polimorfismos de aminoácidos en asociación con las proteínas Spike en casos de COVID-19 en el Reino Unido (variante B.1.1.7), Sudáfrica (variante B.1.351) y Brasil (variante B.1.1.248). Todas estas variantes poseen la mutación N501Y en el dominio de unión al receptor (RBD) de la proteína Spike del SARS-CoV-2, un objetivo principal para la unión del anticuerpo neutralizante (NAb).
Un equipo colaborador de científicos dirigido por la Facultad de Medicina de la Universidad Johns Hopkins (Baltimore, MD, EUA), analizó muestras de células sanguíneas de 30 personas que habían contraído y se habían recuperado de la enfermedad del coronavirus 2019 (COVID-19) antes de la aparición de las variantes del virus. La mayoría (60%) de los individuos incluidos en el análisis eran hombres y las muestras se recolectaron una mediana de 42,5 días (rango intercuartílico, 37,5-48 días) desde el diagnóstico inicial. El equipo tenía como objetivo determinar si las células T CD8+ en la sangre aún podían reconocer las tres variantes principales del SARS-CoV-2. Se recolectaron y examinaron muestras de células mononucleares de sangre periférica (PBMC) de los pacientes en seis haplotipos de antígeno leucocitario humano (HLA) diferentes (HLAA*01:01, HLA-A*02:01, HLA-A03:01, HLA-A*11:01, HLA-A*24:02 y HLA-B*07:02).
Un método de coloración de tetrámero de péptido-MHC multiplexado permitió el cribado de 408 epítopos candidatos potenciales de SARS-CoV-2 para el reconocimiento de células T CD8+. Las células T también se evaluaron usando un panel fenotípico de 28 marcadores (Immunoscape Pte. Ltd, Singapur). Se encontraron un total de 52 respuestas de epítopos únicos y se dirigieron contra varias proteínas virales estructurales y no estructurales. Para los controles, se sondaron las células T CD8 + para determinar la reactividad de hasta 20 péptidos de control no relacionados con SARS-CoV-2 diferentes por HLA (epítopos derivados de Adenovirus, CMV, EBV, Influenza y MART-1).
El equipo informó que solo una mutación encontrada en la proteína Spike de B.1.35 se superpuso con un epítopo previamente identificado (1/52), sugiriendo que prácticamente todas las respuestas de células T anti-SARS-CoV-2 CD8+ deberían reconocer estas variantes recién descritas. Esta mutación es la mutación D80A en la proteína Spike y ocurre en el tercer residuo del epítopo RFDN VLPF. Este es un epítopo restringido a HLA*A24: 02 para el que se detectó una respuesta de linfocitos T CD8+ en 1/5 individuos HLA*A24:02+, y con una frecuencia baja (0,005 del total de linfocitos T CD8 +), lo que indica que no es un epítopo de alta prevalencia.
Los autores concluyeron que sus datos resaltaban el papel significativo potencial de una respuesta de células T multiepítopo en la limitación del escape viral y en parte medían la protección de la enfermedad causada por las variantes del SARS-CoV-2. Es importante que las vacunas utilizadas para campañas generalizadas generen fuertes respuestas de células T multivalentes además de anticuerpos neutralizantes (Nab) y otras respuestas humorales para optimizar la eficacia contra el SARS-CoV-2 actual y las cepas emergentes. Será importante continuar monitoreando la amplitud, magnitud y durabilidad de las respuestas de células T anti-SARS-CoV-2 en individuos recuperados y vacunados como parte de cualquier evaluación para determinar si se necesitan vacunas de refuerzo. El estudio fue publicado el 30 de marzo de 2021 en la revista Open Forum Infectious Diseases.
Enlace relacionado:
Facultad de Medicina de la Universidad Johns Hopkins
Immunoscape Pte. Ltd
Últimas COVID-19 noticias
- Inmunosensor nuevo allana el camino para pruebas rápidas POC para COVID-19 y enfermedades infecciosas emergentes
- Encuentran etiologías de COVID prolongada en muestras de sangre con infección aguda
- Dispositivo novedoso detecta anticuerpos contra la COVID-19 en cinco minutos
- Prueba para COVID-19 mediante CRISPR detecta SARS-CoV-2 en 30 minutos usando tijeras genéticas
- Asocian disbiosis del microbioma intestinal con la COVID-19
- Validan prueba rápida novedosa de antígeno para el SARS-CoV-2 con respecto a su exactitud diagnóstica
- Prueba nueva COVID + Influenza + VSR ayudará a estar preparados para la ‘tripledemia’
- IA elimina las conjeturas de las pruebas de flujo lateral
- Prueba de antígeno del SARS-CoV-2 más rápida, jamás diseñada, permite realizar pruebas de COVID-19 no invasivas en cualquier entorno
- Pruebas rápidas de antígeno detectan las variantes ómicron, delta del SARS-CoV-2
- Prueba en sangre realizada durante la infección inicial predice el riesgo de COVID prolongada
- Investigadores afirman que hay que crear “reservistas” de laboratorio para responder más rápidamente a la próxima pandemia
- Estudio encuentra que los profesionales sanitarios mostraron mayor interés en tecnologías POC durante la pandemia
- Plataforma de análisis de bajo costo para la COVID-19 combina sensibilidad de la PCR y velocidad de pruebas de antígeno
- Prueba de sangre por punción digital identifica inmunidad a la COVID-19
- Kit de prueba rápida determina inmunidad contra la COVID-19 y sus variantes
Canales
Química Clínica
ver canal
Herramienta química a nanoescala 'brillantemente luminosa' mejora detección de enfermedades
Miles de moléculas brillantes disponibles comercialmente, conocidas como fluoróforos, se utilizan comúnmente en imágenes médicas, detección de enfermedades, marcado... Más
Prueba de detección portátil económica transforma detección de enfermedades renales
Millones de personas padecen enfermedad renal, que a menudo permanece sin diagnosticar hasta que alcanza una etapa crítica. Esta epidemia silenciosa no solo disminuye la calidad de vida de los afectados,... MásDiagnóstico Molecular
ver canal
Prueba de biomarcadores sanguíneos podría detectar predisposición genética al Alzheimer
Nuevos medicamentos para la enfermedad de Alzheimer, la forma más común de demencia, están ahora disponibles. Estos tratamientos, conocidos como "anticuerpos amiloides",... Más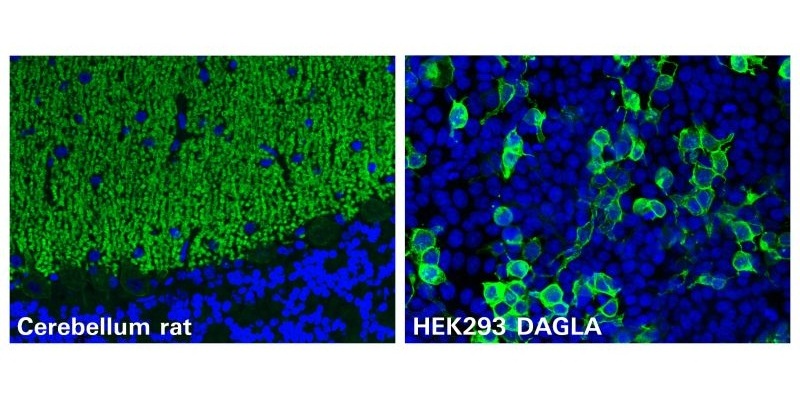
Se descubre nuevo autoanticuerpo contra DAGLA en cerebelitis
Las ataxias cerebelosas autoinmunes son trastornos muy incapacitantes que se caracterizan por una disminución de la habilidad para coordinar el movimiento muscular. Los autoanticuerpos cerebelosos... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásMicrobiología
ver canal
Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
La tuberculosis (TB) sigue siendo la enfermedad infecciosa más mortal a nivel mundial, afectando a aproximadamente 10 millones de personas al año. En 2021, alrededor de 4,2 millones de casos... Más
Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
Las infecciones resistentes a los medicamentos, en particular las causadas por bacterias mortales como la tuberculosis y el estafilococo, se están convirtiendo rápidamente en una emergencia... MásPatología
ver canal
Nuevo método basado en aprendizaje automático detecta contaminación microbiana en cultivos celulares
La terapia celular tiene un gran potencial en el tratamiento de enfermedades como el cáncer, las enfermedades inflamatorias y los trastornos degenerativos crónicos mediante la manipulación o el reemplazo... Más
Nuevo método con corrección de errores detecta cáncer únicamente en muestras de sangre
La tecnología de biopsia líquida, que se basa en análisis de sangre para la detección temprana del cáncer y el seguimiento de la carga oncológica en los pacientes,... MásTecnología
ver canal
Tecnología de microchip desechable podría detectar selectivamente VIH en muestras de sangre completa
A finales de 2023, aproximadamente 40 millones de personas en todo el mundo vivían con VIH, y alrededor de 630.000 personas murieron por enfermedades relacionadas con el sida ese mismo año.... Más
Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
El dolor crónico es una afección generalizada que sigue siendo difícil de controlar, y los métodos clínicos existentes para su tratamiento se basan en gran medida en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más