Descubren biomarcador para patógeno hipervirulento
|
Por el equipo editorial de LabMedica en español Actualizado el 07 Aug 2018 |
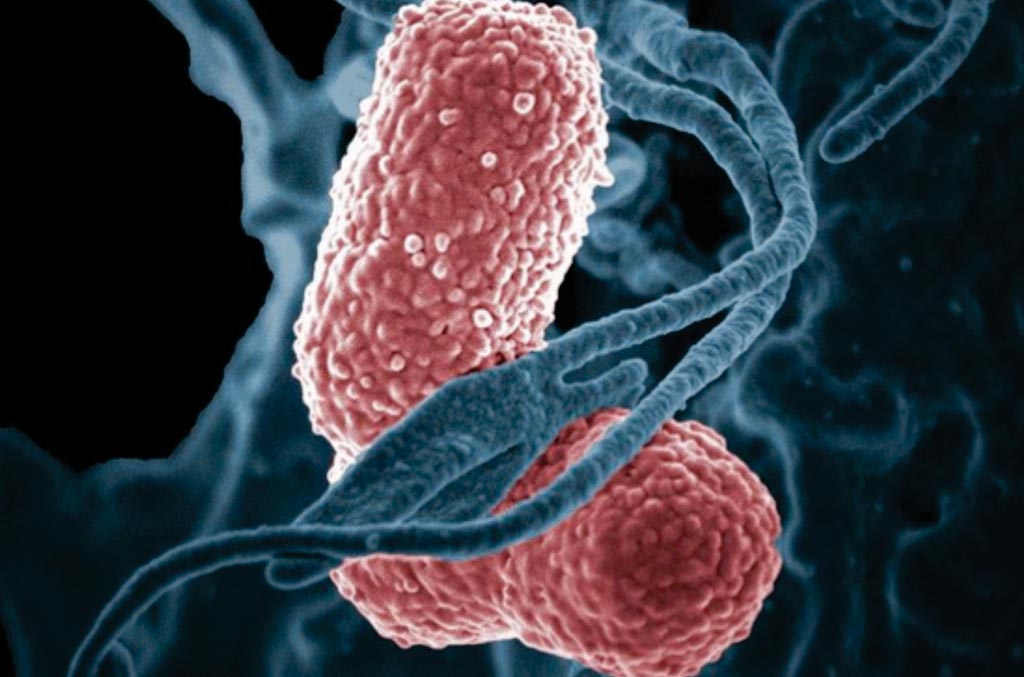
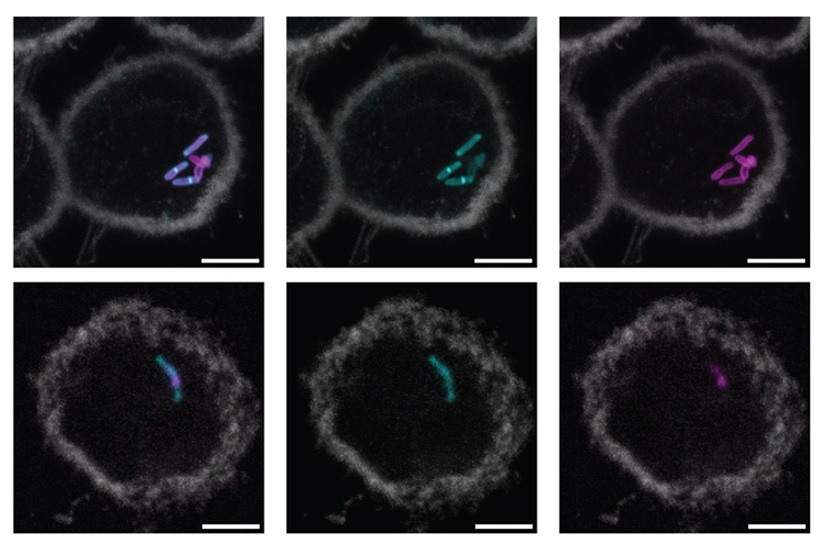
Imagen: Un neutrófilo humano (gris) interactuando con Klebsiella pneumoniae (rosa). Una versión hipervirulenta de este patógeno suscita una preocupación creciente debido a los informes recientes que describen cepas hipervirulentas que son resistentes a los antibióticos (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas).
No existe un método preciso para diferenciar entre la cepa hipervirulenta y la cepa clásica de Klebsiella pneumoniae (cKp), que se observa con mayor frecuencia en el hemisferio occidental, es menos virulenta y generalmente causa infecciones en el entorno hospitalario.
Se han descubierto varios biomarcadores que pueden identificar con exactitud la K. pneumoniae hipervirulenta (hvKp), un patógeno que infecta a personas completamente sanas y puede causar ceguera en un día e infecciones carnívoras, abscesos cerebrales y la muerte en pocos días.
Un equipo internacional de científicos liderado por investigadores de la Universidad de Buffalo (Buffalo, NY, EUA) identificó un biomarcador para diferenciar las cepas de hvKp de las cepas de cKp. Escogieron utilizar datos clínicos para desarrollar cohortes de cepas para evaluación, dado que la inclusión de cualquier información genotípica o fenotípica bacteriana en la definición de cohortes de cepas podría introducir sesgo. La cohorte rica en hvKp consistió en 85 cepas aisladas de pacientes no identificados de Taiwán y EUA Dado que la mayoría de las infecciones por K. pneumoniae en Norteamérica y el Reino Unido presumiblemente se deben a cepas de cKp, la cohorte de cepas rica en 90 cKp se generó a partir de aislados de sangre no identificados elegidos al azar de diferentes países.
Los aislados del estudio se evaluaron para la presencia de 10 genes y marcadores para tipos de cápsulas. Estos incluyen varios genes ubicados en el plásmido de virulencia, que se ha demostrado experimentalmente que contribuyen a la hipervirulencia en los modelos de infección in vivo. Se evaluaron dos rasgos fenotípicos hipotéticamente discriminatorios. La detección basada en reacciones de cadena de polimerasa (PCR) de varios tipos de cápsulas se realizó usando el instrumento Applied Biosystems GeneAmp PCR system 9700 (Foster City, CA, EUA).
Los científicos descubrieron que los genes peg-344, iroB, iucA, prmpA y prmpA2 demostraron una exactitud diagnóstica> 0,95 para identificar cepas en la cohorte rica en hvKp. La producción cuantitativa de sideróforos de ≥ 30 μg/mL también predijo fuertemente las cepas como miembros de la cohorte rica en hvKp (exactitud 0,96) y exhibió una relación de riesgo de 31,7 para enfermedad grave o muerte. La prueba de cuerda, un marcador ampliamente utilizado para las cepas hvKp, tuvo un desempeño inferior, logrando una exactitud de solo 0,90.
Thomas A. Russo, MD, profesor de medicina y autor principal del estudio dijo: “La ventaja de identificar estos biomarcadores genéticos es que se pueden convertir en pruebas rápidas de ácidos nucleicos y, si la Administración de Medicamentos y Alimentos de los EUA los aprueba, podrían proporcionar a los médicos un método exacto para determinar rápidamente si un paciente padece una infección debido a la cepa clásica o a la hipervirulenta”. El estudio fue publicado el 20 de junio de 2018 en la revista Journal of Clinical Microbiology.
Se han descubierto varios biomarcadores que pueden identificar con exactitud la K. pneumoniae hipervirulenta (hvKp), un patógeno que infecta a personas completamente sanas y puede causar ceguera en un día e infecciones carnívoras, abscesos cerebrales y la muerte en pocos días.
Un equipo internacional de científicos liderado por investigadores de la Universidad de Buffalo (Buffalo, NY, EUA) identificó un biomarcador para diferenciar las cepas de hvKp de las cepas de cKp. Escogieron utilizar datos clínicos para desarrollar cohortes de cepas para evaluación, dado que la inclusión de cualquier información genotípica o fenotípica bacteriana en la definición de cohortes de cepas podría introducir sesgo. La cohorte rica en hvKp consistió en 85 cepas aisladas de pacientes no identificados de Taiwán y EUA Dado que la mayoría de las infecciones por K. pneumoniae en Norteamérica y el Reino Unido presumiblemente se deben a cepas de cKp, la cohorte de cepas rica en 90 cKp se generó a partir de aislados de sangre no identificados elegidos al azar de diferentes países.
Los aislados del estudio se evaluaron para la presencia de 10 genes y marcadores para tipos de cápsulas. Estos incluyen varios genes ubicados en el plásmido de virulencia, que se ha demostrado experimentalmente que contribuyen a la hipervirulencia en los modelos de infección in vivo. Se evaluaron dos rasgos fenotípicos hipotéticamente discriminatorios. La detección basada en reacciones de cadena de polimerasa (PCR) de varios tipos de cápsulas se realizó usando el instrumento Applied Biosystems GeneAmp PCR system 9700 (Foster City, CA, EUA).
Los científicos descubrieron que los genes peg-344, iroB, iucA, prmpA y prmpA2 demostraron una exactitud diagnóstica> 0,95 para identificar cepas en la cohorte rica en hvKp. La producción cuantitativa de sideróforos de ≥ 30 μg/mL también predijo fuertemente las cepas como miembros de la cohorte rica en hvKp (exactitud 0,96) y exhibió una relación de riesgo de 31,7 para enfermedad grave o muerte. La prueba de cuerda, un marcador ampliamente utilizado para las cepas hvKp, tuvo un desempeño inferior, logrando una exactitud de solo 0,90.
Thomas A. Russo, MD, profesor de medicina y autor principal del estudio dijo: “La ventaja de identificar estos biomarcadores genéticos es que se pueden convertir en pruebas rápidas de ácidos nucleicos y, si la Administración de Medicamentos y Alimentos de los EUA los aprueba, podrían proporcionar a los médicos un método exacto para determinar rápidamente si un paciente padece una infección debido a la cepa clásica o a la hipervirulenta”. El estudio fue publicado el 20 de junio de 2018 en la revista Journal of Clinical Microbiology.
Últimas Microbiología noticias
- Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
Ensayo automatizado y descentralizado de NGS deADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de ... MásDiagnóstico Molecular
ver canal
Innovadora prueba de diagnóstico molecular señala con precisión principal causa genética de EPOC
La enfermedad pulmonar obstructiva crónica (EPOC) y la deficiencia de alfa-1 antitripsina (DAAT) son afecciones que pueden causar dificultades respiratorias, pero difieren en su origen y herencia.... Más
Prueba diagnóstica de sangre detecta espondiloartritis axial
La espondiloartritis axial (EspAax) es una enfermedad autoinmune inflamatoria crónica que suele afectar a las personas durante sus años más productivos, y cuyos síntomas suelen manifestarse antes de los 45 años.... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más