Prueba rápida detecta resistencia móvil a la colistina en aislados de bacterias
|
Por el equipo editorial de LabMedica en español Actualizado el 06 Mar 2017 |

Imagen: Enriquecimiento de cultivos bacterianos (Fotografía cortesía de DZIF y JLU).
Los científicos han evaluado un ensayo basado en LAMP, disponible comercialmente, para la detección rápida del gen, móvil codificado por plásmidos mcr-1 de resistencia a la colistina, en aislados de Enterobacteriaceae. Con un mayor desarrollo y evaluación, también podrá ser utilizado en los hospitales y para el ganado.
Científicos del Centro Alemán de Investigación de Infecciones (DZIF, Braunschweig, Alemania) y la Universidad Justus Liebig (JLU) Giessen (también Universidad de Giessen, Giessen, Alemania), evaluaron la prueba, que detecta el gen mcr-1, en 20 minutos.
La colistina se utiliza como un antibiótico de último recurso para los patógenos multirresistentes, especialmente en los hospitales. Sin embargo, algunas bacterias intestinales, se han vuelto insensibles a la colistina debido al gen móvil de resistencia a la colistina, mcr-1. A principios de 2016, fueron detectadas, por primera vez, las bacterias portadoras de mcr-1, en Alemania y existe una creciente preocupación de que pueda convertirse en una súperbacteria contra la cual incluso los antibióticos de emergencia ya no son eficaces. El riesgo de propagación adicional de esta resistencia, a la colistina, es elevado porque se produce a través de plásmidos, que son transferidos entre diferentes tipos de bacterias con relativa facilidad.
“Es importante confirmar el gen mcr-1, de resistencia móvil, lo más rápidamente posible para evitar su propagación”, enfatizó Linda Falgenhauer, científica del DZIF en la Universidad de Giessen y co-primera autora de este estudio. Ella, con colegas de la Universidad de Giessen y de la asociación de investigación, RESET, ensayo una prueba de resistencia genotípica rápida para la colistina que ya está disponible comercialmente. “Esta es la única manera con la que la resistencia móvil se puede diferenciar de la resistencia común, porque fenotípicamente son iguales”, dijo el co-primer autor, Can Imirzalioglu, científico del DZIF en la Universidad de Giessen.
Para la evaluación de esta prueba, AmplexBiosystems GmbH (Giessen, Alemania) proporcionó los kits de forma gratuita. Los científicos examinaron 104 aislamientos bacterianos de animales, seres humanos y el medio ambiente: los resultados fueron comparados con los de secuenciación completa del genoma o PCR, y demostraron una sensibilidad y especificidad del 100%. Por lo tanto, la prueba podría diferenciar claramente entre la resistencia común a la colistina y la resistencia móvil, localizada en los plásmidos. Sin embargo, la aplicación de la prueba directamente a muestras clínicas todavía no ha sido evaluada, sólo ha sido aplicada a cultivos bacterianos.
“Los resultados están disponibles en solamente 20 minutos”, dijo la coautora, Judith Schmiedel, del equipo de Giessen. “Con el procedimiento convencional, se necesitan varias horas para obtener los resultados. Además, el sistema es muy sencillo, por lo que se debe desarrollar aún más para su uso futuro en hospitales, así como para la ganadería y la producción de alimentos”. “Con una colaboración interdisciplinaria, estamos siguiendo el enfoque One Health, que tiene en cuenta las conexiones sistémicas entre los seres humanos, los animales, el medio ambiente y la salud con el fin de combatir la resistencia a los antibióticos”, dijo el profesor Trinad Chakraborty, de la JLU Giessen y coordinador del sitio asociado del DZIF, Giessen-Marburg-Langen.
El estudio, por Imirzalioglu C et al, fue publicado, en línea, antes de impresión el 30 de enero de 2017 en la revista Antimicrobial Agents and Chemotherapy.
Últimas Microbiología noticias
- Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
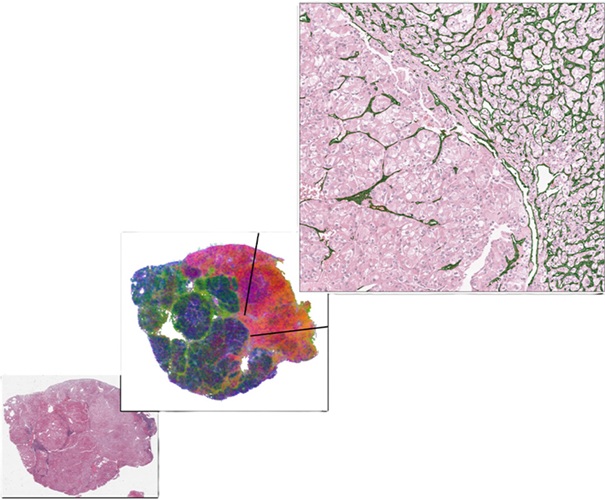
Ensayo automatizado y descentralizado de NGS deADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de ... MásDiagnóstico Molecular
ver canal
Innovadora prueba de diagnóstico molecular señala con precisión principal causa genética de EPOC
La enfermedad pulmonar obstructiva crónica (EPOC) y la deficiencia de alfa-1 antitripsina (DAAT) son afecciones que pueden causar dificultades respiratorias, pero difieren en su origen y herencia.... Más
Prueba diagnóstica de sangre detecta espondiloartritis axial
La espondiloartritis axial (EspAax) es una enfermedad autoinmune inflamatoria crónica que suele afectar a las personas durante sus años más productivos, y cuyos síntomas suelen manifestarse antes de los 45 años.... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más