Dispositivo de microfluidos captura y aísla bacterias intestinales de crecimiento lento
|
Por el equipo editorial de LabMedica en español Actualizado el 13 Aug 2014 |
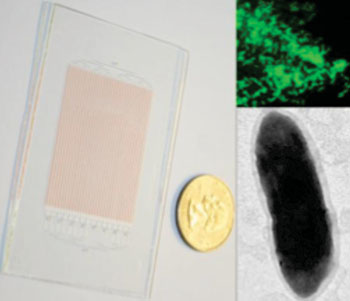
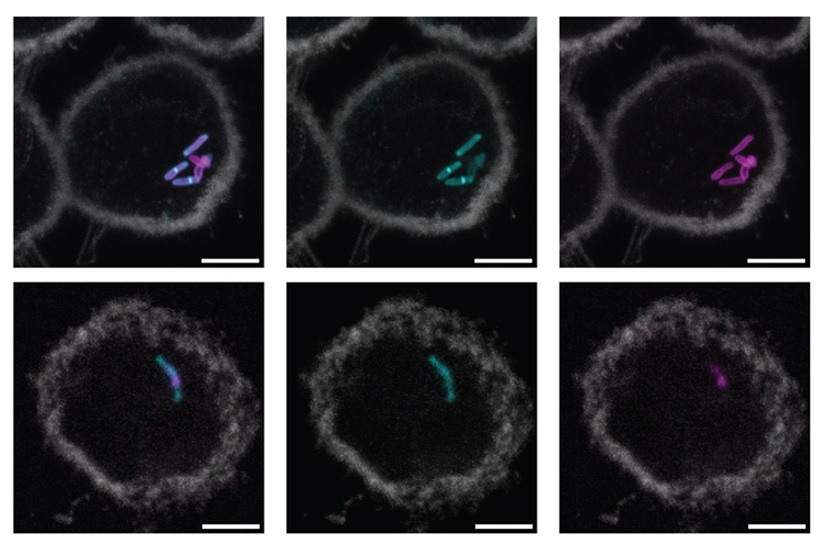
Imagen: El SlipChip de vidrio para el crecimiento de microbios, mostrado al lado de una moneda de 25 centavos norteamericana (izquierda); la imagen de hibridación fluorescente in situ del organismo objetivo (derecha, arriba); imagen de microscopía electrónica de transmisión de una sola célula del organismo objetivo (derecha, abajo) (Fotografía cortesía del Instituto Tecnológico de California).
Los microbiólogos han usado un novedoso método lab en un chip para aislar y cultivar bacterias fastidiosas, de lento crecimiento, provenientes del tracto digestivo humano.
La mayoría de los microbios que comprenden el bioma intestinal humano no han sido cultivados, debido en parte, a las dificultades de identificar tanto las condiciones de crecimiento apropiadas, como de caracterizar y aislar cada especie.
Los investigadores en el Instituto Tecnológico de California (Pasadena, EUA) desarrollaron un método genéticamente dirigido, basado en microfluidos para superar estos problemas. Su dispositivo SlipChip fue construido a partir de dos láminas de vidrio, cada una del tamaño de una tarjeta de crédito, que fueron grabadas con pequeñas ranuras que se convirtieron en canales cuando las superficies ranuradas fueron apiladas una encima de otra. Cuando una muestra, tal como un surtido mixto de especies bacterianas de una biopsia de la colonoscopia, se aplica al dispositivo, los canales interconectados del chip superior convierten los canales en pozos individuales, con cada pozo conteniendo, idealmente, un solo microbio. Una vez secuestrada en un pozo aislado, cada bacteria individual era capaz de dividirse y crecer sin tener que competir por recursos con otros tipos de microbios de crecimiento más rápido.
La belleza del sistema era que cada pozo podía ser dividido en dos compartimentos. Un compartimiento fue utilizado para estudios de secuenciación de ADN y de mapeo, mientras que el otro mantuvo un ejemplo vivo, del microbio, para cultivos y estudios posteriores.
Los investigadores validaron este método cultivando una bacteria a partir de una biopsia cecal humana. El mapeo genético del organismo mostró que se trataba de un representante de un género previamente no identificado de la familia Ruminococcaceae y que su firma genética había sido incluida de la lista de los “más buscados” del Proyecto, del microbioma humano, en el grupo de alta prioridad, de los Institutos Nacionales de Salud de Estados Unidos.
“A pesar de que una secuencia genómica del nuevo organismo es una herramienta útil, se necesitan más estudios para saber cómo esta especie de microbio está implicada en la salud humana”, dijo el autor principal, el Doctor Rustem Ismagilov, profesor de química e ingeniería química en el Instituto Tecnológico de California.
El estudio fue publicado en la edición digital del 25 de junio, 2014, de la revista Proceedings of the National Academy of Sciences of the United States of America (PNAS).
Enlace relacionado:
California Institute of Technology
La mayoría de los microbios que comprenden el bioma intestinal humano no han sido cultivados, debido en parte, a las dificultades de identificar tanto las condiciones de crecimiento apropiadas, como de caracterizar y aislar cada especie.
Los investigadores en el Instituto Tecnológico de California (Pasadena, EUA) desarrollaron un método genéticamente dirigido, basado en microfluidos para superar estos problemas. Su dispositivo SlipChip fue construido a partir de dos láminas de vidrio, cada una del tamaño de una tarjeta de crédito, que fueron grabadas con pequeñas ranuras que se convirtieron en canales cuando las superficies ranuradas fueron apiladas una encima de otra. Cuando una muestra, tal como un surtido mixto de especies bacterianas de una biopsia de la colonoscopia, se aplica al dispositivo, los canales interconectados del chip superior convierten los canales en pozos individuales, con cada pozo conteniendo, idealmente, un solo microbio. Una vez secuestrada en un pozo aislado, cada bacteria individual era capaz de dividirse y crecer sin tener que competir por recursos con otros tipos de microbios de crecimiento más rápido.
La belleza del sistema era que cada pozo podía ser dividido en dos compartimentos. Un compartimiento fue utilizado para estudios de secuenciación de ADN y de mapeo, mientras que el otro mantuvo un ejemplo vivo, del microbio, para cultivos y estudios posteriores.
Los investigadores validaron este método cultivando una bacteria a partir de una biopsia cecal humana. El mapeo genético del organismo mostró que se trataba de un representante de un género previamente no identificado de la familia Ruminococcaceae y que su firma genética había sido incluida de la lista de los “más buscados” del Proyecto, del microbioma humano, en el grupo de alta prioridad, de los Institutos Nacionales de Salud de Estados Unidos.
“A pesar de que una secuencia genómica del nuevo organismo es una herramienta útil, se necesitan más estudios para saber cómo esta especie de microbio está implicada en la salud humana”, dijo el autor principal, el Doctor Rustem Ismagilov, profesor de química e ingeniería química en el Instituto Tecnológico de California.
El estudio fue publicado en la edición digital del 25 de junio, 2014, de la revista Proceedings of the National Academy of Sciences of the United States of America (PNAS).
Enlace relacionado:
California Institute of Technology
Últimas Microbiología noticias
- Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
Ensayo automatizado y descentralizado de NGS deADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de ... MásDiagnóstico Molecular
ver canal
Innovadora prueba de diagnóstico molecular señala con precisión principal causa genética de EPOC
La enfermedad pulmonar obstructiva crónica (EPOC) y la deficiencia de alfa-1 antitripsina (DAAT) son afecciones que pueden causar dificultades respiratorias, pero difieren en su origen y herencia.... Más
Prueba diagnóstica de sangre detecta espondiloartritis axial
La espondiloartritis axial (EspAax) es una enfermedad autoinmune inflamatoria crónica que suele afectar a las personas durante sus años más productivos, y cuyos síntomas suelen manifestarse antes de los 45 años.... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más