Prueba no invasiva de orina para la predicción de la falla del trasplante renal
|
Por el equipo editorial de LabMedica en español Actualizado el 20 Apr 2020 |

Imagen: Las tiras de flujo lateral exponen tres muestras de pacientes que son negativas para el virus BK (13,14,15) y tres muestras de pacientes que son positivas (16,17,18). La presencia de la banda superior indica un resultado positivo en la prueba (Fotografía cortesía del Dr. Michael Kaminski, Centro Max Delbrück de Medicina Molecular)
Una prueba no invasiva que predice un posible fracaso del trasplante de riñón combina la herramienta de edición de genes, CRISPR, con un procedimiento de lectura visual de flujo lateral para analizar muestras de orina de los receptores de trasplante.
Los investigadores del Centro Max Delbrück de Medicina Molecular (Berlín, Alemania) diseñaron un sistema nuevo de análisis que detecta dos virus oportunistas comunes que infectan a los pacientes con trasplante de riñón, el citomegalovirus (CMV) y el poliomavirus BK (BKV), y para el ARNm de CXCL9, cuya expresión aumenta durante el rechazo celular agudo del trasplante de riñón.
La primera etapa del ensayo amplifica el ADN o ARN en la muestra de orina para que CRISPR lo pueda detectar. Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente y separadas entre sí) son segmentos de ADN procariota que contienen repeticiones cortas de secuencias de bases. Cada repetición es seguida por segmentos cortos de “ADN espaciador” de exposiciones previas a un virus o plásmido bacteriano. Desde 2013, se ha utilizado el sistema CRISPR/Cas9 en la investigación para la edición de genes (agregando, interrumpiendo o cambiando la secuencia de genes específicos) y la regulación de genes. A través de la administración de la enzima Cas9 y los ARN guía apropiados (sgARN) a una célula, el genoma del organismo se puede cortar en cualquier ubicación deseada. El sistema convencional CRISPR/Cas9 de Streptococcus pyogenes se compone de dos partes: la enzima Cas9, que escinde la molécula de ADN y guías de ARN específicas que guían la proteína Cas9 al gen objetivo en una cadena de ADN.
Los esfuerzos computacionales recientes para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida contra el ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación de ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, dirigido al ARN guiado por el ARN, que tiene un gran potencial para aplicaciones precisas, robustas y escalables de selección de ARN y de ARN guiado.
Después del paso de amplificación de ADN/ARN, los investigadores aplicaron el protocolo SHERLOCK de SHERLOCK Biotechnologies (Cambridge, MA, EUA). Este es un método usado para la detección de moléculas individuales de objetivos de ácido nucleico y significa desBLOQueo (unLOCKing, en inglés) del Reportero Enzimático Específico de Alta Sensibilidad. El método funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también puede ser cuantificada. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple prueba de tira de papel, en equipos de laboratorio, o para proporcionar una lectura electroquímica que se pueda leer con un teléfono móvil.
El análisis de flujo lateral usando una tira de papel se utilizó para convertir el contenido de la muestra tratada en un resultado fácilmente visualizable. Una línea que aparece en la tira es un resultado negativo, mientras que dos líneas indican que hay un virus presente. Se usó un proceso similar para CXCL9. El mARN se aisló y amplificó, seguido de la detección de objetivo mediada por CRISPR/Cas13.
Los investigadores utilizaron el sistema de análisis para analizar más de 100 muestras de pacientes con trasplante de riñón. Los resultados revelaron que la prueba detectó con precisión el ADN del poliomavirus BK y el ADN del citomegalovirus de muestras de sangre y orina derivadas de los pacientes, así como el ARN mensajero para CXCL9 a niveles elevados en muestras de orina de pacientes que experimentaron un rechazo agudo de trasplante de riñón.
“La mayoría de las personas piensan en la edición de genes cuando piensan en CRISPR, pero esta herramienta tiene un gran potencial para otras aplicaciones, especialmente diagnósticos más baratos y rápidos”, dijo el primer autor, el Dr. Michael Kaminski, jefe del laboratorio de ingeniería de células renales y diagnóstico CRISPR en el Centro Max Delbrück de Medicina Molecular. “El desafío es llegar a concentraciones que sean clínicamente significativas. Realmente hace una gran diferencia si se busca una tonelada de objetivo sintético en su tubo de ensayo, en comparación con si se quiere llegar al nivel de molécula única en el fluido de un paciente”.
El ensayo para determinar el rechazo del trasplante de riñón se describió en la edición en línea del 13 de abril de 2020 de la revista Nature Biomedical Engineering.
Enlace relacionado:
Centro Max Delbrück de Medicina Molecular
SHERLOCK Biotechnologies
Los investigadores del Centro Max Delbrück de Medicina Molecular (Berlín, Alemania) diseñaron un sistema nuevo de análisis que detecta dos virus oportunistas comunes que infectan a los pacientes con trasplante de riñón, el citomegalovirus (CMV) y el poliomavirus BK (BKV), y para el ARNm de CXCL9, cuya expresión aumenta durante el rechazo celular agudo del trasplante de riñón.
La primera etapa del ensayo amplifica el ADN o ARN en la muestra de orina para que CRISPR lo pueda detectar. Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente y separadas entre sí) son segmentos de ADN procariota que contienen repeticiones cortas de secuencias de bases. Cada repetición es seguida por segmentos cortos de “ADN espaciador” de exposiciones previas a un virus o plásmido bacteriano. Desde 2013, se ha utilizado el sistema CRISPR/Cas9 en la investigación para la edición de genes (agregando, interrumpiendo o cambiando la secuencia de genes específicos) y la regulación de genes. A través de la administración de la enzima Cas9 y los ARN guía apropiados (sgARN) a una célula, el genoma del organismo se puede cortar en cualquier ubicación deseada. El sistema convencional CRISPR/Cas9 de Streptococcus pyogenes se compone de dos partes: la enzima Cas9, que escinde la molécula de ADN y guías de ARN específicas que guían la proteína Cas9 al gen objetivo en una cadena de ADN.
Los esfuerzos computacionales recientes para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida contra el ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación de ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, dirigido al ARN guiado por el ARN, que tiene un gran potencial para aplicaciones precisas, robustas y escalables de selección de ARN y de ARN guiado.
Después del paso de amplificación de ADN/ARN, los investigadores aplicaron el protocolo SHERLOCK de SHERLOCK Biotechnologies (Cambridge, MA, EUA). Este es un método usado para la detección de moléculas individuales de objetivos de ácido nucleico y significa desBLOQueo (unLOCKing, en inglés) del Reportero Enzimático Específico de Alta Sensibilidad. El método funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también puede ser cuantificada. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple prueba de tira de papel, en equipos de laboratorio, o para proporcionar una lectura electroquímica que se pueda leer con un teléfono móvil.
El análisis de flujo lateral usando una tira de papel se utilizó para convertir el contenido de la muestra tratada en un resultado fácilmente visualizable. Una línea que aparece en la tira es un resultado negativo, mientras que dos líneas indican que hay un virus presente. Se usó un proceso similar para CXCL9. El mARN se aisló y amplificó, seguido de la detección de objetivo mediada por CRISPR/Cas13.
Los investigadores utilizaron el sistema de análisis para analizar más de 100 muestras de pacientes con trasplante de riñón. Los resultados revelaron que la prueba detectó con precisión el ADN del poliomavirus BK y el ADN del citomegalovirus de muestras de sangre y orina derivadas de los pacientes, así como el ARN mensajero para CXCL9 a niveles elevados en muestras de orina de pacientes que experimentaron un rechazo agudo de trasplante de riñón.
“La mayoría de las personas piensan en la edición de genes cuando piensan en CRISPR, pero esta herramienta tiene un gran potencial para otras aplicaciones, especialmente diagnósticos más baratos y rápidos”, dijo el primer autor, el Dr. Michael Kaminski, jefe del laboratorio de ingeniería de células renales y diagnóstico CRISPR en el Centro Max Delbrück de Medicina Molecular. “El desafío es llegar a concentraciones que sean clínicamente significativas. Realmente hace una gran diferencia si se busca una tonelada de objetivo sintético en su tubo de ensayo, en comparación con si se quiere llegar al nivel de molécula única en el fluido de un paciente”.
El ensayo para determinar el rechazo del trasplante de riñón se describió en la edición en línea del 13 de abril de 2020 de la revista Nature Biomedical Engineering.
Enlace relacionado:
Centro Max Delbrück de Medicina Molecular
SHERLOCK Biotechnologies
Últimas Diagnóstico Molecular noticias
- Innovadora prueba de diagnóstico molecular señala con precisión principal causa genética de EPOC
- Prueba diagnóstica de sangre detecta espondiloartritis axial
- Nueva etiqueta molecular desarrolla pruebas de tuberculosis más sencillas y rápidas
- Descubrimiento de biomarcador abre camino para que análisis de sangre detecten y traten osteoartritis
- Prueba de biopsia líquida detecta recurrencia en pacientes con CCR antes de las imágenes
- Prueba ultrarrápida de líquido sinovial diagnostica osteoartritis y artritis reumatoide en 10 minutos
- Herramienta genética predice supervivencia de pacientes con cáncer de páncreas
- Prueba de orina diagnostica cáncer de próstata inicial
- Nueva herramienta genética analiza sangre del cordón umbilical para predecir enfermedades
- Biomarcador del líquido cefalorraquídeo para enfermedad de Parkinson ofrece diagnóstico temprano y preciso
- Revolucionaria prueba detecta 30 tipos diferentes de cáncer con 98 % de precisión
- Simple análisis de sangre predice mejor riesgo de enfermedad cardíaca
- Nuevo análisis de sangre detecta 12 cánceres antes de que aparezcan síntomas
- Análisis de sangre podría predecir recaída de enfermedad vascular autoinmune
- Prueba de sangre única detecta enfermedades relacionadas con traumatismos
- Gen clave identificado en enfermedad cardíaca común revela potencial diagnóstico que salva vidas
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
Ensayo automatizado y descentralizado de NGS deADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de ... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásMicrobiología
ver canal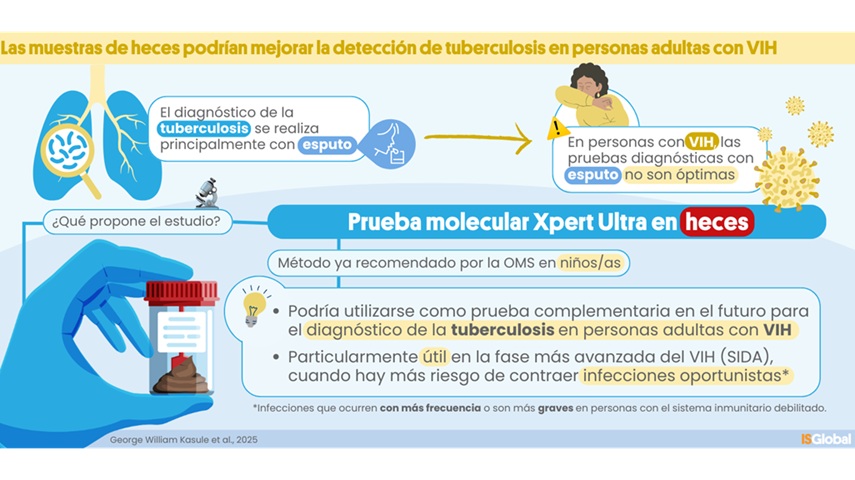
Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
La tuberculosis (TB), causada por la bacteria Mycobacterium tuberculosis, provocó 1,25 millones de muertes en 2023, de las cuales el 13 % se produjeron en personas con VIH. El principal método... Más
Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
La meningitis bacteriana es una afección potencialmente mortal: uno de cada seis pacientes fallece y la mitad de los supervivientes experimentan síntomas persistentes. Por lo tanto, un d... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más