Novedosa tecnología permite detección del Zika con alta resolución
|
Por el equipo editorial de LabMedica en español Actualizado el 07 Jun 2016 |
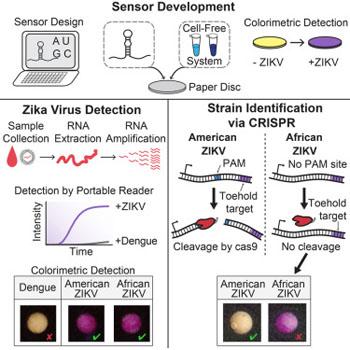
Imagen: Un resumen gráfico de un estudio reciente, donde se presenta un sistema para diagnóstico rápido en papel, con alta resolución y de bajo costo, con el fin de detectar cepas del virus Zika (Imagen cortesía de Pardee y col/Cell).

Imagen: Un cartucho negro que contiene el innovador diagnóstico, basado en papel, para detectar el virus Zika: las zonas que se han vuelto de color púrpura indican muestras infectadas con el virus Zika, mientras que las zonas amarillas indican muestras exentas de Zika (Fotografía cortesía del Instituto Wyss de la Universidad de Harvard).

Imagen: Un cartucho negro que contiene el innovador diagnóstico, basado en papel, para detectar el virus Zika: las zonas que se han vuelto de color púrpura indican muestras infectadas con el virus Zika, mientras que las zonas amarillas indican muestras exentas de Zika (Fotografía cortesía del Instituto Wyss de la Universidad de Harvard).
Partiendo de su desarrollo inicial de novedosos diagnósticos programables, unos investigadores de un equipo internacional y multiinstitucional reunió esfuerzos recientemente para crear rápidamente los prototipos de su prueba rápida y de bajo costo, con el objetivo de que se pudiera utilizar pronto para detectar el virus Zika e identificar sus cepas, en diversas muestras (sangre, orina y saliva).
El equipo, dirigido por el biólogo sintético, el Prof. James Collins, del Instituto Wyss de Ingeniería Inspirada en la Biología, de la Universidad de Harvard (Boston, MA, EUA) y del Instituto Tecnológico de Massachusetts (EUA), ha desarrollado un sistema, basado en papel, para la detección de cepas específicas, como un método de bajo costo para ayudar a frenar la propagación del brote del Zika y potencialmente de otras enfermedades pandémicas en el futuro.
“La creciente crisis sanitaria mundial producida por el virus Zika nos impulsó a aprovechar las tecnologías novedosas que hemos desarrollado en nuestro laboratorio y a utilizarlas para crear un flujo de trabajo que podría diagnosticar a los pacientes con Zika, en el campo, en un lapso de 2 a 3 horas”, dijo el Prof. Collins. “Esperamos que una herramienta como esta pueda ayudar a reducir el impacto de este brote hasta cuando se pueda desarrollar una vacuna”, dijo el primer coautor, el Dr. Keith Pardee, de la Universidad de Toronto (Canadá).
En 2014, dicho equipo desarrolló un método sin precedentes para incluir redes genéticas sintéticas (las cuales podrían ser utilizadas como sensores y diagnósticos programables), en pequeños discos de papel. Motivados por el brote, entonces en curso, del Ébola en África, hicieron una demostración como prueba del concepto, de un diagnóstico mediante un cambio de color, que permitía detectar el Ébola gracias a la incorporación al papel de un novedoso tipo de sensor biomolecular sintético diseñado para la detección de secuencias específicas de ARN, con las cuales se podrían marcar no sólo las firmas genéticas del Ébola, sino también los ARN de otros virus (por ejemplo el Zika, el SARS, el sarampión, la gripe, la hepatitis C o la fiebre del Nilo Occidental).
Un reto importante fue la muy baja concentración de virus que normalmente se encuentran en la sangre, la orina y la saliva. Ahora, utilizando muestras de sangre de monos infectados con el virus Zika, así como virus recuperados de células infectadas en el laboratorio, este equipo ha validado una técnica, de próxima generación, que supera dicho problema.
Teniendo en mente el uso en el campo, el equipo diseñó un sencillo flujo de trabajo modular, que consiste en 3 pasos: amplificación, detección del Zika e identificación de la cepa mediante CRISPR. Una vez que se ha amplificado el ARN de la muestra, se dispensa sobre discos de papel liofilizados que contienen los componentes biomoleculares. La gota de ARN amplificado activa los componentes liofilizados de modo tal que los discos cambiarán de color para indicar un resultado positivo para el virus Zika. Si bien el resultado se puede leer a simple vista, de forma similar a una prueba para embarazo, doméstica, también se puede utilizar un lector electrónico especialmente diseñado, para obtener resultados más rápidos y se podría, en el futuro, cuantificar la carga viral de la muestra.
Si se detecta el Zika, la tercera etapa consiste en mezclar una muestra con un cóctel de CRISPR liofilizadas (por ejemplo CRISPR-Cas9) y luego utilizar esta mezcla para humedecer otro juego de discos de papel que cambian de color. Dependiendo del tipo de cepa del Zika presente en la muestra, estos discos van a presentar otra serie de cambios de color visibles. Aprovechando las ventajas de las CRISPR para el reconocimiento de secuencias, este tercer paso permite discriminar entre cepas cuyos perfiles genéticos difieren en tan sólo un nucleótido, con lo cual se obtiene una resolución de una sola base.
Aunque los biólogos y los ingenieros genéticos suelen poner a trabajar las CRISPR dentro de células vivas, el equipo descubrió que funcionan igual de bien – e incluso mejor en algunos casos – después de haber sido liofilizadas.
“Hemos ensayado nuestros sistemas para diagnóstico frente a cepas estrechamente relacionadas del virus del dengue y se encontró que, dentro de los dos primeros pasos, nuestro sistema puede diferenciar fácilmente el Zika del dengue”, dijo el coautor principal, Prof. Alexander Green, PhD, de la Universidad del Estado de Arizona (EUA), “La adición de la tercera etapa a base de las CRISPR – utilizando por primera vez la Cas9 en una plataforma basada en papel – sólo mejora la exactitud de la detección. Mientras que preparamos esta tecnología para su introducción, tenemos la intención de validar nuestro sistema frente a docenas o incluso cientos de muestras clínicas”.
La capacidad para alcanzar un diagnóstico específico de la cepa en el campo podría resultar valiosa para las organizaciones nacionales y mundiales de salud, si pretenden hacer el seguimiento a la propagación de un brote viral en tiempo real, así como para preparar las estrategias de contención y los planes de tratamiento.
Esta plataforma para el diagnóstico es rentable y podría ser adaptada para identificar una gama de patógenos. Lo que es más, este método es robusto y podría ser utilizado para responder con rapidez y desarrollar nuevos métodos de diagnóstico: “Prevemos que, en respuesta a la aparición de una epidemia, un sistema para diagnóstico, hecho a medida, podría estar listo para su uso en el plazo de una semana”, dijo el profesor Collins. “Actualmente estamos buscando diversas oportunidades para asegurar la financiación privada y pública con el fin de comercializar este sistema para diagnóstico y ponerlo a disposición de los responsables de la atención sanitaria en todo el mundo”.
“La capacidad de condensar la maquinaria genética de las células vivas en un papel ordinario liofilizado proporciona una manera de desarrollar sensores revolucionarios y diagnósticos en una fracción del tiempo y con mayor sensibilidad y especificidad que otros análisis más convencionales”, dijo el director de la fundación del Instituto Wyss, el Prof. Donald Ingber, MD, PhD.
El estudio, realizado por Pardee, Green, Takahashi, Braff, Lambert et al, fue publicado el 6 de Mayo de 2016, en la revista Cell.
Enlaces relacionados:
Enlaces relacionados:
Últimas Microbiología noticias
- Nuevo análisis de sangre detecta hasta cinco enfermedades infecciosas en punto de atención
- Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
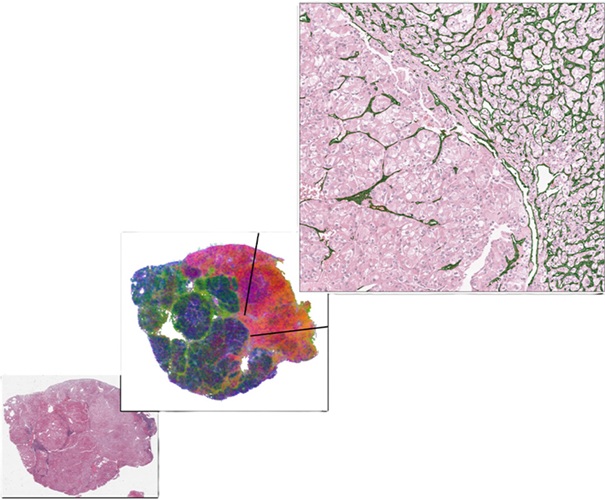
Ensayo automatizado y descentralizado de NGS deADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de ... MásDiagnóstico Molecular
ver canal
Prueba de ADN microbiano libre de células identifica patógenos de neumonía y otras infecciones pulmonares
El lavado broncoalveolar (BAL) es un procedimiento común para diagnosticar infecciones pulmonares, especialmente en pacientes inmunodeprimidos. Sin embargo, las pruebas estándar a menudo... Más
Método sin biopsia transforma diagnóstico de enfermedad celíaca en adultos
En Estados Unidos, el diagnóstico de la enfermedad celíaca en adultos suele basarse en una combinación de pruebas serológicas y una biopsia confirmatoria del intestino delgado... Más
Innovadora prueba de diagnóstico molecular señala con precisión principal causa genética de EPOC
La enfermedad pulmonar obstructiva crónica (EPOC) y la deficiencia de alfa-1 antitripsina (DAAT) son afecciones que pueden causar dificultades respiratorias, pero difieren en su origen y herencia.... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más