Encuentran brechas en mapeo del genoma humano con última tecnología
|
Por el equipo editorial de LabMedica en español Actualizado el 22 Dec 2014 |

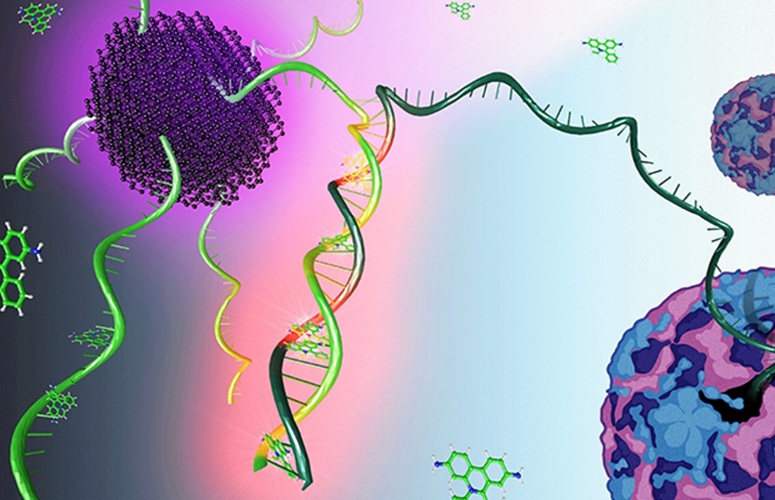
Imagen: El sistema PacBio RSII para la monitorización y el análisis de una sola molécula, mediante reacciones de secuenciación en tiempo real (SMRT) (Fotografía cortesía de Pacific Biosciences)
Se han descubierto miles de variantes genéticas, que nunca se habían visto, en el genoma humano, usando una tecnología nueva de secuenciación del genoma y estos descubrimientos cierran muchas brechas en el mapeo del genoma humano que han resistido la secuenciación durante mucho tiempo.
El genoma humano es sin duda el conjunto de referencia de los mamíferos más completo, sin embargo, permanecen más de 160 lagunas eucromáticas y los aspectos de su variación estructural siguen siendo poco conocidos diez años después de su finalización. La eucromatina es un material cromosómico que es genéticamente activo y se colorea ligeramente con los colorantes básicos.
Un equipo de científicos dirigido por aquellos en la Facultad de Medicina de la Universidad de Washington (Seattle, WA, EUA) identificó la secuencia y variaciones genéticas faltantes, mediante la secuenciación y el análisis de un genoma haploide humano derivado de la quimiotaxina 1 de un leucocito (CHM1) utilizando la secuenciación del ADN en tiempo real de una sola molécula.
La técnica utilizada se llama secuenciación del ADN en tiempo real de una sola molécula, (SMRT), que ahora podrá hacer posible que los investigadores identifiquen posibles mutaciones genéticas detrás de muchas enfermedades cuyas causas genéticas han eludido a los científicos durante largo tiempo. Los datos de secuenciación SMRT de todo el genoma (WGS) con cobertura de secuencia de 41 veces fueron generados mediante un instrumento PacBio RSII (Pacific Biosciences en Menlo Park, California, EUA, www.pacificbiosciences.com) a partir de bibliotecas genómicas generados utilizando una mola hidatiforme completa de ADN (CHM1tert).
Este método identificó con éxito millones de pequeñas variaciones en el genoma humano. Estas variaciones se deben a la sustitución de una sola base de nucleótidos, llamados polimorfismos de un solo nucleótido o SNP. El enfoque estándar también hizo posible poder identificar variaciones muy grandes, típicamente en relación con segmentos de ADN que tienen 5.000 bases de longitud o más. Pero, por razones técnicas, los científicos no habían sido capaces previamente de detectar con fiabilidad variaciones cuyas longitudes están entre, las que van desde alrededor de 50 a 5.000 bases de longitud. La tecnología SMRT utilizada en el estudio ha permitido secuenciar y leer segmentos de ADN de más de 5.000 bases, mucho más largas que con la tecnología de secuenciación de genes estándar. El equipo fue capaz de identificar y secuenciar 26.079 segmentos que eran diferentes de un genoma de referencia humano estándar utilizado en los estudios del genoma y la mayoría de estas variantes, alrededor de 22.000, nunca habían sido reportadas antes.
Evan E. Eichler, profesor de ciencias genómicas y autor principal del estudio, dijo: “En cinco años podría haber una tecnología de lecturas largas de secuencias que les permitirá a los laboratorios clínicos secuenciar los cromosomas de un paciente de punta a punta y decir: ‘Sí; tiene entre tres y cuatro millones de SNPs e inserciones y supresiones, pero también hay unas 30.000-40.000 variantes estructurales. De ellas, algunas variantes estructurales y algunos SNPs son la razón por la que eres susceptible a esta enfermedad”. Conocer toda la variación va a ser un cambio de juego”. El estudio fue publicado el 10 de noviembre de 2014, en la revista Nature.
Enlace relacionado:
University of Washington School of Medicine
El genoma humano es sin duda el conjunto de referencia de los mamíferos más completo, sin embargo, permanecen más de 160 lagunas eucromáticas y los aspectos de su variación estructural siguen siendo poco conocidos diez años después de su finalización. La eucromatina es un material cromosómico que es genéticamente activo y se colorea ligeramente con los colorantes básicos.
Un equipo de científicos dirigido por aquellos en la Facultad de Medicina de la Universidad de Washington (Seattle, WA, EUA) identificó la secuencia y variaciones genéticas faltantes, mediante la secuenciación y el análisis de un genoma haploide humano derivado de la quimiotaxina 1 de un leucocito (CHM1) utilizando la secuenciación del ADN en tiempo real de una sola molécula.
La técnica utilizada se llama secuenciación del ADN en tiempo real de una sola molécula, (SMRT), que ahora podrá hacer posible que los investigadores identifiquen posibles mutaciones genéticas detrás de muchas enfermedades cuyas causas genéticas han eludido a los científicos durante largo tiempo. Los datos de secuenciación SMRT de todo el genoma (WGS) con cobertura de secuencia de 41 veces fueron generados mediante un instrumento PacBio RSII (Pacific Biosciences en Menlo Park, California, EUA, www.pacificbiosciences.com) a partir de bibliotecas genómicas generados utilizando una mola hidatiforme completa de ADN (CHM1tert).
Este método identificó con éxito millones de pequeñas variaciones en el genoma humano. Estas variaciones se deben a la sustitución de una sola base de nucleótidos, llamados polimorfismos de un solo nucleótido o SNP. El enfoque estándar también hizo posible poder identificar variaciones muy grandes, típicamente en relación con segmentos de ADN que tienen 5.000 bases de longitud o más. Pero, por razones técnicas, los científicos no habían sido capaces previamente de detectar con fiabilidad variaciones cuyas longitudes están entre, las que van desde alrededor de 50 a 5.000 bases de longitud. La tecnología SMRT utilizada en el estudio ha permitido secuenciar y leer segmentos de ADN de más de 5.000 bases, mucho más largas que con la tecnología de secuenciación de genes estándar. El equipo fue capaz de identificar y secuenciar 26.079 segmentos que eran diferentes de un genoma de referencia humano estándar utilizado en los estudios del genoma y la mayoría de estas variantes, alrededor de 22.000, nunca habían sido reportadas antes.
Evan E. Eichler, profesor de ciencias genómicas y autor principal del estudio, dijo: “En cinco años podría haber una tecnología de lecturas largas de secuencias que les permitirá a los laboratorios clínicos secuenciar los cromosomas de un paciente de punta a punta y decir: ‘Sí; tiene entre tres y cuatro millones de SNPs e inserciones y supresiones, pero también hay unas 30.000-40.000 variantes estructurales. De ellas, algunas variantes estructurales y algunos SNPs son la razón por la que eres susceptible a esta enfermedad”. Conocer toda la variación va a ser un cambio de juego”. El estudio fue publicado el 10 de noviembre de 2014, en la revista Nature.
Enlace relacionado:
University of Washington School of Medicine
Últimas Diagnóstico Molecular noticias
- Herramienta genética predice supervivencia de pacientes con cáncer de páncreas
- Prueba de orina diagnostica cáncer de próstata inicial
- Nueva herramienta genética analiza sangre del cordón umbilical para predecir enfermedades
- Biomarcador del líquido cefalorraquídeo para enfermedad de Parkinson ofrece diagnóstico temprano y preciso
- Revolucionaria prueba detecta 30 tipos diferentes de cáncer con 98 % de precisión
- Simple análisis de sangre predice mejor riesgo de enfermedad cardíaca
- Nuevo análisis de sangre detecta 12 cánceres antes de que aparezcan síntomas
- Análisis de sangre podría predecir recaída de enfermedad vascular autoinmune
- Prueba de sangre única detecta enfermedades relacionadas con traumatismos
- Gen clave identificado en enfermedad cardíaca común revela potencial diagnóstico que salva vidas
- Prueba económica de ADN libre celular predice con precisión parto prematuro
- Prueba de ARN sanguíneo detecta cánceres y resistencia al tratamiento
- IL-6 supera a pruebas tradicionales para detección temprana de sepsis
- Simple análisis de sangre mejora predicción del riesgo de ataque cardíaco y ACV
- Prueba de biomarcadores sanguíneos podría detectar predisposición genética al Alzheimer
- Se descubre nuevo autoanticuerpo contra DAGLA en cerebelitis
Canales
Química Clínica
ver canal
Monitorización con espectrometría de masas predice e identifica recaída temprana del mieloma
El mieloma, un tipo de cáncer que afecta la médula ósea, es actualmente incurable, aunque muchos pacientes pueden vivir más de 10 años tras el diagnóstico.... Más
Herramienta química a nanoescala 'brillantemente luminosa' mejora detección de enfermedades
Miles de moléculas brillantes disponibles comercialmente, conocidas como fluoróforos, se utilizan comúnmente en imágenes médicas, detección de enfermedades, marcado... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásMicrobiología
ver canal
Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
La meningitis bacteriana es una afección potencialmente mortal: uno de cada seis pacientes fallece y la mitad de los supervivientes experimentan síntomas persistentes. Por lo tanto, un d... Más
Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
La tuberculosis (TB) sigue siendo la enfermedad infecciosa más mortal a nivel mundial, afectando a aproximadamente 10 millones de personas al año. En 2021, alrededor de 4,2 millones de casos... MásPatología
ver canal
Modelo de IA predice respuesta a terapia contra cáncer de riñón
Cada año, cerca de 435.000 personas son diagnosticadas con carcinoma renal de células claras (CRcc), lo que lo convierte en el subtipo más prevalente de cáncer de riñón.... Más
Kits de ensayo de enzima DUB sensibles y específicos requieren configuración mínima sin preparación del sustrato
La ubiquitinación y la desubiquitinación son dos procesos fisiológicos importantes en el sistema ubiquitina-proteasoma, responsable de la degradación de proteínas en... MásTecnología
ver canal
Algoritmo de firma ligera permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... Más
Tecnología de microchip desechable podría detectar selectivamente VIH en muestras de sangre completa
A finales de 2023, aproximadamente 40 millones de personas en todo el mundo vivían con VIH, y alrededor de 630.000 personas murieron por enfermedades relacionadas con el sida ese mismo año.... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más









 Assay.jpg)