Método más rápido identifica cepas de Salmonella
|
Por el equipo editorial de LabMedica en español Actualizado el 22 Jul 2013 |
Imagen: Microfotografía electrónica de barrido (SEM) de la bacteria Salmonella (Fotografía cortesía de los Centros para el Control de Enfermedades de los EE.UU).
Un método de subtipificación basado en secuencia, puede reducir en más de la mitad el tiempo necesario para que los funcionarios de salud identifiquen las cepas de Salmonella.
Se requieren métodos rápidos, eficientes y exactos para la identificación de cepas específicas de la serovar Newport, durante el brote, permitiendo a los investigadores epidemiológicos identificar las cepas exactas de Salmonella que hacen que las personas se enfermen, para encontrar y eliminar más rápidamente el origen de la enfermedad.
Los microbiólogos de la Universidad Estatal de Pennsylvania (White Oak, PA, EUA) desarrollaron un nuevo método para identificar las cepas de Salmonella enterica subespecie enterica serovar Newport. El método se centra en dos genes de virulencia y dos regiones nuevas de ADN de la Salmonella llamadas repeticiones palindrómicas, cortas, interespaciadas, agrupadas regularmente, o CRISPRs. Los científicos idearon un método de tipificación de secuencia multi-virulencia-locus o MVLST, que puede detectar las diferencias específicas de las cepas en el ADN de estas cuatro ubicaciones. El método se denomina CRISPR-MVLST.
Se obtuvieron un total de 84 aislamientos clínicos de la serovar Newport y se seleccionaron sistemáticamente a lo largo de cinco años. Los aislamientos que se recibieron más cercanos al día 1 y el día 15 de cada mes, desde 2007 hasta 2011 fueron seleccionados para representar una colección imparcial de los aislados clínicos humanos. Los investigadores usaron amplificación de reacción en cadena de la polimerasa (PCR), secuenciación del ADN y análisis de la secuencia y el tipo de asignación de secuencias, para estudiar los aislamientos. Hacia el final del proyecto, aplicaron su análisis a un brote de Salmonella, asociado con tomates que sucedió en Pennsylvania, en el verano de 2012, en el que 37 personas se enfermaron.
Los autores llegaron a la conclusión de que las ventajas de CRISPR-MVLST son la automatización potencial del procedimiento, la capacidad para llevar a cabo este análisis en una forma de alto rendimiento, y que los datos de secuencia son inherentemente más manejable para aplicaciones posteriores tales como análisis filogenéticos y evolutivos en comparación con las electroforesis en gel de campo pulsado (PFGE).
Nikki Shariat, PhD, autora principal del estudio, dijo: “En este momento, los laboratorios de salud pública utilizan una técnica llamada PFGE, para hacer la sub-tipificación de las cepas de Salmonella, y la identificación se tarda, normalmente, entre uno y tres días para una cepa específica. La técnica que ideamos, a menudo, toma un solo día. El cincuenta por ciento de las bacterias tiene regiones CRISPR y el uso de éstas para la identificación se ha hecho con un buen número de bacterias, tales como Mycobacterium tuberculosis, así como con algunos que causan enfermedades transmitidas por alimentos, tales como Campylobacter y Escherichia coli”. El estudio fue publicado el 15 de mayo de 2013, en la revista Journal of Clinical Microbiology.
Enlace relacionado:
The Pennsylvania State University
Se requieren métodos rápidos, eficientes y exactos para la identificación de cepas específicas de la serovar Newport, durante el brote, permitiendo a los investigadores epidemiológicos identificar las cepas exactas de Salmonella que hacen que las personas se enfermen, para encontrar y eliminar más rápidamente el origen de la enfermedad.
Los microbiólogos de la Universidad Estatal de Pennsylvania (White Oak, PA, EUA) desarrollaron un nuevo método para identificar las cepas de Salmonella enterica subespecie enterica serovar Newport. El método se centra en dos genes de virulencia y dos regiones nuevas de ADN de la Salmonella llamadas repeticiones palindrómicas, cortas, interespaciadas, agrupadas regularmente, o CRISPRs. Los científicos idearon un método de tipificación de secuencia multi-virulencia-locus o MVLST, que puede detectar las diferencias específicas de las cepas en el ADN de estas cuatro ubicaciones. El método se denomina CRISPR-MVLST.
Se obtuvieron un total de 84 aislamientos clínicos de la serovar Newport y se seleccionaron sistemáticamente a lo largo de cinco años. Los aislamientos que se recibieron más cercanos al día 1 y el día 15 de cada mes, desde 2007 hasta 2011 fueron seleccionados para representar una colección imparcial de los aislados clínicos humanos. Los investigadores usaron amplificación de reacción en cadena de la polimerasa (PCR), secuenciación del ADN y análisis de la secuencia y el tipo de asignación de secuencias, para estudiar los aislamientos. Hacia el final del proyecto, aplicaron su análisis a un brote de Salmonella, asociado con tomates que sucedió en Pennsylvania, en el verano de 2012, en el que 37 personas se enfermaron.
Los autores llegaron a la conclusión de que las ventajas de CRISPR-MVLST son la automatización potencial del procedimiento, la capacidad para llevar a cabo este análisis en una forma de alto rendimiento, y que los datos de secuencia son inherentemente más manejable para aplicaciones posteriores tales como análisis filogenéticos y evolutivos en comparación con las electroforesis en gel de campo pulsado (PFGE).
Nikki Shariat, PhD, autora principal del estudio, dijo: “En este momento, los laboratorios de salud pública utilizan una técnica llamada PFGE, para hacer la sub-tipificación de las cepas de Salmonella, y la identificación se tarda, normalmente, entre uno y tres días para una cepa específica. La técnica que ideamos, a menudo, toma un solo día. El cincuenta por ciento de las bacterias tiene regiones CRISPR y el uso de éstas para la identificación se ha hecho con un buen número de bacterias, tales como Mycobacterium tuberculosis, así como con algunos que causan enfermedades transmitidas por alimentos, tales como Campylobacter y Escherichia coli”. El estudio fue publicado el 15 de mayo de 2013, en la revista Journal of Clinical Microbiology.
Enlace relacionado:
The Pennsylvania State University
Últimas Microbiología noticias
- Nuevo análisis de sangre detecta hasta cinco enfermedades infecciosas en punto de atención
- Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
Ensayo automatizado y descentralizado de NGS de ADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de secuenciación flexible... MásDiagnóstico Molecular
ver canal
Prueba de hisopado oral POC para aumenta posibilidades de embarazo en la FIV
Aproximadamente el 15 % de las parejas en edad reproductiva experimentan infertilidad involuntaria. Una razón importante para esto es la creciente tendencia a retrasar la planificación familiar,... Más
Prueba de ADN microbiano libre de células identifica patógenos de neumonía y otras infecciones pulmonares
El lavado broncoalveolar (BAL) es un procedimiento común para diagnosticar infecciones pulmonares, especialmente en pacientes inmunodeprimidos. Sin embargo, las pruebas estándar a menudo... Más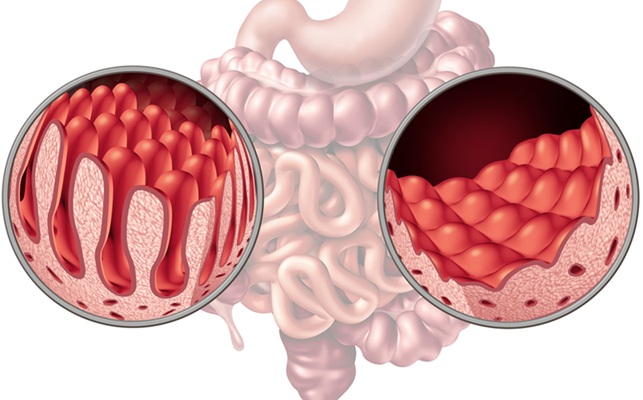
Método sin biopsia transforma diagnóstico de enfermedad celíaca en adultos
En Estados Unidos, el diagnóstico de la enfermedad celíaca en adultos suele basarse en una combinación de pruebas serológicas y una biopsia confirmatoria del intestino delgado... Más
Innovadora prueba de diagnóstico molecular señala con precisión principal causa genética de EPOC
La enfermedad pulmonar obstructiva crónica (EPOC) y la deficiencia de alfa-1 antitripsina (DAAT) son afecciones que pueden causar dificultades respiratorias, pero difieren en su origen y herencia.... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más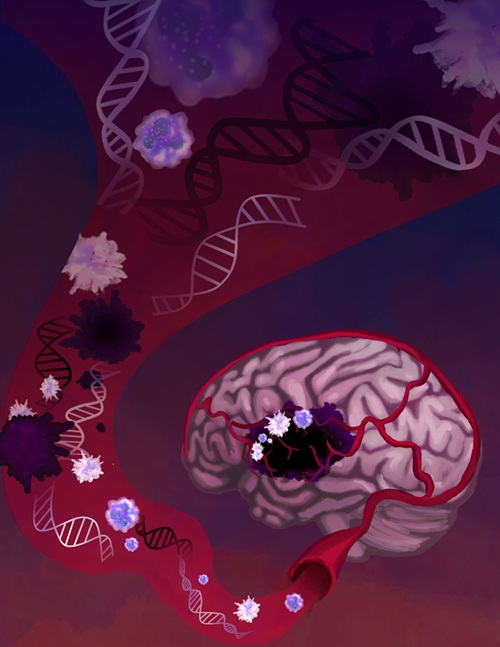
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
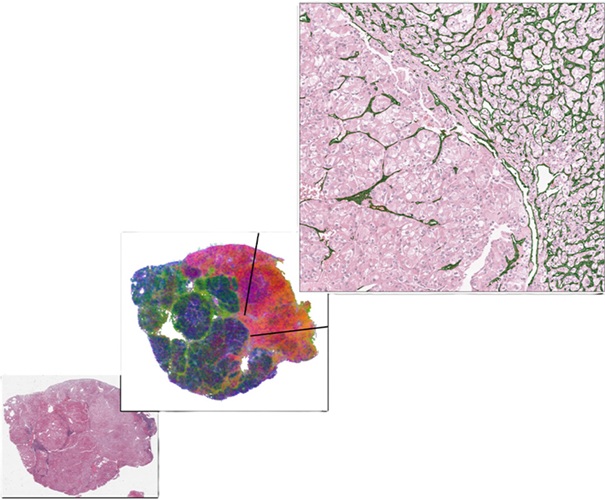
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Qiagen adquiere Genoox, empresa de software de análisis NGS
QIAGEN (Venlo, Países Bajos) ha firmado un acuerdo definitivo para adquirir Genoox (Tel Aviv, Israel), un proveedor de software impulsado por inteligencia artificial (IA) que permite a los laboratorios... Más