Secuenciación podría complementar otros métodos de cribado de recién nacidos
|
Por el equipo editorial de LabMedica en español Actualizado el 16 Sep 2020 |
Imagen: La secuenciación del exoma del recién nacido podría complementar otros métodos de cribado de los recién nacidos (Fotografía cortesía de Sushytska).
El examen de cribado de los recién nacidos (NBS) se estableció como un programa de salud pública en la década de 1960 y es crucial para facilitar la detección de ciertas afecciones médicas en las que la intervención temprana puede prevenir problemas de salud graves y potencialmente mortales.
La secuenciación genómica puede expandir potencialmente la detección de trastornos hereditarios raros, pero muchas preguntas rodean su posible uso para este propósito. Es posible que los métodos basados en la secuenciación no reemplacen el cribado del recién nacido con base bioquímica o fenotípica, pero pueden complementar esos enfoques.
Los genetistas de la Universidad de Carolina del Norte en Chapel Hill (Chapel Hill, NC, EUA) y sus colegas, examinaron el uso de la secuenciación del exoma (SE) para el NBS en el proyecto de Cribado Universal para la Secuenciación del Exoma del Recién Nacido de Carolina del Norte (NC NEXUS), que compara el rendimiento de la SE utilizada en un contexto de detección versus diagnóstico. Reclutaron, en un estudio, a 17 niños con errores innatos del metabolismo, 28 niños con pérdida auditiva y 61 bebés sanos. A cada niño se le realizó un cribado neonatal basado en secuenciación de próxima generación utilizando un panel de 466 genes. De las 46 variantes detectadas en esta cohorte mediante la secuenciación del exoma, 43 se confirmaron ortogonalmente en un laboratorio certificado por CLIA.
El equipo informó que, dentro de la cohorte metabólica, el cribado basado en secuenciación descubrió un resultado anormal en 15 de los 17 participantes. Por ejemplo, siete niños que tenían fenilcetonuria tenían variantes patológicas en HAP, siete niños con deficiencia de acil-coA deshidrogenasa de cadena media, tenían variantes patogénicas en ACADM y un niño con deficiencia primaria de carnitina era homocigoto para una variante patógena sin sentido de SLC22A5.
Sin embargo, en la cohorte de pérdida auditiva, el cribado neonatal basado en secuenciación de próxima generación arrojó resultados positivos solo en cinco de los 28 participantes. Entre ellos había dos niños que eran heterocigotos compuestos para variantes en un gen del síndrome de Usher y un niño que tenía una deleción de cambio de marco de un par de bases en el gen GJB2, que está relacionado con la sordera no sindrómica DFNB1.
En la cohorte completa, el cribado basado en la secuenciación identificó a cuatro niños con resultados positivos, una variante relacionada con la hipercolesterolemia familiar, una variante sin sentido asociada con una deficiencia leve de ornitina transcarbamilasa (OTC), una variante del sitio de empalme en DSC2 vinculada a la displasia ventricular derecha arritmogénica, autosómica, dominante y dos variantes de F11 relacionadas con la deficiencia autosómica recesiva del factor XI.
Los autores concluyeron que, dado que el cribado basado en secuenciación omitió algunos resultados metabólicos y de pérdida auditiva, probablemente no reemplazaría los métodos de cribado actuales, pero que se podía usar en conjunto con ellos. Estos hallazgos sugieren que la secuenciación podría ser útil como complemento de los métodos tradicionales de NBS y que con una mejor detección de variantes y bases de datos interpretativas más extensas, el valor predictivo positivo del cribado genómico puede mejorar. El estudio fue publicado el 26 de agosto de 2020 en la revista American Journal of Human Genetics.
Enlace relacionado:
Universidad de Carolina del Norte en Chapel Hill
La secuenciación genómica puede expandir potencialmente la detección de trastornos hereditarios raros, pero muchas preguntas rodean su posible uso para este propósito. Es posible que los métodos basados en la secuenciación no reemplacen el cribado del recién nacido con base bioquímica o fenotípica, pero pueden complementar esos enfoques.
Los genetistas de la Universidad de Carolina del Norte en Chapel Hill (Chapel Hill, NC, EUA) y sus colegas, examinaron el uso de la secuenciación del exoma (SE) para el NBS en el proyecto de Cribado Universal para la Secuenciación del Exoma del Recién Nacido de Carolina del Norte (NC NEXUS), que compara el rendimiento de la SE utilizada en un contexto de detección versus diagnóstico. Reclutaron, en un estudio, a 17 niños con errores innatos del metabolismo, 28 niños con pérdida auditiva y 61 bebés sanos. A cada niño se le realizó un cribado neonatal basado en secuenciación de próxima generación utilizando un panel de 466 genes. De las 46 variantes detectadas en esta cohorte mediante la secuenciación del exoma, 43 se confirmaron ortogonalmente en un laboratorio certificado por CLIA.
El equipo informó que, dentro de la cohorte metabólica, el cribado basado en secuenciación descubrió un resultado anormal en 15 de los 17 participantes. Por ejemplo, siete niños que tenían fenilcetonuria tenían variantes patológicas en HAP, siete niños con deficiencia de acil-coA deshidrogenasa de cadena media, tenían variantes patogénicas en ACADM y un niño con deficiencia primaria de carnitina era homocigoto para una variante patógena sin sentido de SLC22A5.
Sin embargo, en la cohorte de pérdida auditiva, el cribado neonatal basado en secuenciación de próxima generación arrojó resultados positivos solo en cinco de los 28 participantes. Entre ellos había dos niños que eran heterocigotos compuestos para variantes en un gen del síndrome de Usher y un niño que tenía una deleción de cambio de marco de un par de bases en el gen GJB2, que está relacionado con la sordera no sindrómica DFNB1.
En la cohorte completa, el cribado basado en la secuenciación identificó a cuatro niños con resultados positivos, una variante relacionada con la hipercolesterolemia familiar, una variante sin sentido asociada con una deficiencia leve de ornitina transcarbamilasa (OTC), una variante del sitio de empalme en DSC2 vinculada a la displasia ventricular derecha arritmogénica, autosómica, dominante y dos variantes de F11 relacionadas con la deficiencia autosómica recesiva del factor XI.
Los autores concluyeron que, dado que el cribado basado en secuenciación omitió algunos resultados metabólicos y de pérdida auditiva, probablemente no reemplazaría los métodos de cribado actuales, pero que se podía usar en conjunto con ellos. Estos hallazgos sugieren que la secuenciación podría ser útil como complemento de los métodos tradicionales de NBS y que con una mejor detección de variantes y bases de datos interpretativas más extensas, el valor predictivo positivo del cribado genómico puede mejorar. El estudio fue publicado el 26 de agosto de 2020 en la revista American Journal of Human Genetics.
Enlace relacionado:
Universidad de Carolina del Norte en Chapel Hill
Últimas Patología noticias
- Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
- Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
- Análisis de imágenes de patología digital con IA mejora subtipificación del sarcoma pediátrico
- Modelo de IA predice respuesta a terapia contra cáncer de riñón
- Kits de ensayo de enzima DUB sensibles y específicos requieren configuración mínima sin preparación del sustrato
- Primer modelo de IA para diagnóstico de cáncer de tiroides con precisión superior al 90 %
- Enfoque diagnóstico innovador mejora significativamente la detección de tuberculosis
- Método de detección rápido, ultrasensible y sin PCR hace el análisis genético más accesible
- Prueba de saliva más precisa para identificar riesgo de cáncer de próstata
- Nanotecnología del ADN aumenta sensibilidad de tiras reactivas
- Nuevo método basado en aprendizaje automático detecta contaminación microbiana en cultivos celulares
- Nuevo método con corrección de errores detecta cáncer únicamente en muestras de sangre
- Algoritmo "detector de metales" consigue tumores vulnerables
- Nueva técnica identifica y clasifica subtipos de células de cáncer de páncreas
- Imágenes avanzadas revelan mecanismos que causan enfermedades autoinmunes
- Modelo de IA predice eficazmente resultados de pacientes con cáncer de pulmón
Canales
Química Clínica
ver canal
Análisis de sangre con IA detecta cáncer de ovario
El cáncer de ovario se ubica como la quinta causa principal de muerte por cáncer en mujeres, debido principalmente a diagnósticos en etapas tardías. Si bien más del 90... Más
Ensayo automatizado y descentralizado de NGS deADNlc identifica alteraciones en tumores sólidos avanzados
Los análisis actuales de ADN libre circulante (ADNlc) suelen estar centralizados, lo que requiere un manejo y transporte especializados de las muestras. La introducción de un sistema de ... MásHematología
ver canal
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... Más
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásMicrobiología
ver canal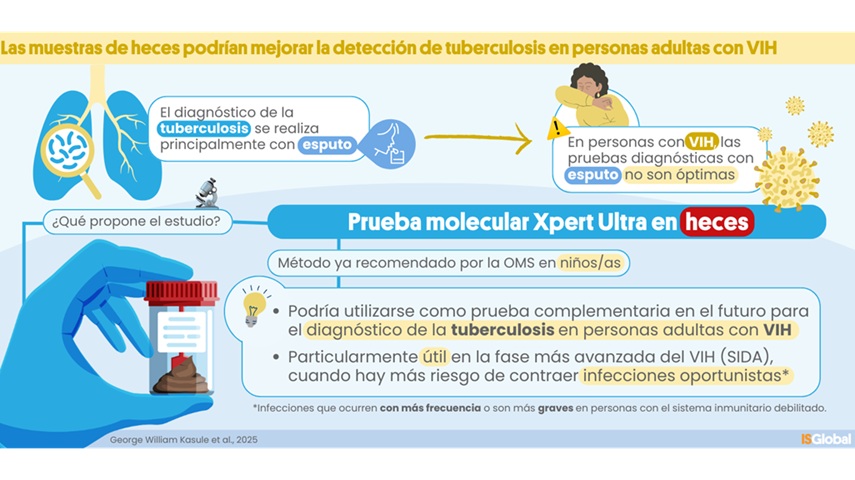
Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
La tuberculosis (TB), causada por la bacteria Mycobacterium tuberculosis, provocó 1,25 millones de muertes en 2023, de las cuales el 13 % se produjeron en personas con VIH. El principal método... Más
Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
La meningitis bacteriana es una afección potencialmente mortal: uno de cada seis pacientes fallece y la mitad de los supervivientes experimentan síntomas persistentes. Por lo tanto, un d... MásPatología
ver canal
Innovador algoritmo de triaje del dolor torácico transforma la atención cardíaca
Las enfermedades cardiovasculares son responsables de un tercio de las muertes en todo el mundo, y el dolor torácico es la segunda causa más común de visitas a urgencias.... Más
Enfoque de biopsia líquida basado en IA revolucionará detección del cáncer cerebral
Detectar cánceres cerebrales sigue siendo extremadamente difícil, ya que muchos pacientes solo reciben un diagnóstico en etapas avanzadas, tras la aparición de síntomas... MásTecnología
ver canal
Algoritmos predictivos avanzados identifican pacientes con cáncer no diagnosticado
Dos algoritmos predictivos avanzados recientemente desarrollados aprovechan el estado de salud de una persona y los resultados de análisis de sangre básicos para predecir con precisión... Más
Algoritmo de firma de luz permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más