Secuencia de todo el genoma para mejor seguimiento de brotes de TB
|
Por el equipo editorial de LabMedica en español Actualizado el 08 Apr 2013 |
Un estudio reveló que una nueva forma de pruebas genéticas de la bacteria que causa la tuberculosis (TB), suministra una información más detallada sobre la transmisión, y así permite el seguimiento de los brotes de TB con más exactitud que las pruebas estándares.
Un equipo de expertos de instituciones de salud pública, institutos de investigación y universidades en Alemania y Francia, dirigido por Stefan Niemann del Forschungszentrum Borstel (Borstel, Alemania), comparó los resultados de los dos tipos de pruebas en 86 aislados de Mycobacterium tuberculosis de un brote de tuberculosis en los estados alemanes de Hamburgo y Schleswig-Holstein (en total, se investigaron 2301 casos de tuberculosis en el período de estudio de 1997 a 2010).
Ellos encontraron que la nueva prueba, basada en la secuenciación de los genomas respectivos enteros (es decir, la secuenciación de todo el genoma, WGS) proporcionan una información más exacta sobre la agrupación temporal y la propagación del patógeno, que las pruebas estándar, que se basan en el análisis de pequeñas regiones del genoma (genotipo clásico). Es importante destacar que, mientras que las pruebas estándar no eran capaces de diferenciar las cepas implicadas, los análisis basados en WGS revelaron que sólo un clon particular comenzó a propagarse en el inicio del brote, lo que sugiere que las diferencias sutiles en el genoma podrían influir en el éxito de la transmisión de patógenos.
“Sólo las investigaciones basadas en del genoma nos han permitido rastrear la propagación de M. tuberculosis con la resolución necesaria para visualizar correctamente los patrones de transmisión”, dijo el Dr. Andreas Rötzer, primer autor del estudio.
La genotipificación de cepas de M. tuberculosis se utiliza generalmente para detectar brotes de tuberculosis y orientar el seguimiento de contactos de casos de TB. Sin embargo, la genotipificación estándar analiza sólo pequeñas partes del genoma, y no puede, por lo tanto, ser capaz de diferenciar entre cepas estrechamente relacionadas que se difunden en cadenas de transmisión diferentes. Esto fue confirmado por este estudio: la tipificación basada en WGS discriminó mejor las diferentes variantes de cepas implicadas en el brote, tenía una mejor concordancia con la información de los contactos conocidos entre los pacientes, y permitió a los investigadores seguir, con mayor precisión, la propagación de clones en el espacio y tiempo.
Basándose en los datos de secuenciación del genoma, los autores también fueron capaces de calcular que el genoma de M. tuberculosis evoluciona en su población huésped natural (individuos infectados) a una tasa de mutación más lenta que otros patógenos bacterianos (0,4 mutaciones por genoma por año). Esta medida de la tasa de mutación de la bacteria será útil para rastrear los brotes futuros y calcular cuándo, y a través de que individuo, se originaron.
Una ventaja adicional del WGS, en comparación con el genotipo normal, es que el WGS permite la identificación de mutaciones de los genes bacterianos que causan mutaciones de resistencia a antibióticos y las variaciones en los genes de virulencia. Esto es especialmente importante ya que están apareciendo, cada vez más, cepas de M. tuberculosis resistentes a los fármacos más potentes, en varias regiones del mundo y la detección rápida de la resistencia es crucial para un tratamiento exitoso.
Los costos del análisis de todo el genoma con base en la Secuenciación de Próxima Generación están disminuyendo; por lo tanto, esta prueba podría convertirse, pronto, en el método estándar para identificar los patrones de transmisión y los tipos de brotes de enfermedades infecciosas.
Además, los autores afirman: “Tenemos la visión de que la aplicación efectiva y progresiva del WGS para la Salud Pública y los diagnósticos médicos, también se verá acelerada por la distribución más amplia de máquinas de secuenciación más accesibles y flexibles y los próximos desarrollos de bioinformática para facilitar la interpretación rápida y pertinente de los datos obtenidos por el personal médico y clínico”. El estudio fue publicado en PLoS Medicine en marzo de 2013.
Enlace relacionado:
Forschungszentrum Borstel
Un equipo de expertos de instituciones de salud pública, institutos de investigación y universidades en Alemania y Francia, dirigido por Stefan Niemann del Forschungszentrum Borstel (Borstel, Alemania), comparó los resultados de los dos tipos de pruebas en 86 aislados de Mycobacterium tuberculosis de un brote de tuberculosis en los estados alemanes de Hamburgo y Schleswig-Holstein (en total, se investigaron 2301 casos de tuberculosis en el período de estudio de 1997 a 2010).
Ellos encontraron que la nueva prueba, basada en la secuenciación de los genomas respectivos enteros (es decir, la secuenciación de todo el genoma, WGS) proporcionan una información más exacta sobre la agrupación temporal y la propagación del patógeno, que las pruebas estándar, que se basan en el análisis de pequeñas regiones del genoma (genotipo clásico). Es importante destacar que, mientras que las pruebas estándar no eran capaces de diferenciar las cepas implicadas, los análisis basados en WGS revelaron que sólo un clon particular comenzó a propagarse en el inicio del brote, lo que sugiere que las diferencias sutiles en el genoma podrían influir en el éxito de la transmisión de patógenos.
“Sólo las investigaciones basadas en del genoma nos han permitido rastrear la propagación de M. tuberculosis con la resolución necesaria para visualizar correctamente los patrones de transmisión”, dijo el Dr. Andreas Rötzer, primer autor del estudio.
La genotipificación de cepas de M. tuberculosis se utiliza generalmente para detectar brotes de tuberculosis y orientar el seguimiento de contactos de casos de TB. Sin embargo, la genotipificación estándar analiza sólo pequeñas partes del genoma, y no puede, por lo tanto, ser capaz de diferenciar entre cepas estrechamente relacionadas que se difunden en cadenas de transmisión diferentes. Esto fue confirmado por este estudio: la tipificación basada en WGS discriminó mejor las diferentes variantes de cepas implicadas en el brote, tenía una mejor concordancia con la información de los contactos conocidos entre los pacientes, y permitió a los investigadores seguir, con mayor precisión, la propagación de clones en el espacio y tiempo.
Basándose en los datos de secuenciación del genoma, los autores también fueron capaces de calcular que el genoma de M. tuberculosis evoluciona en su población huésped natural (individuos infectados) a una tasa de mutación más lenta que otros patógenos bacterianos (0,4 mutaciones por genoma por año). Esta medida de la tasa de mutación de la bacteria será útil para rastrear los brotes futuros y calcular cuándo, y a través de que individuo, se originaron.
Una ventaja adicional del WGS, en comparación con el genotipo normal, es que el WGS permite la identificación de mutaciones de los genes bacterianos que causan mutaciones de resistencia a antibióticos y las variaciones en los genes de virulencia. Esto es especialmente importante ya que están apareciendo, cada vez más, cepas de M. tuberculosis resistentes a los fármacos más potentes, en varias regiones del mundo y la detección rápida de la resistencia es crucial para un tratamiento exitoso.
Los costos del análisis de todo el genoma con base en la Secuenciación de Próxima Generación están disminuyendo; por lo tanto, esta prueba podría convertirse, pronto, en el método estándar para identificar los patrones de transmisión y los tipos de brotes de enfermedades infecciosas.
Además, los autores afirman: “Tenemos la visión de que la aplicación efectiva y progresiva del WGS para la Salud Pública y los diagnósticos médicos, también se verá acelerada por la distribución más amplia de máquinas de secuenciación más accesibles y flexibles y los próximos desarrollos de bioinformática para facilitar la interpretación rápida y pertinente de los datos obtenidos por el personal médico y clínico”. El estudio fue publicado en PLoS Medicine en marzo de 2013.
Enlace relacionado:
Forschungszentrum Borstel
Últimas Diagnóstico Molecular noticias
- Prueba PGx POC permite prescripción rápida y adecuada de antiplaquetarios para pacientes con ACV
- Tecnología de secuenciación rápida de ADN mejora tratamiento de infecciones relacionadas con fracturas
- Nuevo panel de biomarcadores mejora evaluación de enfermedad renal crónica pediátrica
- Puntuación proteómica sanguínea predice longevidad y riesgo de enfermedad
- IA identifica combinaciones de genes que causan enfermedades complejas
- Pruebas genómicas guían decisiones sobre quimioterapia en adultos mayores con cáncer de mama
- Dispositivo portátil detecta mutaciones genéticas raras en una gota de sangre
- Pruebas de sangre de ADNtc mejoran diagnóstico de cáncer de pulmón
- Innovadora prueba de sangre detecta enfermedad celíaca en pacientes sin exposición al gluten
- Plataforma económica y cercana al paciente identifica rápidamente ITS
- Marcadores moleculares prometedores apoyan el diagnóstico temprano del cáncer gástrico
- La prueba MCED basada en sangre permite la detección temprana de múltiples tipos de cáncer
- Primera herramienta impulsada por IA que transforma diagnóstico y tratamiento de diabetes tipo 1
- Sistema POC proporciona resultados de PCR con alta calidad, rápidamente desde muestra de hisopo
- Prueba de ADNtc en sangre demuestra precisa para detección de cáncer colorrectal
- Pruebas MCED detectan cánceres en el torrente sanguíneo tres años antes del diagnóstico
Canales
Química Clínica
ver canal
Simple prueba cutánea podría revolucionar el diagnóstico de esofagitis eosinofílica pediátrica
La esofagitis eosinofílica (EE) es una enfermedad crónica, causada por alérgenos alimentarios, que se caracteriza por inflamación y daño del revestimiento esofágico.... Más
Herramienta de diagnóstico portátil utiliza bioluminiscencia para detectar virus POC
Los diagnósticos en el punto de atención se han convertido en herramientas cruciales en muchos hogares, permitiendo a las personas medir la glucemia, realizar pruebas de embarazo e incluso... Más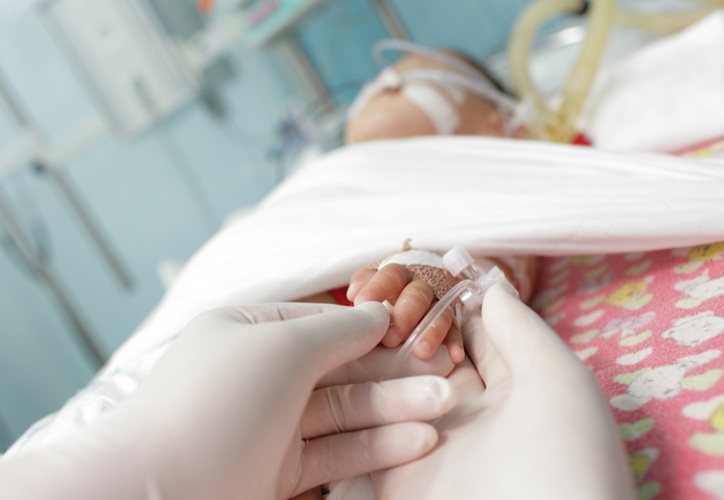
Prueba de madurez pulmonar con IA identifica recién nacidos con mayor riesgo de dificultad respiratoria
Cada año, aproximadamente 300.000 bebés nacen en Estados Unidos entre las 32 y 36 semanas de gestación, según datos nacionales de salud. Este grupo presenta un riesgo elevado de dificultad respiratoria,... MásHematología
ver canal
Prueba de cartucho desechable ofrece resultados de hemograma rápidos y precisos
El hemograma completo (HC) es una de las pruebas de laboratorio más solicitadas, crucial para diagnosticar enfermedades, monitorear terapias y realizar exámenes de salud rutinarios.... Más
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... MásInmunología
ver canal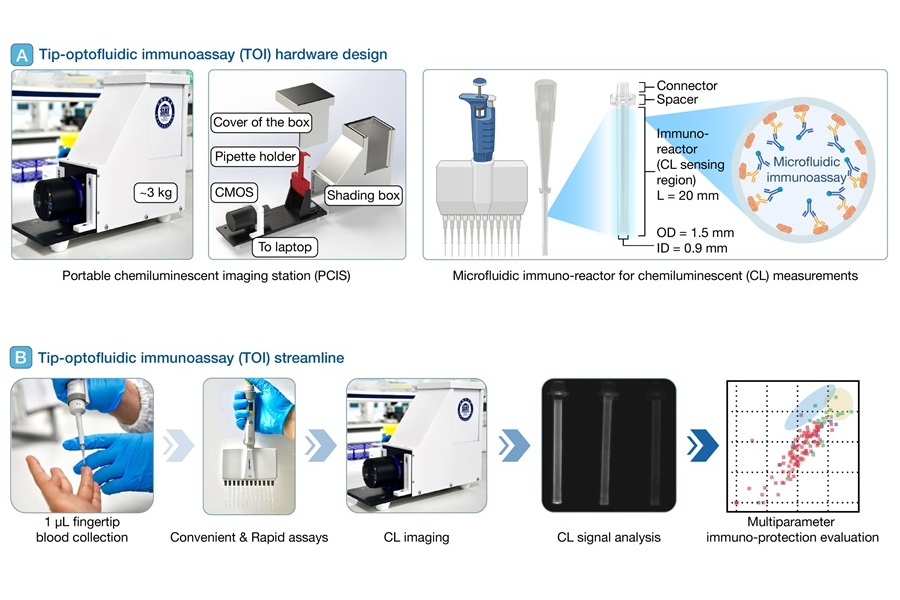
Plataforma de diagnóstico POC realiza análisis inmunológico con una gota de sangre del dedo
A medida que surgen nuevas variantes de la COVID-19 y las personas acumulan historiales complejos de vacunación e infección, existe una necesidad urgente de herramientas de diagnóstico... Más
Cambio de tratamiento guiado por biopsia líquida mejora resultados en pacientes con cáncer de mama
El tratamiento estándar para pacientes con cáncer de mama avanzado con receptor de estrógeno (RE) positivo y HER2 negativo, un subtipo impulsado por los receptores de estrógeno... Más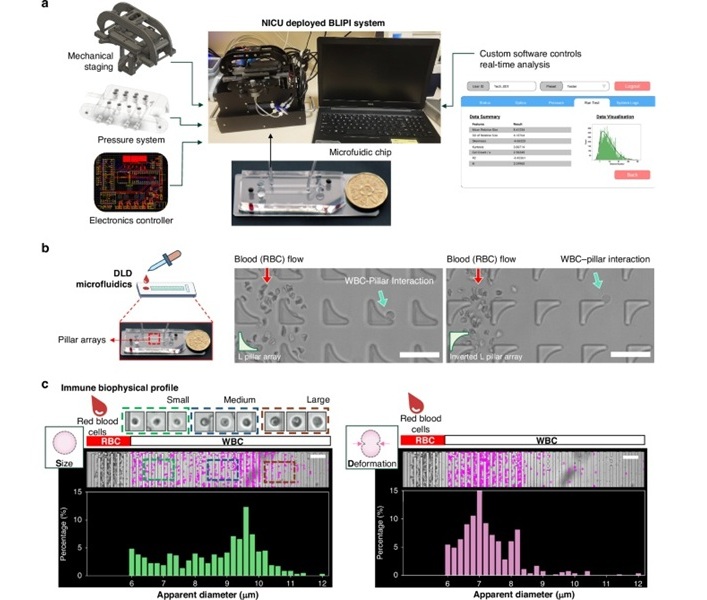
Dispositivo innovador analiza función inmunitaria de recién nacidos con una gota de sangre
Los bebés prematuros son muy susceptibles a enfermedades graves y potencialmente mortales, como la sepsis y la enterocolitis necrosante (ECN). La sepsis neonatal, una infección del torrente sanguíneo que... Más
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... MásMicrobiología
ver canal
Análisis de hemocultivo mejora gestión diagnóstica mediante selección de panel específico
Cada año, alrededor de 250.000 personas en los EUA son diagnosticadas con infecciones del torrente sanguíneo (ITS). La sepsis causada por estas infecciones conlleva una tasa de mortalidad... Más
Secuenciación genómica en tiempo real detecta superbacteria que causa infecciones hospitalarias
Las infecciones por superbacterias causadas por Staphylococcus aureus, o "estafilococo dorado", son notoriamente difíciles de tratar y se cobran más de un millón de vidas... MásPatología
ver canal
Modelo de IA detecta más de 170 tipos de cáncer
El diagnóstico de tumores cerebrales puede ser especialmente difícil cuando el tumor se localiza en una región donde las biopsias presentan un alto riesgo. En estos casos, donde no... Más
Prueba de hemoglobina para smartphones detecta con precisión cáncer colorrectal
A pesar de la disponibilidad de programas de detección del cáncer colorrectal, la participación sigue siendo baja, especialmente en las pruebas inmunoquímicas fecales (PIF),... MásTecnología
ver canal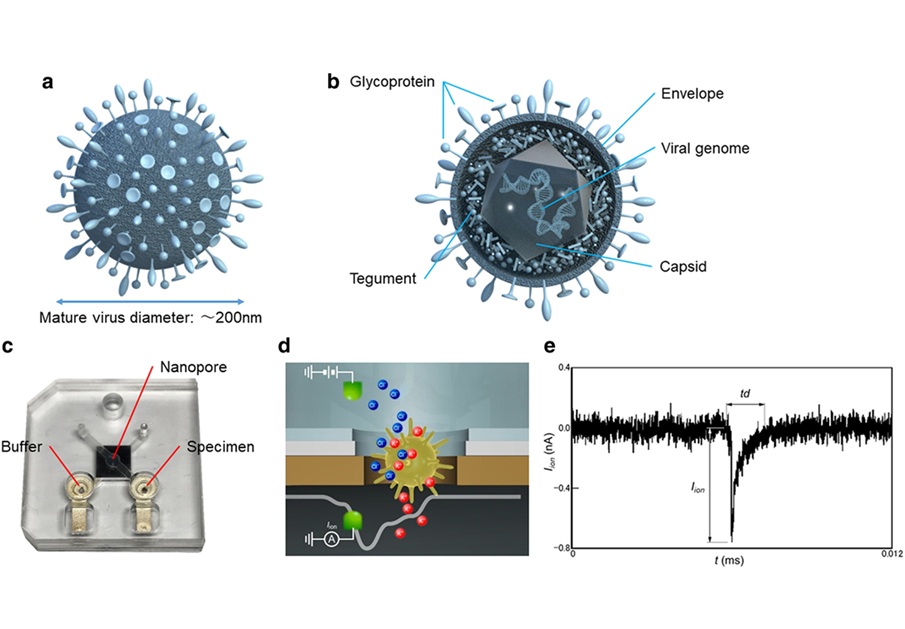
Prueba no basada en ADN con IA identifica infecciones virales
La confirmación de infecciones virales suele implicar pruebas de secuenciación de ADN, que tardan horas y pueden no estar disponibles in situ. Además, estas pruebas no pueden diferenciar... Más
Método de IA predice tasa de supervivencia de pacientes con cáncer de próstata
El adenocarcinoma de próstata (ACP) representa el 99 % de los diagnósticos de cáncer de próstata y es el segundo cáncer más común en hombres a nivel mundial,... MásIndustria
ver canal
Fujirebio colabora con Stanford Medicine para impulsar la investigación de enfermedades infecciosas
Fujirebio (Tokio, Japón) ha iniciado una colaboración con Stanford Medicine (Palo Alto, CA, EUA) para acelerar la adopción de inmunoensayos ultrasensibles que incorporan tecnología... Más