Secuenciación en células individuales revela diversidad clonal entre los pacientes con LMA
|
Por el equipo editorial de LabMedica en español Actualizado el 03 Nov 2020 |

Imagen: El sistema de secuenciación NovaSeq 6000 (Fotografía cortesía de Illumina).
Un cuerpo creciente de evidencia respalda el papel de la diversidad clonal en la resistencia terapéutica, la recurrencia y los malos resultados en el cáncer. La diversidad clonal también refleja la historia de la acumulación de mutaciones somáticas dentro de un tumor.
La capacidad de inferir heterogeneidad clonal y filogenia tumoral a partir de datos de secuenciación masiva es inherentemente limitada, porque las técnicas de secuenciación masiva no pueden inferir de manera confiable ocurrencias simultáneas de mutaciones y, por lo tanto, a menudo fallan en la reconstrucción exacta de la subestructura clonal. La secuenciación de ADN de una sola célula (scADN-seq) puede abordar algunos de estos desafíos.
Un gran equipo de científicos del Centro de Cáncer MD Anderson de la Universidad de Texas (Houston, TX, EUA), analizó 154 muestras (140 células mononucleares de médula ósea (BMMC) y 14 células mononucleares de sangre periférica) de 123 pacientes con leucemia mieloide aguda (LMA) que tenían al menos una mutación somática cubierta por el panel dirigido para scADN-seq. De los 123 pacientes, 108 pacientes fueron analizados antes del tratamiento (N = 98) o en el punto de tiempo de recaída/refractaria (N = 10). Entre 123 pacientes, 97 fueron analizados por scADN-seq, 23 fueron analizados por secuenciación simultánea de ADN de una sola célula y de las proteínas de superficie celular (scADN+protein-seq), y tres fueron analizados por scADN-seq y scADN+protein-seq.
La biblioteca agrupada se secuenció mediante una de las siguientes plataformas de secuenciación, MiSeq, HiSeq 4000 o NovaSeq 6000 (Illumina, San Diego, CA, EUA), con el procesamiento de pares de bases (pb) de 150 o 250 pares multiplexadas en el extremo. El equipo realizó una PCR digital por gotitas (ddPCR) utilizando el sistema digital QX200 Droplet (Bio-Rad Laboratories, Hércules, CA, EUA), para confirmar las variantes que fueron detectadas por scADN-seq, pero que no fueron detectadas por la secuenciación masiva. Se realizó un perfil simultáneo de la mutación del ADN y el inmunofenotipo de la superficie celular (scADN+protein-seq) utilizando el kit de panel de diseño personalizado y 10-15 anticuerpos oligoconjugados (Mission Bio, South San Francisco, CA, EUA). Los inmunofenotipos de las células de la médula ósea de pacientes con LMA se evaluaron mediante citometría de flujo de ocho colores en un instrumento FACSCanto II (BD Biosciences, San José, CA, EUA).
En total, los científicos secuenciaron más de 730.000 células para encontrar 543 mutaciones somáticas en 31 genes relacionados con el cáncer, el 98% de los cuales fueron validados ortogonalmente. Las mutaciones más comunes que detectaron fueron en NPM1, seguidas de las de DNMT3A y NRAS. Además, consiguieron que si bien se encontraron varias mutaciones que eran funcionalmente redundantes en los mismos pacientes, las alteraciones se encontraron a menudo en clones mutuamente excluyentes. Esto se extendió a las alteraciones que afectan a los genes de la vía de señalización del receptor tirosina quinasa (RTK)/Gas GTPasa (RAS)/MAP quinasa (MAPK), así como a las mutaciones IDH1 e IDH2 y a las mutaciones TET2 e IDH. Esto sugirió a los científicos que las células no necesitan dos mutaciones o que, cuando aparecen juntas, las mutaciones son tóxicas, lo que podría sugerir una posible vía de tratamiento para investigar.
Los investigadores también analizaron las correlaciones genotipo-fenotipo entre las células para encontrar, por ejemplo, que las células con mutaciones en NPM1 o IDH expresaban niveles más bajos de CD34 y HLA-DR, mientras que las células con una sola mutación en TP53 tenían un fenotipo CD34+ CD117+, pero que las mutaciones dobles en TP53 tenían un inmunofenotipo monocítico.
Koichi Takahashi, MD, PhD, autor principal del estudio, dijo: “Esta información también está disponible, de alguna manera, a partir de datos de secuenciación masiva longitudinal, pero creo que los datos de una sola célula proporcionan de manera única esta visión meticulosa de la dinámica clon por clon, lo que simplemente no es posible mediante la secuenciación masiva”. El estudio fue publicado el 21 de octubre de 2020 en la revista Nature Communications.
Enlace relacionado:
Centro de Cáncer MD Anderson de la Universidad de Texas
Bio-Rad Laboratories
Mission Bio
BD Biosciences
La capacidad de inferir heterogeneidad clonal y filogenia tumoral a partir de datos de secuenciación masiva es inherentemente limitada, porque las técnicas de secuenciación masiva no pueden inferir de manera confiable ocurrencias simultáneas de mutaciones y, por lo tanto, a menudo fallan en la reconstrucción exacta de la subestructura clonal. La secuenciación de ADN de una sola célula (scADN-seq) puede abordar algunos de estos desafíos.
Un gran equipo de científicos del Centro de Cáncer MD Anderson de la Universidad de Texas (Houston, TX, EUA), analizó 154 muestras (140 células mononucleares de médula ósea (BMMC) y 14 células mononucleares de sangre periférica) de 123 pacientes con leucemia mieloide aguda (LMA) que tenían al menos una mutación somática cubierta por el panel dirigido para scADN-seq. De los 123 pacientes, 108 pacientes fueron analizados antes del tratamiento (N = 98) o en el punto de tiempo de recaída/refractaria (N = 10). Entre 123 pacientes, 97 fueron analizados por scADN-seq, 23 fueron analizados por secuenciación simultánea de ADN de una sola célula y de las proteínas de superficie celular (scADN+protein-seq), y tres fueron analizados por scADN-seq y scADN+protein-seq.
La biblioteca agrupada se secuenció mediante una de las siguientes plataformas de secuenciación, MiSeq, HiSeq 4000 o NovaSeq 6000 (Illumina, San Diego, CA, EUA), con el procesamiento de pares de bases (pb) de 150 o 250 pares multiplexadas en el extremo. El equipo realizó una PCR digital por gotitas (ddPCR) utilizando el sistema digital QX200 Droplet (Bio-Rad Laboratories, Hércules, CA, EUA), para confirmar las variantes que fueron detectadas por scADN-seq, pero que no fueron detectadas por la secuenciación masiva. Se realizó un perfil simultáneo de la mutación del ADN y el inmunofenotipo de la superficie celular (scADN+protein-seq) utilizando el kit de panel de diseño personalizado y 10-15 anticuerpos oligoconjugados (Mission Bio, South San Francisco, CA, EUA). Los inmunofenotipos de las células de la médula ósea de pacientes con LMA se evaluaron mediante citometría de flujo de ocho colores en un instrumento FACSCanto II (BD Biosciences, San José, CA, EUA).
En total, los científicos secuenciaron más de 730.000 células para encontrar 543 mutaciones somáticas en 31 genes relacionados con el cáncer, el 98% de los cuales fueron validados ortogonalmente. Las mutaciones más comunes que detectaron fueron en NPM1, seguidas de las de DNMT3A y NRAS. Además, consiguieron que si bien se encontraron varias mutaciones que eran funcionalmente redundantes en los mismos pacientes, las alteraciones se encontraron a menudo en clones mutuamente excluyentes. Esto se extendió a las alteraciones que afectan a los genes de la vía de señalización del receptor tirosina quinasa (RTK)/Gas GTPasa (RAS)/MAP quinasa (MAPK), así como a las mutaciones IDH1 e IDH2 y a las mutaciones TET2 e IDH. Esto sugirió a los científicos que las células no necesitan dos mutaciones o que, cuando aparecen juntas, las mutaciones son tóxicas, lo que podría sugerir una posible vía de tratamiento para investigar.
Los investigadores también analizaron las correlaciones genotipo-fenotipo entre las células para encontrar, por ejemplo, que las células con mutaciones en NPM1 o IDH expresaban niveles más bajos de CD34 y HLA-DR, mientras que las células con una sola mutación en TP53 tenían un fenotipo CD34+ CD117+, pero que las mutaciones dobles en TP53 tenían un inmunofenotipo monocítico.
Koichi Takahashi, MD, PhD, autor principal del estudio, dijo: “Esta información también está disponible, de alguna manera, a partir de datos de secuenciación masiva longitudinal, pero creo que los datos de una sola célula proporcionan de manera única esta visión meticulosa de la dinámica clon por clon, lo que simplemente no es posible mediante la secuenciación masiva”. El estudio fue publicado el 21 de octubre de 2020 en la revista Nature Communications.
Enlace relacionado:
Centro de Cáncer MD Anderson de la Universidad de Texas
Bio-Rad Laboratories
Mission Bio
BD Biosciences
Últimas Diagnóstico Molecular noticias
- Sencilla prueba de orina revolucionará diagnóstico y tratamiento del cáncer de vejiga
- Análisis snaguíneo para detección más temprana y sencilla de fibrosis hepática
- Marcador genético evita quimioterapia innecesaria a niños con leucemia de células T
- Prueba sanguínea de cuatro genes descarta infección pulmonar bacteriana
- Nueva prueba PCR mejora precisión diagnóstica de vaginosis bacteriana y vaginitis por Candida
- Nueva estrategia de edición de marcadores séricos mejora diagnóstico de enfermedades neurológicas
- Primera prueba genética de riesgo de diabetes tipo 1 permite detección temprana
- Análisis sanguíneo evita cirugías innecesarias en pacientes con cáncer gástrico de bajo riesgo
- Sistema automatizado pionero acelera diagnóstico del mieloma
- Perfiles de proteínas sanguíneas predicen riesgo de mortalidad por intervención médica temprana
- Primer análisis de sangre de su tipo detecta cáncer gástrico en pacientes asintomáticos
- Prueba molecular portátil detecta ITS en POC en 15 minutos
- Analizador de sobremesa ejecuta análisis químicos, inmunoensayos y hematología en un dispositivo
- Prueba POC de Bordetella ofrece resultados rápidos con precisión PCR
- Prueba sanguínea con punción detecta enfermedades 10 años antes de los síntomas
- Valores refinados de proteína C reactiva evaluan riesgo de sepsis en prematuros
Canales
Química Clínica
ver canal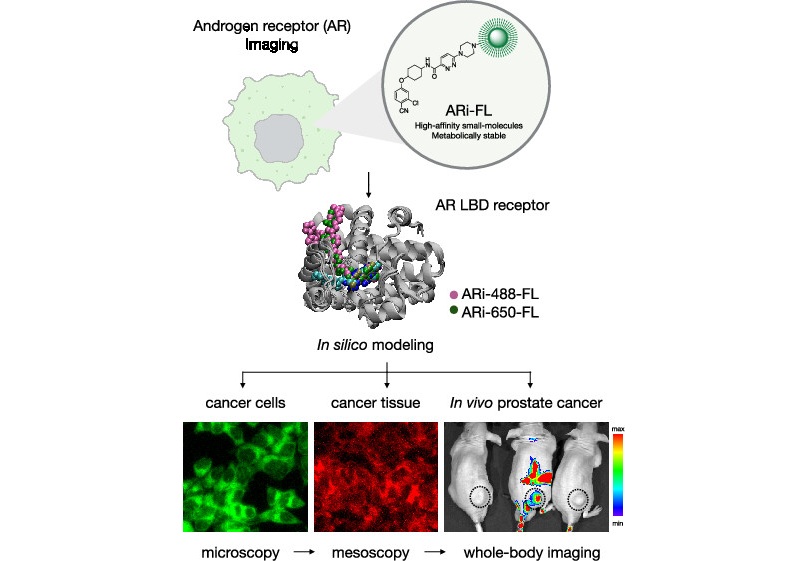
Sonda de imágenes químicas podría rastrear y tratar cáncer de próstata
El cáncer de próstata sigue siendo una de las principales causas de enfermedad y muerte en hombres, y muchos pacientes desarrollan resistencia a las terapias hormonales bloqueantes convencionales.... Más
Discrepancia entre dos pruebas comunes de función renal indica problemas de salud graves
La creatinina ha sido durante mucho tiempo el método estándar para medir la filtración renal, mientras que la cistatina C, una proteína producida por todas las células humanas, se ha recomendado como marcador... MásHematología
ver canal
Análisis sanguíneo de actividad plaquetaria en mediana edad podría identificar riesgo temprano de Alzheimer
La detección temprana de la enfermedad de Alzheimer sigue siendo una de las mayores necesidades insatisfechas en neurología, sobre todo porque los cambios biológicos que subyacen al... Más
Medición de microvesículas podría detectar lesiones vasculares en pacientes con anemia falciforme
Evaluar la gravedad de la enfermedad de células falciformes (ECF) sigue siendo un reto, sobre todo al intentar predecir la hemólisis, el daño vascular y el riesgo de complicaciones... MásInmunología
ver canal
Nueva prueba distingue falsos positivos inducidos por vacuna de infección activa por VIH
Desde que se identificó el VIH en 1983, más de 91 millones de personas han contraído el virus y más de 44 millones han fallecido por causas relacionadas. Hoy en día, casi 40 millones de personas en todo... Más
Prueba de firma genética predice respuesta a tratamientos clave para cáncer de mama
Los inhibidores de DK4/6, combinados con terapia hormonal, se han convertido en un tratamiento fundamental para el cáncer de mama avanzado HR+/HER2–, ya que ralentizan el crecimiento tumoral... MásMicrobiología
ver canal
Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
La sepsis causa la muerte de 11 millones de personas en todo el mundo cada año y genera enormes costos de atención médica. Solo en EUA y Europa, la sepsis representa 100.... MásPrueba rápida de tuberculosis POC proporciona resultados en 15 minutos
La tuberculosis sigue siendo una de las enfermedades infecciosas más mortales del mundo, y la reducción de nuevos casos depende de la identificación de las personas con infección... MásPatología
ver canal
Dispositivo de clasificación celular sintonizable tiene potencial para aplicaciones biomédicas
Aislar células cancerosas raras de la sangre es esencial para diagnosticar metástasis y orientar las decisiones terapéuticas, pero sigue siendo un desafío técnico.... MásHerramienta de IA mejora a medicos detectando anomalías en células sanguíneas
El diagnóstico de trastornos sanguíneos depende del reconocimiento de anomalías sutiles en el tamaño, la forma y la estructura celular. Sin embargo, este proceso es lento, subjetivo y requiere años de... Más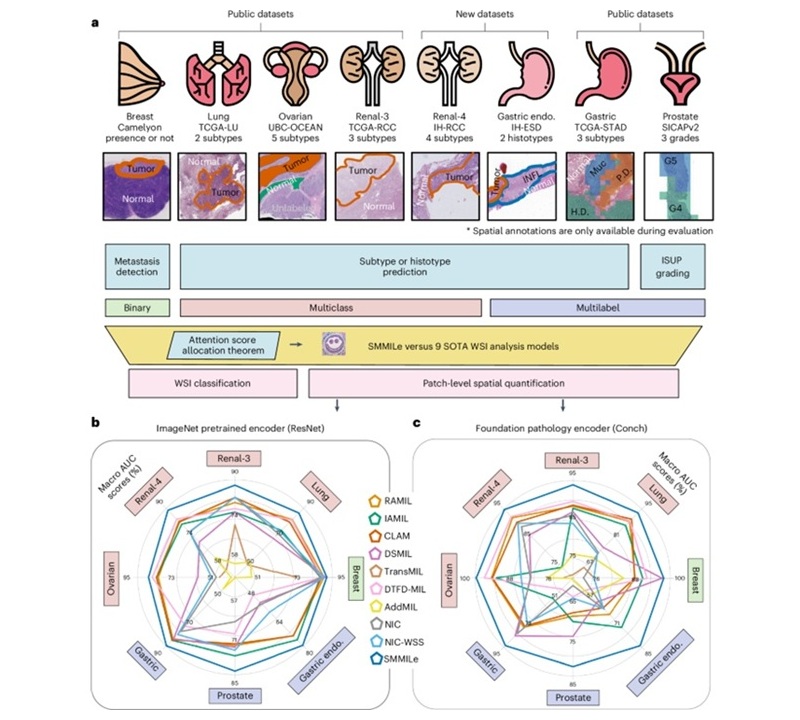
Herramienta de IA analiza rápidamente imágenes complejas de cáncer para tratamiento personalizado
Las imágenes complejas de biopsia digital, que normalmente requieren hasta 20 minutos para ser evaluadas por un patólogo experto, ahora pueden analizarse en aproximadamente un minuto mediante una nueva... MásTecnología
ver canal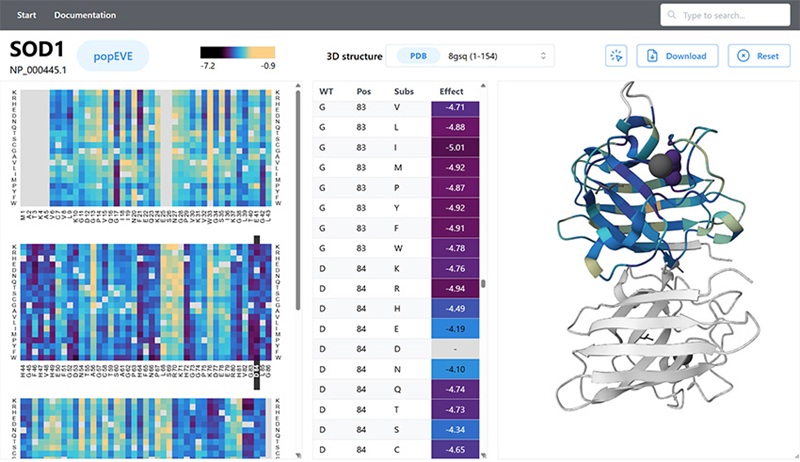
Modelo de inteligencia artificial podría acelerar diagnóstico de enfermedades raras
Identificar qué variantes genéticas causan enfermedades sigue siendo uno de los mayores desafíos de la medicina genómica. Cada persona porta decenas de miles de cambios en el... Más
Sensor de saliva con IA permite detección precoz del cáncer de cabeza y cuello
La detección precoz del cáncer de cabeza y cuello sigue siendo difícil porque la enfermedad produce pocos o ningún síntoma en sus primeras etapas, y las lesiones a menudo... MásIndustria
ver canal
Abbott adquiere Exact Sciences, empresa de detección de cáncer
Abbott (Abbott Park, IL, EUA) ha firmado un acuerdo definitivo para adquirir Exact Sciences (Madison, WI, EUA), lo que le permitirá entrar y liderar en segmentos de diagnóstico de cáncer... Más