Sistema de microarrays basados en CRISPR permite pruebas masivas para patógenos virales
|
Por el equipo editorial de LabMedica en español Actualizado el 11 May 2020 |

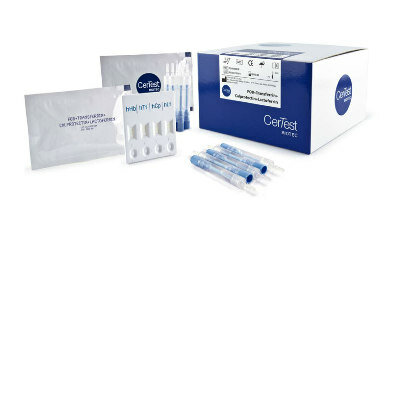
Imagen: Fotografía del chip de micropozos CARMEN (Fotografía cortesía de Michael James Butts)
Un novedoso sistema de diagnóstico microfluídico basado en ácidos nucleicos es capaz de detectar un virus específico de un catálogo de más de 160 patógenos humanos, incluido el coronavirus COVID-19, simultáneamente en más de mil muestras.
Para permitir la vigilancia de rutina y las aplicaciones de diagnóstico integrales, existe la necesidad de tecnologías de detección que se puedan ampliar para analizar muchas muestras y, al mismo tiempo, detectar múltiples patógenos individuales. Para lograr esto, los investigadores del Instituto Broad del MIT y Harvard (Cambridge, MA, EUA) desarrollaron una plataforma de diagnóstico llamada CARMEN (sigla en inglés para Reacciones Arregladas Combinadas para la Evaluación Multiplexada de Ácidos Nucleicos). El sistema CARMEN depende de gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR/Cas 13.
Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente y separadas entre sí) son segmentos de ADN procariótico que contienen repeticiones cortas de secuencias de bases. Cada repetición es seguida por segmentos cortos de “ADN espaciador” de exposiciones previas a un virus o plásmido bacteriano.
Los esfuerzos computacionales recientes para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida al ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación de ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, dirigido por el ARN contra el ARN y que tiene un gran potencial para aplicaciones precisas, robustas y escalables de orientación por el ARN contra el ARN.
La plataforma CARMEN consta de un chip de goma, un poco más grande que un teléfono inteligente, que contiene decenas de miles de micropozos diseñados para contener un par de gotitas del tamaño de nanolitros. Una gota contiene material genético viral de una muestra y la otra contiene reactivos de detección de virus.
La detección de ácidos nucleicos virales se realiza mediante una modificación del protocolo SHERLOCK. Este es un método para la detección de moléculas individuales de objetivos de ácido nucleico y significa desbloqueo específico del reportero enzimático de alta sensibilidad. Funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también se puede cuantificar. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple prueba de tira de papel, en equipos de laboratorio o para proporcionar una lectura electroquímica que se pueda leer con un teléfono móvil.
En la plataforma CARMEN, las gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR se autoorganizan en la matriz de micropozos para emparejarse con las gotas de muestras amplificadas, analizando cada muestra contra cada ARN CRISPR (ARNcr) en replicado. La combinación de detección CARMEN y Cas13 (CARMEN-Cas13) permitió realizar pruebas robustas de más de 4.500 pares de ARNc-objetivo en una sola matriz. El protocolo completo, desde la extracción de ARN hasta los resultados, requirió menos de ocho horas.
Empleando el método CARMEN-Cas13, los investigadores desarrollaron un ensayo multiplexado que simultáneamente diferenciaba 169 virus asociados con humanos con más de 10 secuencias genómicas publicadas e incorporaba rápidamente un ARNcr adicional para detectar el agente causante del coronavirus pandémico COVID-19. CARMEN-Cas13 permitió además el subtipo integral de cepas de influenza A y la identificación multiplexada de docenas de mutaciones de resistencia a los medicamentos contra el VIH.
“Este enfoque miniaturizado para el diagnóstico es eficiente en recursos y fácil de implementar”, dijo el coautor principal, el Dr. Paul Blainey, profesor asociado de ingeniería biológica en el Instituto de Tecnología de Massachusetts. “Las nuevas herramientas requieren creatividad e innovación, y con estos avances en química y microfluídica, estamos entusiasmados con el potencial de CARMEN a medida que la comunidad trabaja para vencer a la COVID-19 y las futuras amenazas de enfermedades infecciosas”.
El método CARMEN-Cas13 se describió en la edición en línea del 29 de abril de 2020 de la revista Nature.
Enlace relacionado:
Instituto Broad del MIT y Harvard
Para permitir la vigilancia de rutina y las aplicaciones de diagnóstico integrales, existe la necesidad de tecnologías de detección que se puedan ampliar para analizar muchas muestras y, al mismo tiempo, detectar múltiples patógenos individuales. Para lograr esto, los investigadores del Instituto Broad del MIT y Harvard (Cambridge, MA, EUA) desarrollaron una plataforma de diagnóstico llamada CARMEN (sigla en inglés para Reacciones Arregladas Combinadas para la Evaluación Multiplexada de Ácidos Nucleicos). El sistema CARMEN depende de gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR/Cas 13.
Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente y separadas entre sí) son segmentos de ADN procariótico que contienen repeticiones cortas de secuencias de bases. Cada repetición es seguida por segmentos cortos de “ADN espaciador” de exposiciones previas a un virus o plásmido bacteriano.
Los esfuerzos computacionales recientes para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida al ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación de ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, dirigido por el ARN contra el ARN y que tiene un gran potencial para aplicaciones precisas, robustas y escalables de orientación por el ARN contra el ARN.
La plataforma CARMEN consta de un chip de goma, un poco más grande que un teléfono inteligente, que contiene decenas de miles de micropozos diseñados para contener un par de gotitas del tamaño de nanolitros. Una gota contiene material genético viral de una muestra y la otra contiene reactivos de detección de virus.
La detección de ácidos nucleicos virales se realiza mediante una modificación del protocolo SHERLOCK. Este es un método para la detección de moléculas individuales de objetivos de ácido nucleico y significa desbloqueo específico del reportero enzimático de alta sensibilidad. Funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también se puede cuantificar. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple prueba de tira de papel, en equipos de laboratorio o para proporcionar una lectura electroquímica que se pueda leer con un teléfono móvil.
En la plataforma CARMEN, las gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR se autoorganizan en la matriz de micropozos para emparejarse con las gotas de muestras amplificadas, analizando cada muestra contra cada ARN CRISPR (ARNcr) en replicado. La combinación de detección CARMEN y Cas13 (CARMEN-Cas13) permitió realizar pruebas robustas de más de 4.500 pares de ARNc-objetivo en una sola matriz. El protocolo completo, desde la extracción de ARN hasta los resultados, requirió menos de ocho horas.
Empleando el método CARMEN-Cas13, los investigadores desarrollaron un ensayo multiplexado que simultáneamente diferenciaba 169 virus asociados con humanos con más de 10 secuencias genómicas publicadas e incorporaba rápidamente un ARNcr adicional para detectar el agente causante del coronavirus pandémico COVID-19. CARMEN-Cas13 permitió además el subtipo integral de cepas de influenza A y la identificación multiplexada de docenas de mutaciones de resistencia a los medicamentos contra el VIH.
“Este enfoque miniaturizado para el diagnóstico es eficiente en recursos y fácil de implementar”, dijo el coautor principal, el Dr. Paul Blainey, profesor asociado de ingeniería biológica en el Instituto de Tecnología de Massachusetts. “Las nuevas herramientas requieren creatividad e innovación, y con estos avances en química y microfluídica, estamos entusiasmados con el potencial de CARMEN a medida que la comunidad trabaja para vencer a la COVID-19 y las futuras amenazas de enfermedades infecciosas”.
El método CARMEN-Cas13 se describió en la edición en línea del 29 de abril de 2020 de la revista Nature.
Enlace relacionado:
Instituto Broad del MIT y Harvard
Últimas Tecnología noticias
- Algoritmo de firma ligera permite diagnósticos médicos más rápidos y precisos
- Tecnología de microchip desechable podría detectar selectivamente VIH en muestras de sangre completa
- Dispositivo microfluídico Dolor en un Chip determina tipos de dolor crónico desde muestras de sangre
- Innovador sensor fluorométrico sin etiquetas permite detección más sensible del ARN viral
- Teléfonos inteligentes podrían diagnosticar enfermedades mediante escáneres infrarrojos
- Nueva tecnología de sensores permite diagnóstico temprano de trastornos metabólicos y cardiovasculares
- Avance en impresión 3D permite desarrollo a gran escala de diminutos dispositivos microfluídicos
- Plataforma de sensores en papel transforma diagnóstico cardíaco
- Estudio explora impacto de pruebas POC en el futuro de los diagnósticos
- Sensor económico de respuesta rápida permite detección temprana y precisa del cáncer de pulmón
- Nanotecnología para diagnósticar cáncer de cuello uterino podría sustituir pruebas de Papanicolaou
- Plataforma de laboratorio en chip agilizar diagnóstico del cáncer
- Plataforma de biosensores detecta simultáneamente vitamina C y SARS-CoV-2
- Nuevo método analiza lágrimas para detectar enfermedades de forma temprana
- Sensores basados en FET abren camino a dispositivos de diagnóstico portátiles para detectar múltiples enfermedades
- Biosensor basado en papel para detectar glucosa mediante sudor revoluciona tratamiento de diabetes
Canales
Química Clínica
ver canal
Monitorización con espectrometría de masas predice e identifica recaída temprana del mieloma
El mieloma, un tipo de cáncer que afecta la médula ósea, es actualmente incurable, aunque muchos pacientes pueden vivir más de 10 años tras el diagnóstico.... Más
Herramienta química a nanoescala 'brillantemente luminosa' mejora detección de enfermedades
Miles de moléculas brillantes disponibles comercialmente, conocidas como fluoróforos, se utilizan comúnmente en imágenes médicas, detección de enfermedades, marcado... MásDiagnóstico Molecular
ver canal
Herramienta genética predice supervivencia de pacientes con cáncer de páncreas
Un marcador tumoral es una sustancia presente en el organismo que puede indicar la presencia de cáncer. Estas sustancias, que pueden incluir proteínas, genes, moléculas u otros compuestos... Más
Prueba de orina diagnostica cáncer de próstata inicial
El cáncer de próstata es una de las principales causas de muerte en hombres a nivel mundial. Un desafío importante para diagnosticar la enfermedad es la ausencia de biomarcadores confiables... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... Más
Análisis de sangre con aprendizaje automático predice respuesta a inmunoterapia en pacientes con linfoma
La terapia de células T con receptores de antígenos quiméricos (CAR) se ha convertido en uno de los avances recientes más prometedores en el tratamiento de los cánceres... MásPatología
ver canal
Modelo de IA predice respuesta a terapia contra cáncer de riñón
Cada año, cerca de 435.000 personas son diagnosticadas con carcinoma renal de células claras (CRcc), lo que lo convierte en el subtipo más prevalente de cáncer de riñón.... Más
Kits de ensayo de enzima DUB sensibles y específicos requieren configuración mínima sin preparación del sustrato
La ubiquitinación y la desubiquitinación son dos procesos fisiológicos importantes en el sistema ubiquitina-proteasoma, responsable de la degradación de proteínas en... MásTecnología
ver canal
Algoritmo de firma ligera permite diagnósticos médicos más rápidos y precisos
Cada material o molécula interactúa con la luz de forma única, creando un patrón distintivo, similar a una huella dactilar. La espectroscopia óptica, que consiste en... Más
Tecnología de microchip desechable podría detectar selectivamente VIH en muestras de sangre completa
A finales de 2023, aproximadamente 40 millones de personas en todo el mundo vivían con VIH, y alrededor de 630.000 personas murieron por enfermedades relacionadas con el sida ese mismo año.... MásIndustria
ver canal
Cepheid y Oxford Nanopore se unen para desarrollar soluciones con secuenciación automatizada
Cepheid (Sunnyvale, CA, EUA), una empresa líder en diagnóstico molecular, y Oxford Nanopore Technologies (Oxford, Reino Unido), la empresa detrás de una nueva generación de... Más