Análisis de los tumores metastásicos conduce a objetivos de tratamiento potenciales
|
Por el equipo editorial de LabMedica en español Actualizado el 12 Nov 2019 |

Imagen: La Serie HiSeq X incorpora tecnología de celdas de flujo con diseño para generar un nivel excepcional de eficiencia para la secuenciación del genoma completo (Fotografía cortesía de Illumina).
El cáncer metastásico es una causa importante de muerte y se asocia con una mala eficacia del tratamiento. Se requiere una comprensión mejor de las características del cáncer en etapa tardía para ayudar a adaptar los tratamientos personalizados, reducir los tratamientos exagerados y mejorar los resultados.
En los últimos años, varios esfuerzos de análisis de secuenciación de genoma completo (WGS) a gran escala, han arrojado conocimientos valiosos sobre la diversidad de los procesos moleculares que impulsan los diferentes tipos de cáncer de adultos y pediátricos, y han alimentado las promesas de poder ofrecer atención oncológica basada en el genoma.
Un gran equipo de científicos liderados por la Fundación Médica Hartwig (Ámsterdam, Países Bajos) incluyó en un estudio a pacientes con cáncer avanzado que no eran curables por las opciones locales de tratamiento, que eran candidatos para cualquier tipo de tratamiento sistémico y que habían recibido cualquier línea de tratamiento como parte de dos estudios clínicos. Se tomaron muestras de las biopsias con aguja gruesa de la lesión metastásica, o cuando se consideró no factible o no seguro, del sitio del tumor primario y se congelaron en nitrógeno líquido. Un patólogo experimentado recolectó una sola sección de 6 μm para la tinción de hematoxilina y eosina (H-E) y la estimación de la celularidad del tumor y se recogieron 25 cortes de 20 μm en un tubo para aislar el ADN.
Los investigadores aislaron el ADN de biopsias (> 30% de celularidad tumoral y de sangre) usando el kit DSP DNA Midi para sangre y el Mini kit QIAsymphony DSP DNA para tejidos (Qiagen, Hilden, Alemania). Se utilizó un total de 50-200 ng de ADN (cortado a una longitud de fragmento promedio de 450nt) como entrada para la preparación de la biblioteca TruSeq Nano LT (Illumina, San Diego, CA, EUA). Las bibliotecas con código de barras se secuenciaron como agrupaciones en el Illumina HiSeqX generando 2 × 150 pares de lectura utilizando configuraciones estándar. Se realizaron varios otros métodos para dilucidar el estudio.
Los científicos secuenciaron los genomas de 2.520 muestras tumorales y lo compararon con el tejido normal de 2.399 individuos con cáncer metastásico . Al examinar más de 70 millones de cambios somáticos en los tumores, incluidas mutaciones puntuales, pequeñas inserciones y deleciones, cambios en el número de copias y otras características, pudieron diferenciar entre las mutaciones presentes en las primeras etapas de la enfermedad y las que surgieron durante el tratamiento. Las secuencias descubrieron diferencias de mutación somática de un tipo de cáncer al siguiente, por ejemplo, incluyendo un gran número de mutaciones puntuales en tumores derivados de cáncer de pulmón o melanoma. También destacaron los cambios del gen conductor y los genes mutados recurrentemente que parecen ser importantes en el entorno del cáncer metastásico.
En más de la mitad de las metástasis, por ejemplo, el equipo descubrió eventos de duplicación del genoma completo, con hasta el 80% de los tumores esofágicos mostrando estas duplicaciones. Mientras tanto, alrededor del 62% de los pacientes tenían alteraciones que fueron marcadas como clínicamente accionables; ya sea usando tratamientos aprobados o drogas experimentales. Las alteraciones identificadas en el 18% de los pacientes condujeron a estrategias de tratamiento en la etiqueta, informó el equipo, mientras que aproximadamente el 13% tenía mutaciones que podrían ser susceptibles de tratamiento sin tener en cuenta la etiqueta. Otro 31% de los casos involucró alteraciones tumorales que coincidieron con la elegibilidad para ensayos clínicos específicos.
Los autores concluyeron que sus resultados demostraron que el análisis de secuenciación del genoma completo del cáncer metastásico puede proporcionar ideas novedosas y relevantes, y son fundamentales para abordar algunos de los desafíos clave en la medicina de precisión en el cáncer y que el conjunto de muestras analizado para el estudio actual proporciona un recurso complementario valioso para datos basados en la secuencia del genoma completo de los tumores primarios. El estudio fue publicado el 23 de octubre de 2019 en la revista Nature.
Enlace relacionado:
Fundación Médica Hartwig
Qiagen
Illumina
En los últimos años, varios esfuerzos de análisis de secuenciación de genoma completo (WGS) a gran escala, han arrojado conocimientos valiosos sobre la diversidad de los procesos moleculares que impulsan los diferentes tipos de cáncer de adultos y pediátricos, y han alimentado las promesas de poder ofrecer atención oncológica basada en el genoma.
Un gran equipo de científicos liderados por la Fundación Médica Hartwig (Ámsterdam, Países Bajos) incluyó en un estudio a pacientes con cáncer avanzado que no eran curables por las opciones locales de tratamiento, que eran candidatos para cualquier tipo de tratamiento sistémico y que habían recibido cualquier línea de tratamiento como parte de dos estudios clínicos. Se tomaron muestras de las biopsias con aguja gruesa de la lesión metastásica, o cuando se consideró no factible o no seguro, del sitio del tumor primario y se congelaron en nitrógeno líquido. Un patólogo experimentado recolectó una sola sección de 6 μm para la tinción de hematoxilina y eosina (H-E) y la estimación de la celularidad del tumor y se recogieron 25 cortes de 20 μm en un tubo para aislar el ADN.
Los investigadores aislaron el ADN de biopsias (> 30% de celularidad tumoral y de sangre) usando el kit DSP DNA Midi para sangre y el Mini kit QIAsymphony DSP DNA para tejidos (Qiagen, Hilden, Alemania). Se utilizó un total de 50-200 ng de ADN (cortado a una longitud de fragmento promedio de 450nt) como entrada para la preparación de la biblioteca TruSeq Nano LT (Illumina, San Diego, CA, EUA). Las bibliotecas con código de barras se secuenciaron como agrupaciones en el Illumina HiSeqX generando 2 × 150 pares de lectura utilizando configuraciones estándar. Se realizaron varios otros métodos para dilucidar el estudio.
Los científicos secuenciaron los genomas de 2.520 muestras tumorales y lo compararon con el tejido normal de 2.399 individuos con cáncer metastásico . Al examinar más de 70 millones de cambios somáticos en los tumores, incluidas mutaciones puntuales, pequeñas inserciones y deleciones, cambios en el número de copias y otras características, pudieron diferenciar entre las mutaciones presentes en las primeras etapas de la enfermedad y las que surgieron durante el tratamiento. Las secuencias descubrieron diferencias de mutación somática de un tipo de cáncer al siguiente, por ejemplo, incluyendo un gran número de mutaciones puntuales en tumores derivados de cáncer de pulmón o melanoma. También destacaron los cambios del gen conductor y los genes mutados recurrentemente que parecen ser importantes en el entorno del cáncer metastásico.
En más de la mitad de las metástasis, por ejemplo, el equipo descubrió eventos de duplicación del genoma completo, con hasta el 80% de los tumores esofágicos mostrando estas duplicaciones. Mientras tanto, alrededor del 62% de los pacientes tenían alteraciones que fueron marcadas como clínicamente accionables; ya sea usando tratamientos aprobados o drogas experimentales. Las alteraciones identificadas en el 18% de los pacientes condujeron a estrategias de tratamiento en la etiqueta, informó el equipo, mientras que aproximadamente el 13% tenía mutaciones que podrían ser susceptibles de tratamiento sin tener en cuenta la etiqueta. Otro 31% de los casos involucró alteraciones tumorales que coincidieron con la elegibilidad para ensayos clínicos específicos.
Los autores concluyeron que sus resultados demostraron que el análisis de secuenciación del genoma completo del cáncer metastásico puede proporcionar ideas novedosas y relevantes, y son fundamentales para abordar algunos de los desafíos clave en la medicina de precisión en el cáncer y que el conjunto de muestras analizado para el estudio actual proporciona un recurso complementario valioso para datos basados en la secuencia del genoma completo de los tumores primarios. El estudio fue publicado el 23 de octubre de 2019 en la revista Nature.
Enlace relacionado:
Fundación Médica Hartwig
Qiagen
Illumina
Últimas Diagnóstico Molecular noticias
- Análisis de ADN dos en uno mejora precisión diagnóstica y ahorra tiempo y costes
- Nueva herramienta mapea cambios cromosómicos en células cancerosas para predecir evolución de tumores
- Análisis sanguíneo identifica pacientes con cáncer mamario inflamatorio con mayor riesgo de metástasis cerebral
- Biomarcadores de Parkinson recientemente identificados permitirán diagnóstico temprano mediante análisis sanguíneo
- Nuevo análisis snaguíneo podría detectar cáncer de páncreas en etapa más tratable
- Primer análisis de sangre del mundo distingue entre nódulos pulmonares benignos y cancerosos
- Biopsia líquida podría reemplazar a biopsia quirúrgica para diagnosticar linfoma nervioso central primario
- Herramienta revela debilidad metabólica oculta en cánceres de sangre
- Prueba rápida utiliza teléfono móvil para identificar casos graves de malaria importada en minutos
- Análisis sanguíneo detecta historial de exposición a infecciones
- Prueba de ERM rastrea respuesta a cirugía de cáncer de vejiga
- Firmas del microbioma intestinal predicen resultados a largo plazo en pancreatitis aguda
- Análisis sanguíneo podría identificar personas con alto riesgo de diabetes tipo 2
- Análisis sanguíneo promete respuestas más rápidas a infecciones fúngicas mortales
- Análisis sanguíneo podría detectar códigos de barras moleculares que distinguen tipos de cáncer
- Algoritmo de IA predice metástasis del cáncer y riesgo de recurrencia
Canales
Química Clínica
ver canal
Modelo pronóstico basado en APE mejora evaluación del riesgo de cáncer
El cáncer de próstata es la segunda causa principal de muerte por cáncer en los hombres estadounidenses, y aproximadamente uno de cada ocho será diagnosticado a lo largo de su vida.... Más
Vesículas extracelulares se relacionan con riesgo de insuficiencia cardíaca en pacientes con ERC
La enfermedad renal crónica (ERC) afecta a más de 1 de cada 7 estadounidenses y está estrechamente asociada con complicaciones cardiovasculares, las cuales representan más de... MásHematología
ver canal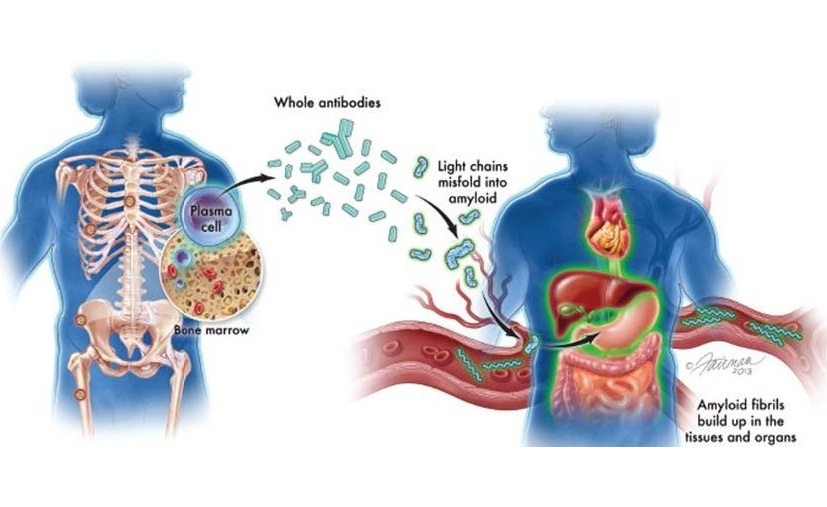
Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
La amiloidosis de cadenas ligeras (AL) es un trastorno poco común y potencialmente mortal de la médula ósea, en el que las proteínas amiloides anormales se acumulan en los órganos. Aproximadamente 3.... Más
Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
Las secciones de hemostasia de alto volumen requieren una rápida respuesta, a la vez que gestionan repeticiones y pruebas reflejas. La manipulación de los tubos y las comprobaciones prea... MásInmunología
ver canalAnálisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
El cáncer de pulmón de células pequeñas (CPCP) es una enfermedad agresiva con opciones de tratamiento limitadas, e incluso las inmunoterapias recientemente aprobadas no benefician... Más
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... MásMicrobiología
ver canal
Nuevo estudio identifica características del microbioma intestinal asociadas con enfermedad de Alzheimer
La enfermedad de Alzheimer afecta a aproximadamente 6,7 millones de personas en Estados Unidos y a casi 50 millones en todo el mundo; sin embargo, el deterioro cognitivo temprano sigue siendo difícil de... MásPlataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
Las infecciones causadas por la levadura patógena Candida auris representan una amenaza significativa para los pacientes hospitalizados, en particular para aquellos con sistemas inmunitarios debilitados... MásPatología
ver canal
Prueba pionera identifica riesgo de autismo al nacer
El trastorno del espectro autista es tratable, y una amplia investigación demuestra que la intervención temprana puede mejorar significativamente los resultados cognitivos, sociales y conductuales.... Más
IA mejora detección de mutaciones genéticas en diagnóstico del cáncer
La identificación precisa de mutaciones genéticas es fundamental para el diagnóstico del cáncer y la investigación genómica, pero los métodos actuales presentan... MásTecnología
ver canal
Tecnología robótica para extracciones de sangre diagnósticas automatizadas
La extracción rutinaria de sangre para diagnóstico es una tarea de alto volumen que puede sobrecargar al personal e introducir variabilidad dependiente del factor humano, con implicaciones... Más
ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
Los laboratorios clínicos generan miles de millones de resultados de pruebas cada año, lo que crea un cúmulo de datos con el potencial de facilitar pruebas más personalizadas,... MásIndustria
ver canal
Prueba de cáncer cervical con IA lista para lanzamiento en América Latina
Noul Co., una empresa coreana especializada en diagnósticos de sangre y cáncer basados en IA, anunció que suministrará su solución de diagnóstico de cáncer... MásNueva colaboración lleva espectrometría de masas automatizada a pruebas de laboratorio de rutina
La espectrometría de masas es una potente técnica analítica que identifica y cuantifica moléculas en función de su masa y carga eléctrica. Su alta selectividad,... Más
Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
Diasorin (Saluggia, Italia) ha firmado un acuerdo de distribución exclusivo con Fisher Scientific, parte de Thermo Fisher Scientific (Waltham, MA, EUA), para la plataforma molecular de punto de... Más