Subespecies de malaria caracterizadas por novedoso análisis molecular
|
Por el equipo editorial de LabMedica en español Actualizado el 03 Mar 2015 |
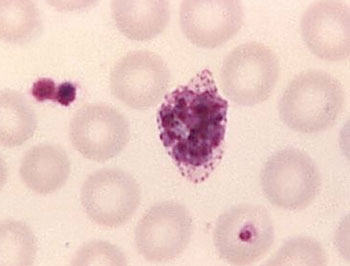
Imagen: Un trofozoito maduro de Plasmodium ovale en un extendido de sangre (Fotografía cortesía de los CDC).
Las subespecies de Plasmodium ovale son similares, basado en la morfología y la distribución geográfica, pero las diferencias alélicas indican que P. ovale curtisi y P. ovale wallikeri son genéticamente divergentes.
Las diferencias en duración y latencia clínicas potenciales, entre P. ovale curtisi y P. ovale wallikeri demuestran la necesidad de investigación sobre la contribución de este parásito desatendido de la malaria a la carga mundial de la malaria.
Científicos de la Universidad de Servicios Uniformados (Bethesda, MD, EUA) quienes trabajaron con colegas en Kenia, recolectaron muestras de sangre total de individuos adultos asintomáticos clínicamente sanos, en la Provincia de Nyanza, Kenia. Las muestras fueron estudiadas inicialmente con la prueba de Diagnóstico Rápido Parascreen Pan/Pf malaria (Zephyr Biomedicals, Verna, Goa, India), con el fin de detectar la presencia/ausencia de parásitos de la malaria entre marzo y septiembre de 2008. Se examinaron frotis de sangre periférica y preparaciones en gota gruesa, posteriormente, por un máximo de cinco microscopistas expertos para la designación de especies de la malaria y la estimación de la parasitemia cuantitativa.
Las 22 muestras identificadas como positivas para P. ovale a través de microscopía, en las que todas las infecciones estaban mezcladas con otras especies de malaria, fueron designadas para la extracción de ADN y un análisis basado en la reacción en cadena de la polimerasa (PCR). El equipo utilizó la genotipificación multilocus para discriminar entre P. ovale curtisi y P. ovale wallikeri y todos los PCRs convencionales fueron realizados en el termociclador DNA Engine PTC-200 (MJ Research, Waltham, MA, EUA). La PCR en tiempo real fue realizada en la plataforma ABI 7500 rápida PCR en tiempo real (qPCR) (Life Technologies, Carlsbad, CA, EUA).
Las alineaciones de secuencias de genes de antígenos ricos en triptófano (tra) revelaron que nueve muestras (40,9%) dieron resultados positivos para P. ovale curtisi tipo 1, dos muestras (9,1%) dieron resultados positivos para P. ovale curtisi tipo 2, seis muestras (27,3%) fueron positivas para P. ovale wallikeri tipo 1, y tres muestras (13,6%) fueron positivas para P. ovale wallikeri tipo 2. Los estudios de especificidad mostraron la capacidad de los ensayos de qPCR para la proteína de unión a los reticulocitos 2 (rbp2) para detectar los niveles bajos de P. ovale en la presencia de otras especies de parásitos de la malaria, incluyendo P. falciparum, P. vivax y P. malariae. El equipo identificó P. ovale curtisi y P. ovale wallikeri en Kenia Occidental, mediante secuenciación de ADN del gen para el antígeno rico en triptófano, el gen para el ARN de la sub-unidad ribosómica pequeña, y el gen rbp2.
Los autores concluyeron que su análisis novedoso de qPCR para P. ovale rbp2 detecta P. ovale curtisi y P. ovale wallikeri, simultáneamente, y puede ser utilizado para caracterizar la prevalencia, distribución, y la carga de P. ovale en regiones endémicas de malaria. El uso de la genotipificación multilocus también proporcionó la primera descripción de la prevalencia de P. ovale curtisi y P. ovale wallikeri en Kenia occidental, una región holoendémica para la transmisión de la malaria. El estudio fue publicado el 15 de enero de 2015, en la revista Public Library of Science Neglected Tropical Diseases.
Enlaces relacionados:
Uniformed Services University
Zephyr Biomedicals
MJ Research
Life Technologies
Las diferencias en duración y latencia clínicas potenciales, entre P. ovale curtisi y P. ovale wallikeri demuestran la necesidad de investigación sobre la contribución de este parásito desatendido de la malaria a la carga mundial de la malaria.
Científicos de la Universidad de Servicios Uniformados (Bethesda, MD, EUA) quienes trabajaron con colegas en Kenia, recolectaron muestras de sangre total de individuos adultos asintomáticos clínicamente sanos, en la Provincia de Nyanza, Kenia. Las muestras fueron estudiadas inicialmente con la prueba de Diagnóstico Rápido Parascreen Pan/Pf malaria (Zephyr Biomedicals, Verna, Goa, India), con el fin de detectar la presencia/ausencia de parásitos de la malaria entre marzo y septiembre de 2008. Se examinaron frotis de sangre periférica y preparaciones en gota gruesa, posteriormente, por un máximo de cinco microscopistas expertos para la designación de especies de la malaria y la estimación de la parasitemia cuantitativa.
Las 22 muestras identificadas como positivas para P. ovale a través de microscopía, en las que todas las infecciones estaban mezcladas con otras especies de malaria, fueron designadas para la extracción de ADN y un análisis basado en la reacción en cadena de la polimerasa (PCR). El equipo utilizó la genotipificación multilocus para discriminar entre P. ovale curtisi y P. ovale wallikeri y todos los PCRs convencionales fueron realizados en el termociclador DNA Engine PTC-200 (MJ Research, Waltham, MA, EUA). La PCR en tiempo real fue realizada en la plataforma ABI 7500 rápida PCR en tiempo real (qPCR) (Life Technologies, Carlsbad, CA, EUA).
Las alineaciones de secuencias de genes de antígenos ricos en triptófano (tra) revelaron que nueve muestras (40,9%) dieron resultados positivos para P. ovale curtisi tipo 1, dos muestras (9,1%) dieron resultados positivos para P. ovale curtisi tipo 2, seis muestras (27,3%) fueron positivas para P. ovale wallikeri tipo 1, y tres muestras (13,6%) fueron positivas para P. ovale wallikeri tipo 2. Los estudios de especificidad mostraron la capacidad de los ensayos de qPCR para la proteína de unión a los reticulocitos 2 (rbp2) para detectar los niveles bajos de P. ovale en la presencia de otras especies de parásitos de la malaria, incluyendo P. falciparum, P. vivax y P. malariae. El equipo identificó P. ovale curtisi y P. ovale wallikeri en Kenia Occidental, mediante secuenciación de ADN del gen para el antígeno rico en triptófano, el gen para el ARN de la sub-unidad ribosómica pequeña, y el gen rbp2.
Los autores concluyeron que su análisis novedoso de qPCR para P. ovale rbp2 detecta P. ovale curtisi y P. ovale wallikeri, simultáneamente, y puede ser utilizado para caracterizar la prevalencia, distribución, y la carga de P. ovale en regiones endémicas de malaria. El uso de la genotipificación multilocus también proporcionó la primera descripción de la prevalencia de P. ovale curtisi y P. ovale wallikeri en Kenia occidental, una región holoendémica para la transmisión de la malaria. El estudio fue publicado el 15 de enero de 2015, en la revista Public Library of Science Neglected Tropical Diseases.
Enlaces relacionados:
Uniformed Services University
Zephyr Biomedicals
MJ Research
Life Technologies
Últimas Microbiología noticias
- Prueba de diagnóstico rápido reduce mortalidad por sepsis 39 %
- Análisis de hemocultivo mejora gestión diagnóstica mediante selección de panel específico
- Secuenciación genómica en tiempo real detecta superbacteria que causa infecciones hospitalarias
- Prueba diagnóstica detecta con precisión cáncer colorrectal al identificar firma microbiana en bacterias intestinales
- Prueba rápida junto al paciente predice sepsis con más de 90 % de precisión
- Nuevo análisis de sangre detecta hasta cinco enfermedades infecciosas en punto de atención
- Prueba molecular de heces muestra potencial para diagnosticar tuberculosis en adultos con VIH
- Nueva prueba diagnostica meningitis bacteriana con rapidez y precisión
- Dispositivo portátil ofrece resultados de tuberculosis económico y rápido
- Método basado en IA mejora diagnóstico de infecciones resistentes a fármacos
- Innovadora tecnología identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
Canales
Química Clínica
ver canal
Simple prueba cutánea podría revolucionar el diagnóstico de esofagitis eosinofílica pediátrica
La esofagitis eosinofílica (EE) es una enfermedad crónica, causada por alérgenos alimentarios, que se caracteriza por inflamación y daño del revestimiento esofágico.... Más
Herramienta de diagnóstico portátil utiliza bioluminiscencia para detectar virus POC
Los diagnósticos en el punto de atención se han convertido en herramientas cruciales en muchos hogares, permitiendo a las personas medir la glucemia, realizar pruebas de embarazo e incluso... Más
Prueba de madurez pulmonar con IA identifica recién nacidos con mayor riesgo de dificultad respiratoria
Cada año, aproximadamente 300.000 bebés nacen en Estados Unidos entre las 32 y 36 semanas de gestación, según datos nacionales de salud. Este grupo presenta un riesgo elevado de dificultad respiratoria,... MásDiagnóstico Molecular
ver canal
Microbios intestinales podrían permitir detección temprana y tratamiento del cáncer de páncreas
El cáncer de páncreas sigue siendo una de las enfermedades más graves y desafiantes en oncología debido a su dificultad para detectarlo y a sus limitadas opciones de tratamiento.... Más
Prueba altamente sensible y específica permite detección temprana del cáncer colorrectal
Una prueba diagnóstica altamente sensible y específica para la detección temprana y el tratamiento del cáncer colorrectal podría permitir el tratamiento en las etapas... MásHematología
ver canal
Prueba de cartucho desechable ofrece resultados de hemograma rápidos y precisos
El hemograma completo (HC) es una de las pruebas de laboratorio más solicitadas, crucial para diagnosticar enfermedades, monitorear terapias y realizar exámenes de salud rutinarios.... Más
Primera prueba de monitorización de heparina POC proporciona resultados rápidos
La dosificación de heparina requiere un manejo cuidadoso para evitar complicaciones hemorrágicas y de coagulación. En situaciones de alto riesgo, como la oxigenación por membrana... MásInmunología
ver canal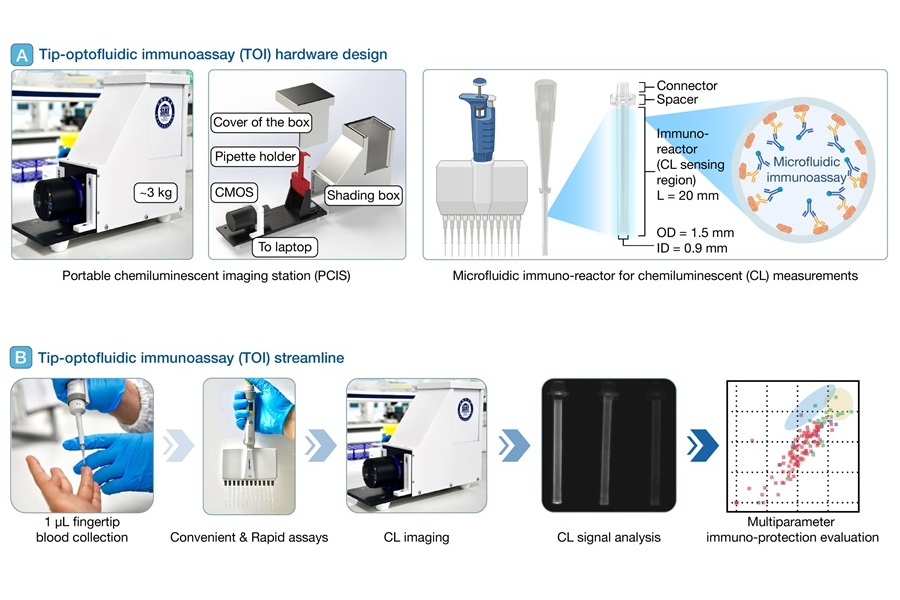
Plataforma de diagnóstico POC realiza análisis inmunológico con una gota de sangre del dedo
A medida que surgen nuevas variantes de la COVID-19 y las personas acumulan historiales complejos de vacunación e infección, existe una necesidad urgente de herramientas de diagnóstico... Más
Cambio de tratamiento guiado por biopsia líquida mejora resultados en pacientes con cáncer de mama
El tratamiento estándar para pacientes con cáncer de mama avanzado con receptor de estrógeno (RE) positivo y HER2 negativo, un subtipo impulsado por los receptores de estrógeno... Más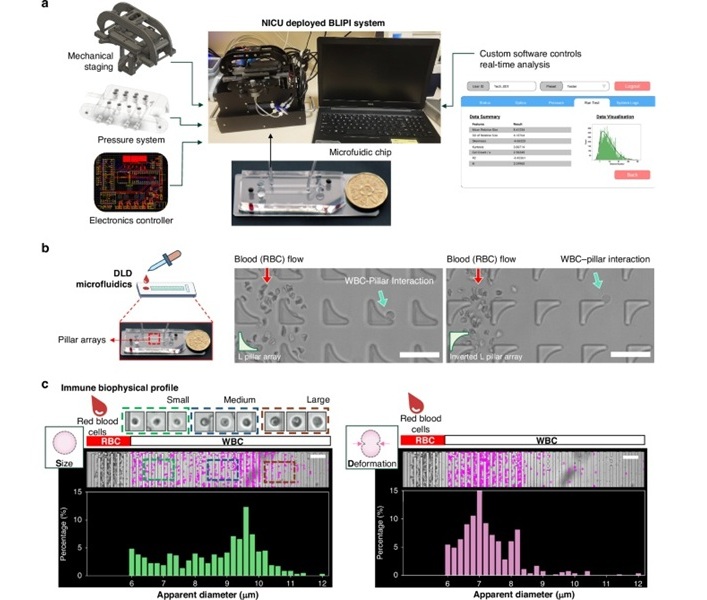
Dispositivo innovador analiza función inmunitaria de recién nacidos con una gota de sangre
Los bebés prematuros son muy susceptibles a enfermedades graves y potencialmente mortales, como la sepsis y la enterocolitis necrosante (ECN). La sepsis neonatal, una infección del torrente sanguíneo que... Más
Prueba de células madre predice resultado del tratamiento en cáncer de ovario resistente al platino
El cáncer de ovario epitelial suele responder inicialmente a la quimioterapia, pero con el tiempo, el tumor desarrolla resistencia a la terapia, lo que provoca su recrecimiento. Esta resistencia... MásPatología
ver canal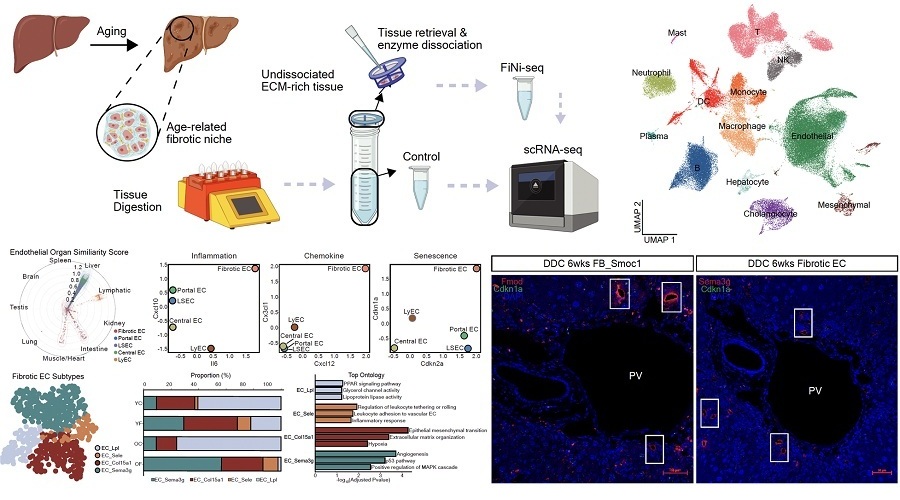
Nueva plataforma tecnológica predice enfermedades mediante detección temprana de señales de envejecimiento en tejido hepático
El envejecimiento y las enfermedades crónicas se caracterizan por la lenta acumulación de sutiles alteraciones tisulares durante largos periodos. Sin embargo, captar cuantitativamente estos... Más
Modelo de IA detecta más de 170 tipos de cáncer
El diagnóstico de tumores cerebrales puede ser especialmente difícil cuando el tumor se localiza en una región donde las biopsias presentan un alto riesgo. En estos casos, donde no... MásTecnología
ver canal
Encuesta revela que gestión mejorada de datos de laboratorio e IA son cruciales para laboratorios del futuro
Los datos desempeñan un papel fundamental en la transformación de los laboratorios digitales actuales, actuando como un desafío clave y como un catalizador para la innovación,... Más
Prueba no basada en ADN con IA identifica infecciones virales
La confirmación de infecciones virales suele implicar pruebas de secuenciación de ADN, que tardan horas y pueden no estar disponibles in situ. Además, estas pruebas no pueden diferenciar... MásIndustria
ver canal
Universidad de Aston y BG Research se asocian para comercializar diagnóstico médico innovador
La tecnología que puede diagnosticar rápidamente enfermedades infecciosas de alto impacto dará un gran paso hacia su comercialización gracias a una nueva asociación.... Más