Secuenciación genómica de infección por SARM predice gravedad de la enfermedad
|
Por el equipo editorial de LabMedica en español Actualizado el 04 Jun 2014 |

Imagen: El Analizador de Genoma IIx (Fotografía cortesía de Illumina).
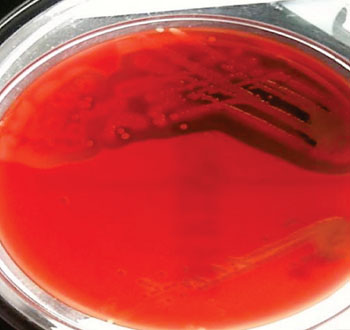
Imagen B: La cepa altamente tóxica de Staphylococcus aureus resistente a la meticilina (SARM) (arriba) y la cepa menos tóxica (abajo) cultivadas en placas de agar sangre (Fotografía cortesía de Ruth Massey).
Los patógenos bacterianos, como el Staphylococcus aureus resistente a la meticilina (SARM) causan, la enfermedad debido a la toxicidad o la capacidad de la bacteria de dañar el tejido del huésped.
La diseminación del patógeno resistente a los antibióticos sigue siendo una preocupación de salud pública, especialmente entre los médicos que están tratando de determinar las opciones de tratamiento apropiadas para los pacientes infectados.
Los microbiólogos de la Universidad de Bath (Reino Unido) y un equipo de científicos internacionales utilizaron secuencias de todo el genoma de 90 aislados de SARM para identificar más de 100 loci genéticos asociados con la toxicidad. Se evaluó la adhesión bacteriana a la fibronectina y el fibrinógeno humano y se calcularon las bacterias adherentes utilizando el método de cristal violeta y la absorbancia medida a A595 usando un lector de placas de microtitulación. La toxicidad de los aislados individuales fue analizada de tres formas.
Se estudió la identificación de la variación genética en los aislados clínicos utilizando bibliotecas únicas de índice de etiquetado creadas para cada muestra y se secuenciaron hasta 12 bibliotecas independientes en cada uno de los ocho canales en las células del analizador Genome Analyzer GAIIx (Illumina; San Diego, CA, EUA) con 75 lecturas de pares de bases finales.
Los autores encontraron que mediante el uso de las secuencias de todo el genoma a partir de 90 aislados de SARM fueron capaces de identificar más de 100 loci genéticos asociados a la toxicidad y, a pesar de pertenecer al mismo clon ST239, los aislados variaron en mucho con respecto a la toxicidad. Es importante destacar que los aislados altamente tóxicos compartieron una firma genética común. Mediante el estudio de esta firma en el genoma del SARM, los investigadores fueron capaces de predecir cuáles aislados eran los más tóxicos y por lo tanto más propensos a causar una enfermedad grave cuando se utilizaron para infectar a los ratones.
Ruth C. Massey, PhD, autora principal del estudio, dijo: “A medida que el costo y la velocidad de la secuenciación del genoma disminuye, se hace cada vez más factible secuenciar el genoma de un organismo infeccioso. En un entorno clínico, la secuenciación puede ser útil para decidir el tipo de tratamiento para el SARM. Por ejemplo, un médico puede tratar una infección altamente tóxica más agresivamente, incluyendo la formulación de ciertos antibióticos conocidos para reducir la expresión de la toxina. El paciente también puede recibir un seguimiento más de cerca en busca de las complicaciones y aislarlo de los demás para ayudar a controlar la propagación de la infección”. El estudio fue publicado el 9 de abril de 2014, en la revista Genome Research.
Enlaces relacionados:
University of Bath
Illumina
La diseminación del patógeno resistente a los antibióticos sigue siendo una preocupación de salud pública, especialmente entre los médicos que están tratando de determinar las opciones de tratamiento apropiadas para los pacientes infectados.
Los microbiólogos de la Universidad de Bath (Reino Unido) y un equipo de científicos internacionales utilizaron secuencias de todo el genoma de 90 aislados de SARM para identificar más de 100 loci genéticos asociados con la toxicidad. Se evaluó la adhesión bacteriana a la fibronectina y el fibrinógeno humano y se calcularon las bacterias adherentes utilizando el método de cristal violeta y la absorbancia medida a A595 usando un lector de placas de microtitulación. La toxicidad de los aislados individuales fue analizada de tres formas.
Se estudió la identificación de la variación genética en los aislados clínicos utilizando bibliotecas únicas de índice de etiquetado creadas para cada muestra y se secuenciaron hasta 12 bibliotecas independientes en cada uno de los ocho canales en las células del analizador Genome Analyzer GAIIx (Illumina; San Diego, CA, EUA) con 75 lecturas de pares de bases finales.
Los autores encontraron que mediante el uso de las secuencias de todo el genoma a partir de 90 aislados de SARM fueron capaces de identificar más de 100 loci genéticos asociados a la toxicidad y, a pesar de pertenecer al mismo clon ST239, los aislados variaron en mucho con respecto a la toxicidad. Es importante destacar que los aislados altamente tóxicos compartieron una firma genética común. Mediante el estudio de esta firma en el genoma del SARM, los investigadores fueron capaces de predecir cuáles aislados eran los más tóxicos y por lo tanto más propensos a causar una enfermedad grave cuando se utilizaron para infectar a los ratones.
Ruth C. Massey, PhD, autora principal del estudio, dijo: “A medida que el costo y la velocidad de la secuenciación del genoma disminuye, se hace cada vez más factible secuenciar el genoma de un organismo infeccioso. En un entorno clínico, la secuenciación puede ser útil para decidir el tipo de tratamiento para el SARM. Por ejemplo, un médico puede tratar una infección altamente tóxica más agresivamente, incluyendo la formulación de ciertos antibióticos conocidos para reducir la expresión de la toxina. El paciente también puede recibir un seguimiento más de cerca en busca de las complicaciones y aislarlo de los demás para ayudar a controlar la propagación de la infección”. El estudio fue publicado el 9 de abril de 2014, en la revista Genome Research.
Enlaces relacionados:
University of Bath
Illumina
Últimas Microbiología noticias
- Innovadora tecnología disgnóstica identifica infecciones bacterianas con precisión de casi 100 % en tres horas
- Sistema de identificación y PSA ayuda a diagnosticar enfermedades infecciosas y combatir RAM
- Panel gastrointestinal permite detección rápida de cinco patógenos bacterianos comunes
- Pruebas rápidas PCR en UCI mejoran uso de antibióticos
- Firma genética única predice resistencia a fármacos en bacterias
- Sistema de código de barras rastrea bacterias de neumonía mientras infectan el torrente sanguíneo
- Prueba rápida de diagnóstico de sepsis demuestra mejor atención al paciente y ahorro en aplicaciones hospitalarias
- Sistema de diagnóstico rápido detecta sepsis neonatal en horas
- Nueva prueba diagnostica neumonía bacteriana directamente a partir de sangre completa
- Ensayo de liberación de interferón-γ es eficaz en pacientes con EPOC y tuberculosis pulmonar
- Nuevas pruebas en punto de atención ayudan a reducir uso excesivo de antibióticos
- Prueba de sepsis rápida permite diferenciar infecciones bacterianas, virales y enfermedades no infecciosas
- Prueba CRISPR-TB permite diagnóstico temprano de enfermedad y cribado de la población
- Panel sindrómico ofrece respuestas rápidas para diagnóstico ambulatorio de enfermedades gastrointestinales
- Plataforma sin cultivo identifica rápidamente infecciones del torrente sanguíneo
- Prueba de PCR POC detecta rápidamente meningitis bacteriana directamente en punto de recolección de muestra
Canales
Química Clínica
ver canal
Nanotubos de carbono ayudan a construir sensores precisos para monitoreo continuo de la salud
Los sensores actuales pueden medir diversos indicadores de salud, como los niveles de glucosa en sangre. Sin embargo, es necesario desarrollar materiales para sensores más precisos y sensibles que... Más
Dispositivo basado en papel mejora la precisión de prueba del VIH
En las regiones donde el acceso a las clínicas para realizar análisis de sangre rutinarios presenta obstáculos financieros y logísticos, los pacientes con VIH pueden recolectar... MásDiagnóstico Molecular
ver canal
Análisis de sangre identifica múltiples biomarcadores para diagnóstico rápido de lesiones de médula espinal
Los Institutos Nacionales de Salud estiman que 18.000 personas en Estados Unidos sufren lesiones de la médula espinal (LME) anualmente, lo que resulta en una asombrosa carga financiera de más de 9.... Más
Análisis de sangre muy preciso diagnostica Alzheimer y mide progresión de demencia
Actualmente existen varios análisis de sangre que ayudan a los médicos a diagnosticar la enfermedad de Alzheimer en personas con síntomas cognitivos. Sin embargo, estas pruebas no... MásHematología
ver canal
Nuevo sistema de puntuación predice riesgo de cáncer a partir de un trastorno sanguíneo común
La citopenia clonal de significado incierto (CCSI) es un trastorno sanguíneo común en adultos mayores, caracterizado por mutaciones en las células sanguíneas y un recuento ... Más
Prueba prenatal no invasiva para determinar estado RhD del feto es 100 % precisa
En los Estados Unidos, aproximadamente el 15 % de las embarazadas son RhD negativas. Sin embargo, en aproximadamente el 40 % de estos casos, el feto también es RhD negativo, lo que hace innecesaria la... MásInmunología
ver canal
Análisis de sangre podría orientar decisiones futuras sobre tratamiento del cáncer
En el continuo avance de la medicina personalizada, un nuevo estudio ha aportado evidencia que respalda el uso de una herramienta que detecta moléculas derivadas del cáncer en la sangre de... Más
Prueba de líquido cefalorraquídeo predice efecto secundario peligroso del tratamiento del cáncer
En los últimos años, la inmunoterapia contra el cáncer se ha convertido en un enfoque prometedor que aprovecha el sistema inmunitario del paciente para combatir el cáncer.... MásPatología
ver canal
Modelo de IA predice respuesta al tratamiento del cáncer de vejiga
Cada año en Estados Unidos, se diagnostican alrededor de 81.000 nuevos casos de cáncer de vejiga, lo que provoca aproximadamente 17.000 muertes al año. El cáncer de vejiga ... Más
Nuevo método basado en láser acelera diagnóstico del cáncer
Investigadores han desarrollado un método para mejorar el diagnóstico del cáncer y otras enfermedades. El colágeno, una proteína estructural clave, desempeña diversas funciones en la actividad celular.... Más
Nuevo modelo de IA predice efectos de variantes genéticas en enfermedades específicas
En los últimos años, la inteligencia artificial (IA) ha mejorado considerablemente nuestra capacidad para identificar un gran número de variantes genéticas en poblaciones cada... Más
Herramienta de IA diagnostica enfermedad celíaca en imágenes de biopsia con precisión superior al 97%
La enfermedad celíaca es un trastorno autoinmune desencadenado por el consumo de gluten, que causa síntomas como calambres estomacales, diarrea, erupciones cutáneas, pérdida de peso, fatiga y anemia.... MásTecnología
ver canal
Teléfonos inteligentes podrían diagnosticar enfermedades mediante escáneres infrarrojos
Los rápidos avances tecnológicos pronto permitirán que las personas eviten procedimientos médicos invasivos simplemente subiendo una captura de pantalla de sus resultados de... Más
Nueva tecnología de sensores permite diagnóstico temprano de trastornos metabólicos y cardiovasculares
Los metabolitos son compuestos cruciales que impulsan las funciones vitales, desempeñando un papel clave en la producción de energía, la regulación de la actividad celular y... MásIndustria
ver canalLeica Biosystems y Bio-Techne amplían su colaboración multiómica espacial
Bio-Techne Corporation (Minneapolis, MN, EUA) ha ampliado la larga colaboración entre su marca de biología espacial, Advanced Cell Diagnostics (ACD, Newark, CA, EUA), y Leica Biosystems (Nussloch,... Más