Secuenciación genómica de infección por SARM predice gravedad de la enfermedad
|
Por el equipo editorial de LabMedica en español Actualizado el 04 Jun 2014 |

Imagen: El Analizador de Genoma IIx (Fotografía cortesía de Illumina).
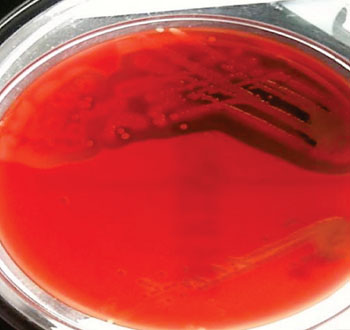
Imagen B: La cepa altamente tóxica de Staphylococcus aureus resistente a la meticilina (SARM) (arriba) y la cepa menos tóxica (abajo) cultivadas en placas de agar sangre (Fotografía cortesía de Ruth Massey).
Los patógenos bacterianos, como el Staphylococcus aureus resistente a la meticilina (SARM) causan, la enfermedad debido a la toxicidad o la capacidad de la bacteria de dañar el tejido del huésped.
La diseminación del patógeno resistente a los antibióticos sigue siendo una preocupación de salud pública, especialmente entre los médicos que están tratando de determinar las opciones de tratamiento apropiadas para los pacientes infectados.
Los microbiólogos de la Universidad de Bath (Reino Unido) y un equipo de científicos internacionales utilizaron secuencias de todo el genoma de 90 aislados de SARM para identificar más de 100 loci genéticos asociados con la toxicidad. Se evaluó la adhesión bacteriana a la fibronectina y el fibrinógeno humano y se calcularon las bacterias adherentes utilizando el método de cristal violeta y la absorbancia medida a A595 usando un lector de placas de microtitulación. La toxicidad de los aislados individuales fue analizada de tres formas.
Se estudió la identificación de la variación genética en los aislados clínicos utilizando bibliotecas únicas de índice de etiquetado creadas para cada muestra y se secuenciaron hasta 12 bibliotecas independientes en cada uno de los ocho canales en las células del analizador Genome Analyzer GAIIx (Illumina; San Diego, CA, EUA) con 75 lecturas de pares de bases finales.
Los autores encontraron que mediante el uso de las secuencias de todo el genoma a partir de 90 aislados de SARM fueron capaces de identificar más de 100 loci genéticos asociados a la toxicidad y, a pesar de pertenecer al mismo clon ST239, los aislados variaron en mucho con respecto a la toxicidad. Es importante destacar que los aislados altamente tóxicos compartieron una firma genética común. Mediante el estudio de esta firma en el genoma del SARM, los investigadores fueron capaces de predecir cuáles aislados eran los más tóxicos y por lo tanto más propensos a causar una enfermedad grave cuando se utilizaron para infectar a los ratones.
Ruth C. Massey, PhD, autora principal del estudio, dijo: “A medida que el costo y la velocidad de la secuenciación del genoma disminuye, se hace cada vez más factible secuenciar el genoma de un organismo infeccioso. En un entorno clínico, la secuenciación puede ser útil para decidir el tipo de tratamiento para el SARM. Por ejemplo, un médico puede tratar una infección altamente tóxica más agresivamente, incluyendo la formulación de ciertos antibióticos conocidos para reducir la expresión de la toxina. El paciente también puede recibir un seguimiento más de cerca en busca de las complicaciones y aislarlo de los demás para ayudar a controlar la propagación de la infección”. El estudio fue publicado el 9 de abril de 2014, en la revista Genome Research.
Enlaces relacionados:
University of Bath
Illumina
La diseminación del patógeno resistente a los antibióticos sigue siendo una preocupación de salud pública, especialmente entre los médicos que están tratando de determinar las opciones de tratamiento apropiadas para los pacientes infectados.
Los microbiólogos de la Universidad de Bath (Reino Unido) y un equipo de científicos internacionales utilizaron secuencias de todo el genoma de 90 aislados de SARM para identificar más de 100 loci genéticos asociados con la toxicidad. Se evaluó la adhesión bacteriana a la fibronectina y el fibrinógeno humano y se calcularon las bacterias adherentes utilizando el método de cristal violeta y la absorbancia medida a A595 usando un lector de placas de microtitulación. La toxicidad de los aislados individuales fue analizada de tres formas.
Se estudió la identificación de la variación genética en los aislados clínicos utilizando bibliotecas únicas de índice de etiquetado creadas para cada muestra y se secuenciaron hasta 12 bibliotecas independientes en cada uno de los ocho canales en las células del analizador Genome Analyzer GAIIx (Illumina; San Diego, CA, EUA) con 75 lecturas de pares de bases finales.
Los autores encontraron que mediante el uso de las secuencias de todo el genoma a partir de 90 aislados de SARM fueron capaces de identificar más de 100 loci genéticos asociados a la toxicidad y, a pesar de pertenecer al mismo clon ST239, los aislados variaron en mucho con respecto a la toxicidad. Es importante destacar que los aislados altamente tóxicos compartieron una firma genética común. Mediante el estudio de esta firma en el genoma del SARM, los investigadores fueron capaces de predecir cuáles aislados eran los más tóxicos y por lo tanto más propensos a causar una enfermedad grave cuando se utilizaron para infectar a los ratones.
Ruth C. Massey, PhD, autora principal del estudio, dijo: “A medida que el costo y la velocidad de la secuenciación del genoma disminuye, se hace cada vez más factible secuenciar el genoma de un organismo infeccioso. En un entorno clínico, la secuenciación puede ser útil para decidir el tipo de tratamiento para el SARM. Por ejemplo, un médico puede tratar una infección altamente tóxica más agresivamente, incluyendo la formulación de ciertos antibióticos conocidos para reducir la expresión de la toxina. El paciente también puede recibir un seguimiento más de cerca en busca de las complicaciones y aislarlo de los demás para ayudar a controlar la propagación de la infección”. El estudio fue publicado el 9 de abril de 2014, en la revista Genome Research.
Enlaces relacionados:
University of Bath
Illumina
Últimas Microbiología noticias
- Virus intestinales ocultos vinculados al riesgo de cáncer colorrectal
- Panel de tres pruebas para detección de infecciones por duelas del hígado
- Prueba rápida promete respuestas inmediatas a infecciones resistentes a medicamentos
- Tecnología con CRISPR neutraliza bacterias resistentes a antibióticos
- Nuevo estudio identifica características del microbioma intestinal asociadas con enfermedad de Alzheimer
- Plataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- Método de diagnóstico en sangre identifica infecciones IVRI pediátricas
- Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
Canales
Química Clínica
ver canalNuevo índice sanguíneo permite la detección más temprana de la fibrosis hepática
La enfermedad del hígado graso metabólico es muy prevalente y a menudo asintomática; sin embargo, puede progresar a fibrosis, cirrosis e insuficiencia hepática.... Más
Nariz electrónica detecta signos tempranos de cáncer de ovario en sangre
El cáncer de ovario suele diagnosticarse en una etapa tardía debido a que sus síntomas son vagos y similares a los de afecciones más comunes. A diferencia del cáncer... MásDiagnóstico Molecular
ver canal
Prueba con hisopo ayuda a pacientes trasplantados a recibir la dosis correcta de medicación antirrechazo
El tacrolimus se utiliza ampliamente para prevenir el rechazo de órganos en receptores de trasplante, pero alcanzar la dosis correcta desde el inicio es fundamental. Si los niveles son demasiado... Más
Análisis de sangre predice qué pacientes con cáncer de vejiga pueden evitar la cirugía
El cáncer de vejiga músculo-invasivo (CVMI) a menudo requiere la extirpación de la vejiga, un procedimiento que afecta significativamente la calidad de vida y conlleva riesgo de complicaciones.... MásHematología
ver canal
Prueba rápida en cartuchos busca ampliar acceso al diagnóstico de trastornos de hemoglobina
La anemia de células falciformes y la beta talasemia son trastornos de la hemoglobina que a menudo requieren la derivación a laboratorios especializados para un diagnóstico definitivo,... Más
Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
La amiloidosis de cadenas ligeras (AL) es un trastorno poco común y potencialmente mortal de la médula ósea, en el que las proteínas amiloides anormales se acumulan en los órganos. Aproximadamente 3.... MásInmunología
ver canal
Nuevo biomarcador predice respuesta a quimioterapia en cáncer de mama triple negativo
El cáncer de mama triple negativo es una forma agresiva de cáncer de mama en la que las pacientes suelen mostrar respuestas muy variables a la quimioterapia. Predecir quién se ben... MásAnálisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
El cáncer de pulmón de células pequeñas (CPCP) es una enfermedad agresiva con opciones de tratamiento limitadas, e incluso las inmunoterapias recientemente aprobadas no benefician... Más
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... MásPatología
ver canal
Imágenes moleculares podrían reducir la necesidad de biopsias de melanoma
El melanoma es el tipo más mortal de cáncer de piel y causa la gran mayoría de las muertes relacionadas con esta enfermedad. Dado que los melanomas en sus etapas iniciales pueden parecerse... Más
Sistema de recolección de orina mejora precisión y eficiencia del diagnóstico
El análisis de orina es una herramienta diagnóstica crucial y no invasiva que se utiliza para detectar afecciones como embarazo, infecciones del tracto urinario, trastornos metabólicos,... MásTecnología
ver canal
“Relojes” de análisis sanguíneos predicen inicio de síntomas del Alzheimer
Más de 7 millones de estadounidenses viven con la enfermedad de Alzheimer, y se proyecta que los costos de salud y atención a largo plazo relacionados alcancen casi 400 mil millones de dólares... Más
Biomarcador de IA predice riesgo de cáncer de hígado
El cáncer de hígado, o carcinoma hepatocelular, causa más de 800.000 muertes al año en todo el mundo y, a menudo, no se detecta hasta las últimas etapas.... MásIndustria
ver canal
Cepheid se une a iniciativa del CDC para fortalecer pruebas detección de pandemias
Cepheid (Sunnyvale, CA, EUA) ha sido seleccionado por los Centros para el Control y la Prevención de Enfermedades (CDC) de EUA como uno de los cuatro colaboradores nacionales en una iniciativa federal... Más