Secuenciación metagenómica identifica rápidamente los patógenos en los fluidos corporales
|
Por el equipo editorial de LabMedica en español Actualizado el 25 Nov 2020 |

Imagen: La plataforma de secuenciación portátil MinION se procesa en una celda de flujo y el GridION de escritorio puede procesar hasta cinco celdas de flujo en cualquier momento (Fotografía cortesía de Oxford Nanopore).
La secuenciación metagenómica de próxima generación (mNGS) es un enfoque de secuenciación tipo escopeta en el que todo el ácido nucleico (ADN y ARN) de una muestra clínica se secuencia a una profundidad muy alta, de 10 a 20 millones de secuencias por muestra.
Ser capaz de determinar rápidamente la causa de las infecciones de los pacientes puede informar a los médicos y orientar el tipo de tratamiento además de guiar la selección de antibióticos. La identificación rápida y exacta de patógenos no siempre es posible en la clínica, ya que los cultivos tardan en crecer y las pruebas de reacción en cadena de la polimerasa (PCR) requieren una idea de qué microbio podría ser la fuente de la infección.
Un equipo de científicos médicos de laboratorio de la Universidad de California, San Francisco (San Francisco, CA, EUA), recolectó una variedad de muestras de fluidos corporales, como abscesos, fluidos pleurales y cerebroespinales de 158 pacientes, la mayoría de los cuales estaban hospitalizados. De estas, 127 muestras fueron positivas para un patógeno por cultivo, nueve fueron cultivo-negativos, pero PCR-positivas y 34 fueron controles negativos. Luego, los investigadores aplicaron el protocolo de prueba de mNGS, que desarrollaron, a esas muestras. Este protocolo, señalaron, es compatible con las plataformas de secuenciación de Oxford Nanopore (Oxford, Reino Unido) y de Illumina (San Diego, CA, EUA), que puede analizar todo los tipos de fluidos corporales y se puede automatizar en laboratorios de microbiología clínica. Las lecturas generadas se analizan mediante el software de identificación ultrarrápida de patógenos (SURPI) basado en secuencias para determinar qué patógeno está presente, si es que había alguno.
El equipo evaluó las dos plataformas de secuenciación en comparación con las pruebas microbiológicas utilizando cultivo, PCR bacteriana 16S y/o PCR fúngica con espaciador genético ribosomal transcrito internamente 28S (28S-ITS). Determinaron que su método podía detectar bacterias con un 79,2% y un 90,6% de especificidad mediante la secuenciación de Illumina, y con un 75% de sensibilidad y un 81,4% de especificidad mediante un método de secuenciación de nanoporos. El desempeño de la prueba varió ligeramente según el tipo de muestra, con la mayor exactitud derivada de las muestras de LCR. Además, el equipo notó que la secuenciación de nanoporos comenzó a dar resultados en tan solo 50 minutos y tuvo un tiempo promedio de respuesta de muestra a respuesta de aproximadamente seis horas. Mientras tanto, la secuenciación de Illumina tuvo un tiempo de respuesta promedio de aproximadamente 24 horas.
El método también podría detectar patógenos fúngicos, con una sensibilidad del 91% y una especificidad del 89% utilizando la secuenciación de Illumina y con una sensibilidad del 91% y el 100% mediante la secuenciación de nanoporos. Además, en una serie de casos de una docena de pacientes cuyas muestras resultaron negativas en cultivo y PCR, pero que finalmente se determinó que tenían una infección, los científicos encontraron que siete dieron positivo mediante mNGS. Sólo uno de los controles negativos fue un falso positivo para un patógeno bacteriano por mNGS, y de los casos falsos negativos, Staphylococcus aureus fue la bacteria que más comúnmente se pasó por alto. El equipo sugirió que la menor sensibilidad en la detección de S. aureus, especialmente mediante la secuenciación de nanoporos, se podría deber a niveles más altos de ADN de fondo del huésped humano.
Los autores concluyeron que habían demostrado la utilidad de la mNGS para ampliar el alcance de las pruebas de diagnóstico convencionales a múltiples tipos de fluidos corporales. El tiempo de respuesta alcanzable, de menos de seis horas mediante la secuenciación de nanoporos, también puede ser esencial para infecciones como la sepsis y la neumonía que exigen una respuesta rápida y un diagnóstico oportuno. El estudio fue publicado el 9 de noviembre de 2020 en la revista Nature Medicine.
Enlace relacionado:
Universidad de California, San Francisco
Oxford Nanopore
Ser capaz de determinar rápidamente la causa de las infecciones de los pacientes puede informar a los médicos y orientar el tipo de tratamiento además de guiar la selección de antibióticos. La identificación rápida y exacta de patógenos no siempre es posible en la clínica, ya que los cultivos tardan en crecer y las pruebas de reacción en cadena de la polimerasa (PCR) requieren una idea de qué microbio podría ser la fuente de la infección.
Un equipo de científicos médicos de laboratorio de la Universidad de California, San Francisco (San Francisco, CA, EUA), recolectó una variedad de muestras de fluidos corporales, como abscesos, fluidos pleurales y cerebroespinales de 158 pacientes, la mayoría de los cuales estaban hospitalizados. De estas, 127 muestras fueron positivas para un patógeno por cultivo, nueve fueron cultivo-negativos, pero PCR-positivas y 34 fueron controles negativos. Luego, los investigadores aplicaron el protocolo de prueba de mNGS, que desarrollaron, a esas muestras. Este protocolo, señalaron, es compatible con las plataformas de secuenciación de Oxford Nanopore (Oxford, Reino Unido) y de Illumina (San Diego, CA, EUA), que puede analizar todo los tipos de fluidos corporales y se puede automatizar en laboratorios de microbiología clínica. Las lecturas generadas se analizan mediante el software de identificación ultrarrápida de patógenos (SURPI) basado en secuencias para determinar qué patógeno está presente, si es que había alguno.
El equipo evaluó las dos plataformas de secuenciación en comparación con las pruebas microbiológicas utilizando cultivo, PCR bacteriana 16S y/o PCR fúngica con espaciador genético ribosomal transcrito internamente 28S (28S-ITS). Determinaron que su método podía detectar bacterias con un 79,2% y un 90,6% de especificidad mediante la secuenciación de Illumina, y con un 75% de sensibilidad y un 81,4% de especificidad mediante un método de secuenciación de nanoporos. El desempeño de la prueba varió ligeramente según el tipo de muestra, con la mayor exactitud derivada de las muestras de LCR. Además, el equipo notó que la secuenciación de nanoporos comenzó a dar resultados en tan solo 50 minutos y tuvo un tiempo promedio de respuesta de muestra a respuesta de aproximadamente seis horas. Mientras tanto, la secuenciación de Illumina tuvo un tiempo de respuesta promedio de aproximadamente 24 horas.
El método también podría detectar patógenos fúngicos, con una sensibilidad del 91% y una especificidad del 89% utilizando la secuenciación de Illumina y con una sensibilidad del 91% y el 100% mediante la secuenciación de nanoporos. Además, en una serie de casos de una docena de pacientes cuyas muestras resultaron negativas en cultivo y PCR, pero que finalmente se determinó que tenían una infección, los científicos encontraron que siete dieron positivo mediante mNGS. Sólo uno de los controles negativos fue un falso positivo para un patógeno bacteriano por mNGS, y de los casos falsos negativos, Staphylococcus aureus fue la bacteria que más comúnmente se pasó por alto. El equipo sugirió que la menor sensibilidad en la detección de S. aureus, especialmente mediante la secuenciación de nanoporos, se podría deber a niveles más altos de ADN de fondo del huésped humano.
Los autores concluyeron que habían demostrado la utilidad de la mNGS para ampliar el alcance de las pruebas de diagnóstico convencionales a múltiples tipos de fluidos corporales. El tiempo de respuesta alcanzable, de menos de seis horas mediante la secuenciación de nanoporos, también puede ser esencial para infecciones como la sepsis y la neumonía que exigen una respuesta rápida y un diagnóstico oportuno. El estudio fue publicado el 9 de noviembre de 2020 en la revista Nature Medicine.
Enlace relacionado:
Universidad de California, San Francisco
Oxford Nanopore
Últimas Microbiología noticias
- Plataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- Método de diagnóstico en sangre identifica infecciones IVRI pediátricas
- Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Prueba rápida no invasiva utiliza huella de azúcar para detectar infecciones por hongos
- Dispositivo de diagnóstico rápido de sepsis para atención crítica personalizada a pacientes de UCI
- Plataforma microfluídica evalúa función de neutrófilos en pacientes con sepsis
- Nuevo método diagnóstico confirma sepsis de forma más temprana
Canales
Química Clínica
ver canal
Modelo pronóstico basado en APE mejora evaluación del riesgo de cáncer
El cáncer de próstata es la segunda causa principal de muerte por cáncer en los hombres estadounidenses, y aproximadamente uno de cada ocho será diagnosticado a lo largo de su vida.... Más
Vesículas extracelulares se relacionan con riesgo de insuficiencia cardíaca en pacientes con ERC
La enfermedad renal crónica (ERC) afecta a más de 1 de cada 7 estadounidenses y está estrechamente asociada con complicaciones cardiovasculares, las cuales representan más de... MásDiagnóstico Molecular
ver canal
Biopsia líquida podría reemplazar a biopsia quirúrgica para diagnosticar linfoma nervioso central primario
El linfoma primario del sistema nervioso central (LPSNC) se diagnostica habitualmente mediante biopsia quirúrgica, que sigue siendo el método de referencia, pero conlleva un riesgo considerable.... Más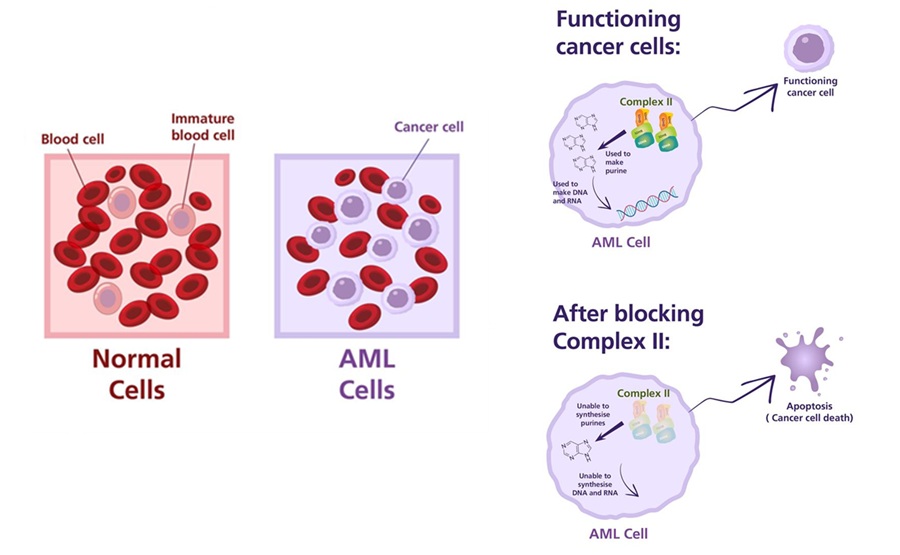
Herramienta revela debilidad metabólica oculta en cánceres de sangre
La leucemia mieloide aguda (LMA) es uno de los cánceres de sangre más agresivos, caracterizado por bajas tasas de supervivencia y limitadas opciones de tratamiento, especialmente en pacientes que no responden... MásHematología
ver canal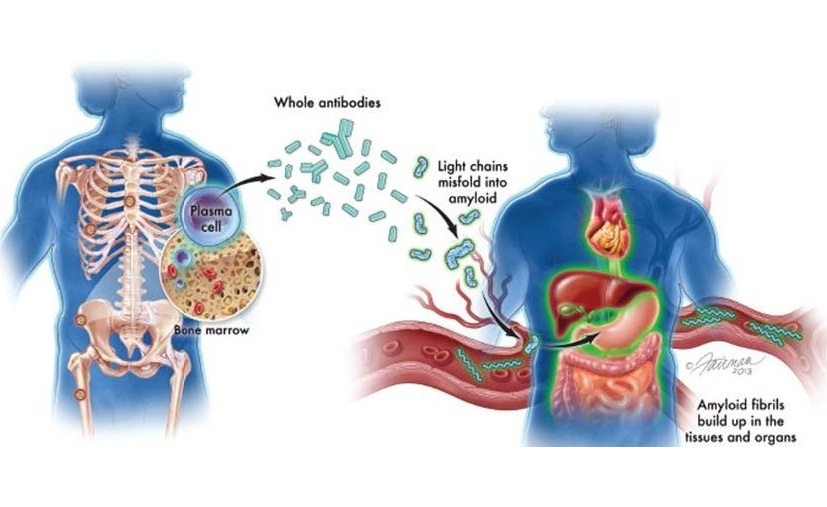
Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
La amiloidosis de cadenas ligeras (AL) es un trastorno poco común y potencialmente mortal de la médula ósea, en el que las proteínas amiloides anormales se acumulan en los órganos. Aproximadamente 3.... Más
Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
Las secciones de hemostasia de alto volumen requieren una rápida respuesta, a la vez que gestionan repeticiones y pruebas reflejas. La manipulación de los tubos y las comprobaciones prea... MásInmunología
ver canalAnálisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
El cáncer de pulmón de células pequeñas (CPCP) es una enfermedad agresiva con opciones de tratamiento limitadas, e incluso las inmunoterapias recientemente aprobadas no benefician... Más
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... MásPatología
ver canal
IA mejora detección de mutaciones genéticas en diagnóstico del cáncer
La identificación precisa de mutaciones genéticas es fundamental para el diagnóstico del cáncer y la investigación genómica, pero los métodos actuales presentan... Más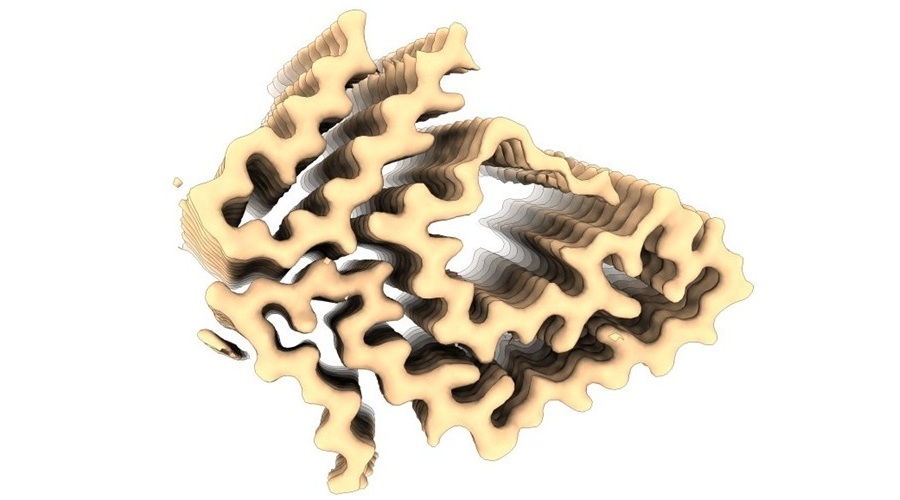
Biopsia de piel ofrece nuevo método diagnóstico para enfermedades neurodegenerativas
La amiloidosis por transtiretina (ATTR) es una enfermedad rara, progresiva y muy agresiva causada por el plegamiento incorrecto de una proteína específica que se acumula en forma de filamentos... MásTecnología
ver canal
ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
Los laboratorios clínicos generan miles de millones de resultados de pruebas cada año, lo que crea un cúmulo de datos con el potencial de facilitar pruebas más personalizadas,... MásTecnología de biosensores de aptámeros transforma detección de virus
La detección rápida y fiable de virus es esencial para controlar brotes, desde la gripe estacional hasta pandemias globales como la de COVID-19. Los métodos de diagnóstico ... MásIndustria
ver canal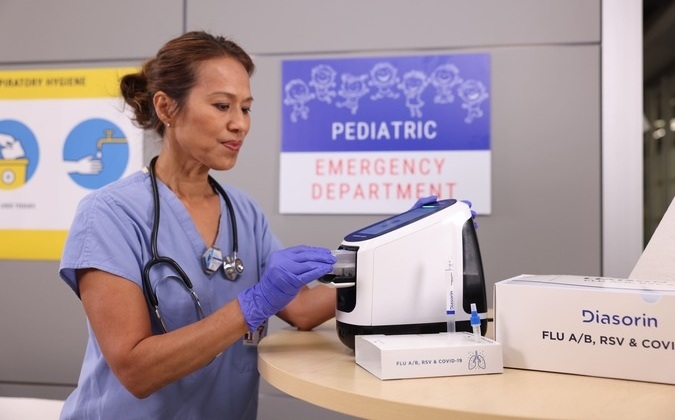
Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
Diasorin (Saluggia, Italia) ha firmado un acuerdo de distribución exclusivo con Fisher Scientific, parte de Thermo Fisher Scientific (Waltham, MA, EUA), para la plataforma molecular de punto de... Más










 (3) (1).png)