Bacterias como supermicrobios resistentes a los antibióticos
|
Por el equipo editorial de LabMedica en español Actualizado el 23 Apr 2015 |
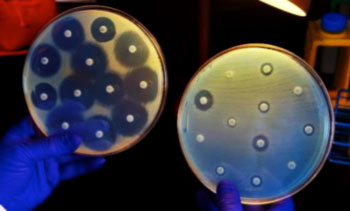
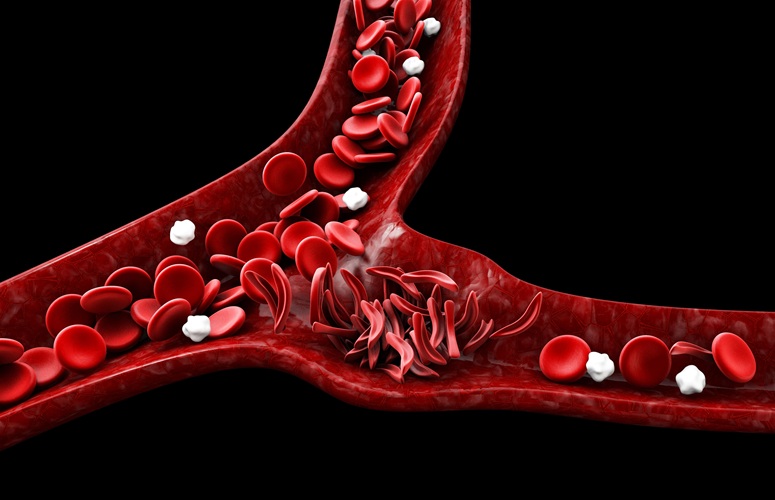
Imagen: La placa de la derecha fue inoculada con una Enterobacteria resistente al Carbapenem (CRE) que resultó ser resistente a todos los antibióticos ensayados; las bacterias en la placa izquierda son susceptibles a los antibióticos en los discos (Fotografía cortesía de James Gathany).
Las enterobacterias patógenas, incluyendo Escherichia coli y Klebsiella pneumoniae, son las principales causas de infecciones resistente a múltiples antibióticos (MDR) en los hospitales de todo el mundo y éstos patógenos han demostrado recientemente que han adquirido resistencia a los carbapenémicos.
Se han identificado dos genes que confieren resistencia contra una clase particularmente fuerte de antibióticos y que pueden ser compartidos fácilmente entre una familia de bacterias responsables de una parte importante de infecciones respiratorias y urinarias adquiridas en los hospitales.
Los científicos de la Facultad de Medicina de la Universidad de Washington (St. Louis, MO, EUA) recolectaron 450 aislados bacterianos, incluyendo 195 Enterobacterias en Pakistán, durante febrero de 2012 hasta marzo de 2013. Los investigadores seleccionaron al azar 55 aislados de Enterobacterias para hacerles la secuenciación de todo el genoma. Luego seleccionaron 23 aislamientos de muestras obtenidas de pacientes en los EUA entre enero de 2010 y junio de 2013, en el Hospital Judío Barnes (St. Louis, MO, EUA), que tenían una proporción similar de susceptibilidad y resistencia a los β-lactámicos a los recolectados en Pakistán para la secuenciación.
Los investigadores también secuenciaron una porción especial de material genético de las bacterias, llamados plásmidos. La mayor parte del ADN de una bacteria se encuentra en su cromosoma, pero las bacterias también tienen muchos pedazos adicionales, más pequeños y circulares de ADN conocidos como plásmidos que fácilmente pueden pasar de una cepa bacteriana a otra. Un plásmido es como un camión de reparto de genes bacterianos; es la forma principal como los genes de resistencia a los antibióticos se difunden entre las bacterias.
La variedad de cepas que descubrieron que codificaban la carbpenemasa de Klebsiella pneumoniae (KPC) y la metalo-β-Lacatamasa-1 de Nueva Delhi (NDM-1) fue consistente con la evidencia existente de que la transferencia horizontal de genes (HGT) es un factor importante en su propagación. Ellos identificaron algunos casos claves en los que el plásmido que llevaba NDM-1 o KPC eran casi idénticos, lo que significa que esto fácilmente podría facilitar la propagación de la resistencia a los antibióticos entre las bacterias que causan enfermedades que se encuentran en los EUA y en el sur de Asia.
Gautam Dantas, PhD, profesor asociado de patología e inmunología y autor principal del estudio, dijo: “Nuestros resultados también sugieren que va a ser más fácil para las cepas de estas bacterias, que aún no son resistentes, captar un gen que les permite sobrevivir el tratamiento con carbapenem. Los carbapenémicos son uno de nuestros últimos recursos para el tratamiento de infecciones bacterianas, los que utilizamos cuando nada más funciona. A medida que las formas resistentes a los medicamentos de Enterobacterias se vuelvan más generalizadas, las probabilidades aumentarán de transmitir una de estas súperbacterias a un amigo con un sistema inmunológico debilitado que realmente puede ser afectado por ellas”. El estudio fue publicado en línea en marzo de 2015 para la edición de junio de 2015 de la revista Emerging Infectious Diseases.
Enlaces relacionados:
Escuela de Medicina de la Universidad Washington
Hospital Barnes Jewish
Se han identificado dos genes que confieren resistencia contra una clase particularmente fuerte de antibióticos y que pueden ser compartidos fácilmente entre una familia de bacterias responsables de una parte importante de infecciones respiratorias y urinarias adquiridas en los hospitales.
Los científicos de la Facultad de Medicina de la Universidad de Washington (St. Louis, MO, EUA) recolectaron 450 aislados bacterianos, incluyendo 195 Enterobacterias en Pakistán, durante febrero de 2012 hasta marzo de 2013. Los investigadores seleccionaron al azar 55 aislados de Enterobacterias para hacerles la secuenciación de todo el genoma. Luego seleccionaron 23 aislamientos de muestras obtenidas de pacientes en los EUA entre enero de 2010 y junio de 2013, en el Hospital Judío Barnes (St. Louis, MO, EUA), que tenían una proporción similar de susceptibilidad y resistencia a los β-lactámicos a los recolectados en Pakistán para la secuenciación.
Los investigadores también secuenciaron una porción especial de material genético de las bacterias, llamados plásmidos. La mayor parte del ADN de una bacteria se encuentra en su cromosoma, pero las bacterias también tienen muchos pedazos adicionales, más pequeños y circulares de ADN conocidos como plásmidos que fácilmente pueden pasar de una cepa bacteriana a otra. Un plásmido es como un camión de reparto de genes bacterianos; es la forma principal como los genes de resistencia a los antibióticos se difunden entre las bacterias.
La variedad de cepas que descubrieron que codificaban la carbpenemasa de Klebsiella pneumoniae (KPC) y la metalo-β-Lacatamasa-1 de Nueva Delhi (NDM-1) fue consistente con la evidencia existente de que la transferencia horizontal de genes (HGT) es un factor importante en su propagación. Ellos identificaron algunos casos claves en los que el plásmido que llevaba NDM-1 o KPC eran casi idénticos, lo que significa que esto fácilmente podría facilitar la propagación de la resistencia a los antibióticos entre las bacterias que causan enfermedades que se encuentran en los EUA y en el sur de Asia.
Gautam Dantas, PhD, profesor asociado de patología e inmunología y autor principal del estudio, dijo: “Nuestros resultados también sugieren que va a ser más fácil para las cepas de estas bacterias, que aún no son resistentes, captar un gen que les permite sobrevivir el tratamiento con carbapenem. Los carbapenémicos son uno de nuestros últimos recursos para el tratamiento de infecciones bacterianas, los que utilizamos cuando nada más funciona. A medida que las formas resistentes a los medicamentos de Enterobacterias se vuelvan más generalizadas, las probabilidades aumentarán de transmitir una de estas súperbacterias a un amigo con un sistema inmunológico debilitado que realmente puede ser afectado por ellas”. El estudio fue publicado en línea en marzo de 2015 para la edición de junio de 2015 de la revista Emerging Infectious Diseases.
Enlaces relacionados:
Escuela de Medicina de la Universidad Washington
Hospital Barnes Jewish
Últimas Microbiología noticias
- Método de diagnóstico en sangre identifica infecciones IVRI pediátricas
- Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Prueba rápida no invasiva utiliza huella de azúcar para detectar infecciones por hongos
- Dispositivo de diagnóstico rápido de sepsis para atención crítica personalizada a pacientes de UCI
- Plataforma microfluídica evalúa función de neutrófilos en pacientes con sepsis
- Nuevo método diagnóstico confirma sepsis de forma más temprana
- Nuevos marcadores podrían predecir riesgo de infección grave por clamidia
- Espectroscopia portátil detecta de forma rápida y no invasiva bacterias en fluido vaginal
- Prueba de saliva basada en CRISPR detecta tuberculosis en esputo
- Análisis de orina diagnostica infección pulmonar común en personas inmunodeprimidas
- Prueba salival detecta riesgos microbianos relacionados con implantes
Canales
Química Clínica
ver canal
Herramienta en línea detecta exposición a medicamentos directamente en muestras de pacientes
Los médicos suelen basarse en entrevistas con pacientes y sus historiales médicos para determinar qué medicamentos ha tomado una persona, pero esta información suele ser incompleta.... Más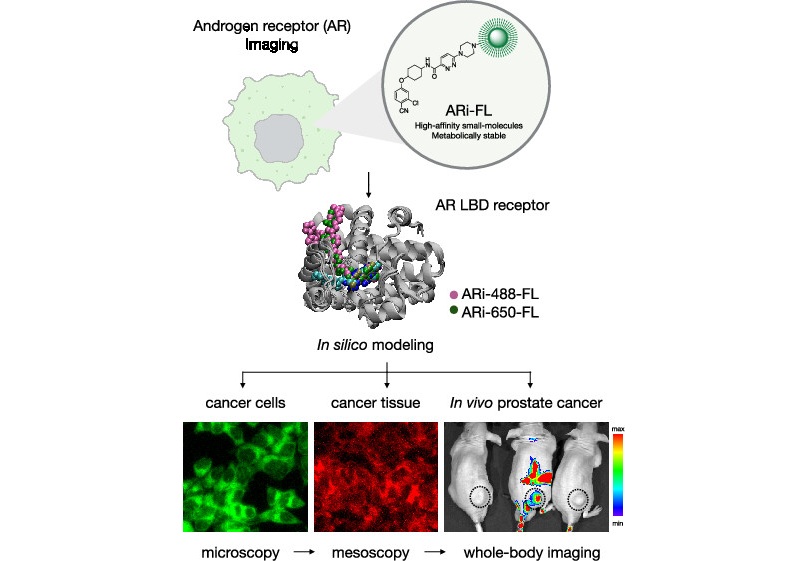
Sonda de imágenes químicas podría rastrear y tratar cáncer de próstata
El cáncer de próstata sigue siendo una de las principales causas de enfermedad y muerte en hombres, y muchos pacientes desarrollan resistencia a las terapias hormonales bloqueantes convencionales.... Más
Discrepancia entre dos pruebas comunes de función renal indica problemas de salud graves
La creatinina ha sido durante mucho tiempo el método estándar para medir la filtración renal, mientras que la cistatina C, una proteína producida por todas las células humanas, se ha recomendado como marcador... MásDiagnóstico Molecular
ver canal
Método ultrasensible detecta mutaciones cancerígenas de baja frecuencia
La biopsia líquida se ha convertido en un método prometedor para la detección del cáncer y la monitorización del tratamiento, aunque su impacto clínico se ha visto... Más
Análisis sanguíneo permite detección no invasiva de endometriosis
La endometriosis es un trastorno ginecológico crónico, complejo y relativamente común, que afecta a 1 de cada 10 mujeres adultas y adolescentes. La endometriosis provoca el crecimiento... MásHematología
ver canal
Pruebas de MRD podrían predecir supervivencia en pacientes con leucemia
La leucemia mieloide aguda es un cáncer sanguíneo agresivo que altera la producción normal de células sanguíneas y suele recaer incluso después de un tratamiento intensivo.... Más
Análisis sanguíneo de actividad plaquetaria en mediana edad podría identificar riesgo temprano de Alzheimer
La detección temprana de la enfermedad de Alzheimer sigue siendo una de las mayores necesidades insatisfechas en neurología, sobre todo porque los cambios biológicos que subyacen al... MásInmunología
ver canal
Análisis de sangre rutinario puede predecir mayor beneficiario de terapia con células T CAR
La terapia con células T CAR ha transformado el tratamiento para pacientes con linfoma no Hodgkin en recaída o resistente al tratamiento. Sin embargo, muchos pacientes finalmente recaen a... Más
Nueva prueba distingue falsos positivos inducidos por vacuna de infección activa por VIH
Desde que se identificó el VIH en 1983, más de 91 millones de personas han contraído el virus y más de 44 millones han fallecido por causas relacionadas. Hoy en día, casi 40 millones de personas en todo... MásPatología
ver canal
Pruebas rápidas y económicas pueden prevenir muertes infantiles por jarabes medicinales contaminados
Jarabes medicinales contaminados con sustancias químicas tóxicas han causado la muerte de cientos de niños en todo el mundo, lo que revela una grave deficiencia en el análisis... Más
Señales tumorales en saliva y sangre permiten monitorización no invasiva del cáncer de cabeza y cuello
Los cánceres de cabeza y cuello se encuentran entre las neoplasias malignas más agresivas del mundo, con casi 900.000 nuevos casos diagnosticados cada año. El seguimiento de estos... MásTecnología
ver canal
Modelos de aprendizaje automático diagnostican ELA antes mediante biomarcadores sanguíneos
La esclerosis lateral amiotrófica (ELA) es una enfermedad neurodegenerativa de rápida progresión, notoriamente difícil de diagnosticar en sus etapas iniciales.... Más
Modelo de inteligencia artificial podría acelerar diagnóstico de enfermedades raras
Identificar qué variantes genéticas causan enfermedades sigue siendo uno de los mayores desafíos de la medicina genómica. Cada persona porta decenas de miles de cambios en el... MásIndustria
ver canal
Abbott adquiere Exact Sciences, empresa de detección de cáncer
Abbott (Abbott Park, IL, EUA) ha firmado un acuerdo definitivo para adquirir Exact Sciences (Madison, WI, EUA), lo que le permitirá entrar y liderar en segmentos de diagnóstico de cáncer... Más