Desarrollan prueba novedosa para detectar e identificar patógenos
|
Por el equipo editorial de LabMedica en español Actualizado el 05 Oct 2021 |
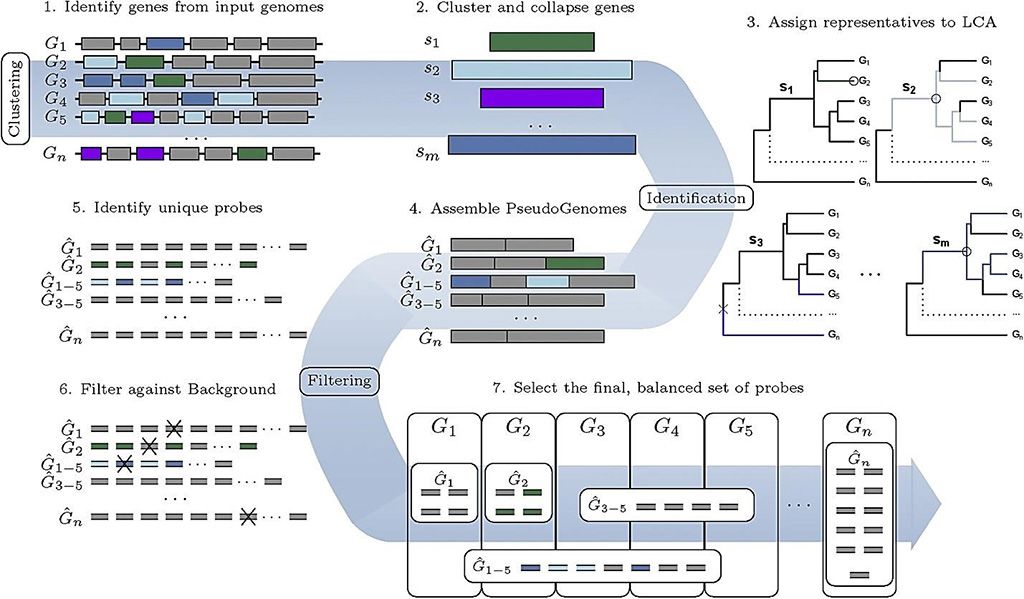
Imagen: El flujo de trabajo de la línea de productos en proyecto, HUBDesign: diseño de sonda para la captura simultánea y dirigida de diversos objetivos metagenómicos (Fotografía cortesía de la Universidad McMaster)
Los patógenos en entornos clínicos o de vida silvestre, como las muestras de sangre o saliva, por ejemplo, son particularmente difíciles de aislar, ya que pueden constituir fácilmente menos de una millonésima parte de una muestra, especialmente en las primeras etapas de una infección, cuando las concentraciones todavía son bajas y la detección es más crítica para los pacientes.
Una amplia gama de esfuerzos de estudios metagenómicos se ven obstaculizados por el mismo desafío: bajas concentraciones de objetivos de interés combinadas con cantidades abrumadoras de señal de fondo. Aunque se puede utilizar PCR o captura de ADN sin tratamiento previo cuando hay una pequeña cantidad de organismos de interés, los desafíos de diseño se vuelven insostenibles para una gran cantidad de objetivos.
Un equipo de científicos de la Universidad McMaster (Hamilton, ON, Canadá) y sus colegas desarrollaron una herramienta sofisticada nueva que podría ayudar a proporcionar una alerta temprana de virus raros y desconocidos en el medio ambiente e identificar patógenos bacterianos potencialmente mortales que causan sepsis. El nuevo algoritmo es una herramienta avanzada que puede ayudar a desarrollar sondas para capturar trazas de patógenos, tanto conocidos como desconocidos provenientes de una amplia variedad de situaciones, como la transmisión de infecciones de animal a humano como el SARS-CoV-2 o los reservorios de monitoreo en el medio ambiente por posibles patógenos emergentes.
El equipo probó con éxito las sondas en toda la familia de coronavirus, incluido el SARS-CoV-2. Las sondas proporcionan un atajo al apuntar, aislar e identificar, las secuencias de ADN, de manera específica y simultánea, que se comparten entre organismos relacionados, con mayor frecuencia debido a la historia evolutiva o la ascendencia. Para demostrar las capacidades y la eficacia de la herramienta, el Diseño de Cebos Jerárquicos Únicos (The Hierarchical Unique Bait Design, HUBDesign), diseñaron y probaron dos conjuntos de sondas: un conjunto de sondas de coronavirus capaz de detectar simultáneamente todos los coronavirus secuenciados y un conjunto de sondas dirigidas a patógenos bacterianos asociados con la sepsis.
La mayoría (62,5%) de las sondas tienen objetivos específicos para un virus. De las sondas que se dirigen a varios virus, la mayoría (78,1%) se dirigen a dos o tres. Los tres conjuntos restantes de sondas se dirigen a loci específicos de merbecovirus y embecovirus (ambos son subgéneros de Betacoronavirus) y loci comunes al género Deltacoronavirus. Tanto el SARS-CoV-2 como el HCoV-NL63 tienen sondas en dos niveles en la jerarquía.
El conjunto de sondas HUBDesign para patógenos de la sepsis contenía 26.870 sondas dirigidas a patógenos bacterianos, que cubren el 2,09% de todos los nucleótidos en el conjunto de datos de entrada a una profundidad media de cobertura de 3,64x. Los investigadores demostraron la efectividad de las sondas para capturar la increíble variedad de patógenos asociados con la sepsis, una condición potencialmente mortal y de rápido desarrollo que ocurre cuando el cuerpo reacciona de forma exagerada a una infección que generalmente comienza en los pulmones, el tracto urinario, la piel o el tracto gastrointestinal.
Hendrik Poinar, PhD, profesor de Genética Evolutiva Molecular y autor principal del estudio, dijo: “Actualmente necesitamos formas más rápidas, económicas y concisas de detectar patógenos en muestras humanas y ambientales que democraticen la caza y esta línea de productos en proyecto hace exactamente eso”. El estudio fue publicado el 15 de septiembre de 2021 en la revista Cell Reports Methods.
Enlace relacionado:
Universidad McMaster
Una amplia gama de esfuerzos de estudios metagenómicos se ven obstaculizados por el mismo desafío: bajas concentraciones de objetivos de interés combinadas con cantidades abrumadoras de señal de fondo. Aunque se puede utilizar PCR o captura de ADN sin tratamiento previo cuando hay una pequeña cantidad de organismos de interés, los desafíos de diseño se vuelven insostenibles para una gran cantidad de objetivos.
Un equipo de científicos de la Universidad McMaster (Hamilton, ON, Canadá) y sus colegas desarrollaron una herramienta sofisticada nueva que podría ayudar a proporcionar una alerta temprana de virus raros y desconocidos en el medio ambiente e identificar patógenos bacterianos potencialmente mortales que causan sepsis. El nuevo algoritmo es una herramienta avanzada que puede ayudar a desarrollar sondas para capturar trazas de patógenos, tanto conocidos como desconocidos provenientes de una amplia variedad de situaciones, como la transmisión de infecciones de animal a humano como el SARS-CoV-2 o los reservorios de monitoreo en el medio ambiente por posibles patógenos emergentes.
El equipo probó con éxito las sondas en toda la familia de coronavirus, incluido el SARS-CoV-2. Las sondas proporcionan un atajo al apuntar, aislar e identificar, las secuencias de ADN, de manera específica y simultánea, que se comparten entre organismos relacionados, con mayor frecuencia debido a la historia evolutiva o la ascendencia. Para demostrar las capacidades y la eficacia de la herramienta, el Diseño de Cebos Jerárquicos Únicos (The Hierarchical Unique Bait Design, HUBDesign), diseñaron y probaron dos conjuntos de sondas: un conjunto de sondas de coronavirus capaz de detectar simultáneamente todos los coronavirus secuenciados y un conjunto de sondas dirigidas a patógenos bacterianos asociados con la sepsis.
La mayoría (62,5%) de las sondas tienen objetivos específicos para un virus. De las sondas que se dirigen a varios virus, la mayoría (78,1%) se dirigen a dos o tres. Los tres conjuntos restantes de sondas se dirigen a loci específicos de merbecovirus y embecovirus (ambos son subgéneros de Betacoronavirus) y loci comunes al género Deltacoronavirus. Tanto el SARS-CoV-2 como el HCoV-NL63 tienen sondas en dos niveles en la jerarquía.
El conjunto de sondas HUBDesign para patógenos de la sepsis contenía 26.870 sondas dirigidas a patógenos bacterianos, que cubren el 2,09% de todos los nucleótidos en el conjunto de datos de entrada a una profundidad media de cobertura de 3,64x. Los investigadores demostraron la efectividad de las sondas para capturar la increíble variedad de patógenos asociados con la sepsis, una condición potencialmente mortal y de rápido desarrollo que ocurre cuando el cuerpo reacciona de forma exagerada a una infección que generalmente comienza en los pulmones, el tracto urinario, la piel o el tracto gastrointestinal.
Hendrik Poinar, PhD, profesor de Genética Evolutiva Molecular y autor principal del estudio, dijo: “Actualmente necesitamos formas más rápidas, económicas y concisas de detectar patógenos en muestras humanas y ambientales que democraticen la caza y esta línea de productos en proyecto hace exactamente eso”. El estudio fue publicado el 15 de septiembre de 2021 en la revista Cell Reports Methods.
Enlace relacionado:
Universidad McMaster
Últimas Diagnóstico Molecular noticias
- Nuevo análisis sanguíneo detecta enfermedad hepática oculta relacionada con alcohol
- Nuevo análisis de sangre predice quienes tienen más probabilidades de vivir más
- Prueba genética predice riesgo de radioterapia en pacientes con cáncer de próstata
- Prueba genética facilita detección temprana y mejora tratamiento del cáncer
- Nueva técnica de secuenciación genómica mide virus de Epstein-Barr en sangre
- Análisis de sangre mejora detección temprana del cáncer cerebral
- Monitoreo molecular ayuda a pacientes con cáncer de vejiga a evitar cirugía
- Pruebas genéticas aceleran el diagnóstico de trastornos linfáticos
- Cambios en vasos linfáticos pueden ayudar a identificación temprana del cáncer oral agresivo
- Nuevo kit de extracción permite aislamiento consistente y escalable de ADNlc de biofluidos
- Ensayo de biopsia líquida de LCR revela información genómica sobre tumores del SNC
- Biopsia líquida impulsada por IA clasifica tumores cerebrales pediátricos con alta precisión
- Prueba molecular de estreptococo A ofrece resultados definitivos en POC en 15 minutos
- Prueba molecular rápida identifica pacientes con sepsis con mayor probabilidad de hemocultivos positivos
- Sensor basado en luz detecta signos moleculares tempranos de cáncer en sangre
- Nuevo método probatorio predice recuperación de pacientes con trauma con antelación
Canales
Química Clínica
ver canal
Nariz electrónica detecta signos tempranos de cáncer de ovario en sangre
El cáncer de ovario suele diagnosticarse en una etapa tardía debido a que sus síntomas son vagos y similares a los de afecciones más comunes. A diferencia del cáncer... Más
Simple análisis sanguíneo ofrece nueva vía para evaluación del Alzheimer en atención primaria
La evaluación oportuna de los síntomas cognitivos en atención primaria suele verse limitada por el acceso restringido a diagnósticos especializados y procedimientos confirmatorios... MásHematología
ver canal
Prueba rápida en cartuchos busca ampliar acceso al diagnóstico de trastornos de hemoglobina
La anemia de células falciformes y la beta talasemia son trastornos de la hemoglobina que a menudo requieren la derivación a laboratorios especializados para un diagnóstico definitivo,... Más
Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
La amiloidosis de cadenas ligeras (AL) es un trastorno poco común y potencialmente mortal de la médula ósea, en el que las proteínas amiloides anormales se acumulan en los órganos. Aproximadamente 3.... MásInmunología
ver canal
Nuevo biomarcador predice respuesta a quimioterapia en cáncer de mama triple negativo
El cáncer de mama triple negativo es una forma agresiva de cáncer de mama en la que las pacientes suelen mostrar respuestas muy variables a la quimioterapia. Predecir quién se ben... MásAnálisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
El cáncer de pulmón de células pequeñas (CPCP) es una enfermedad agresiva con opciones de tratamiento limitadas, e incluso las inmunoterapias recientemente aprobadas no benefician... Más
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... MásMicrobiología
ver canal
Virus intestinales ocultos vinculados al riesgo de cáncer colorrectal
El cáncer colorrectal (CCR) sigue siendo una de las principales causas de mortalidad por cáncer en muchos países occidentales, y los enfoques actuales de estratificación del... Más
Panel de tres pruebas para detección de infecciones por duelas del hígado
Las infecciones parasitarias por duelas hepáticas siguen siendo endémicas en algunas zonas de Asia, donde la transmisión se produce comúnmente a través del consumo de... MásPatología
ver canal
Sistema de recolección de orina mejora precisión y eficiencia del diagnóstico
El análisis de orina es una herramienta diagnóstica crucial y no invasiva que se utiliza para detectar afecciones como embarazo, infecciones del tracto urinario, trastornos metabólicos,... Más
Escaneo 3D con IA acelera detección del cáncer
La citología sigue siendo fundamental en la detección del cáncer, ya que requiere que los especialistas examinen los fluidos y células corporales bajo el microscopio.... MásTecnología
ver canal
“Relojes” de análisis sanguíneos predicen inicio de síntomas del Alzheimer
Más de 7 millones de estadounidenses viven con la enfermedad de Alzheimer, y se proyecta que los costos de salud y atención a largo plazo relacionados alcancen casi 400 mil millones de dólares... Más
Biomarcador de IA predice riesgo de cáncer de hígado
El cáncer de hígado, o carcinoma hepatocelular, causa más de 800.000 muertes al año en todo el mundo y, a menudo, no se detecta hasta las últimas etapas.... MásIndustria
ver canal
Cepheid se une a iniciativa del CDC para fortalecer pruebas detección de pandemias
Cepheid (Sunnyvale, CA, EUA) ha sido seleccionado por los Centros para el Control y la Prevención de Enfermedades (CDC) de EUA como uno de los cuatro colaboradores nacionales en una iniciativa federal... Más