Cepas de Salmonella resistentes a los medicamentos causan infecciones del torrente sanguíneo
|
Por el equipo editorial de LabMedica en español Actualizado el 09 Oct 2019 |
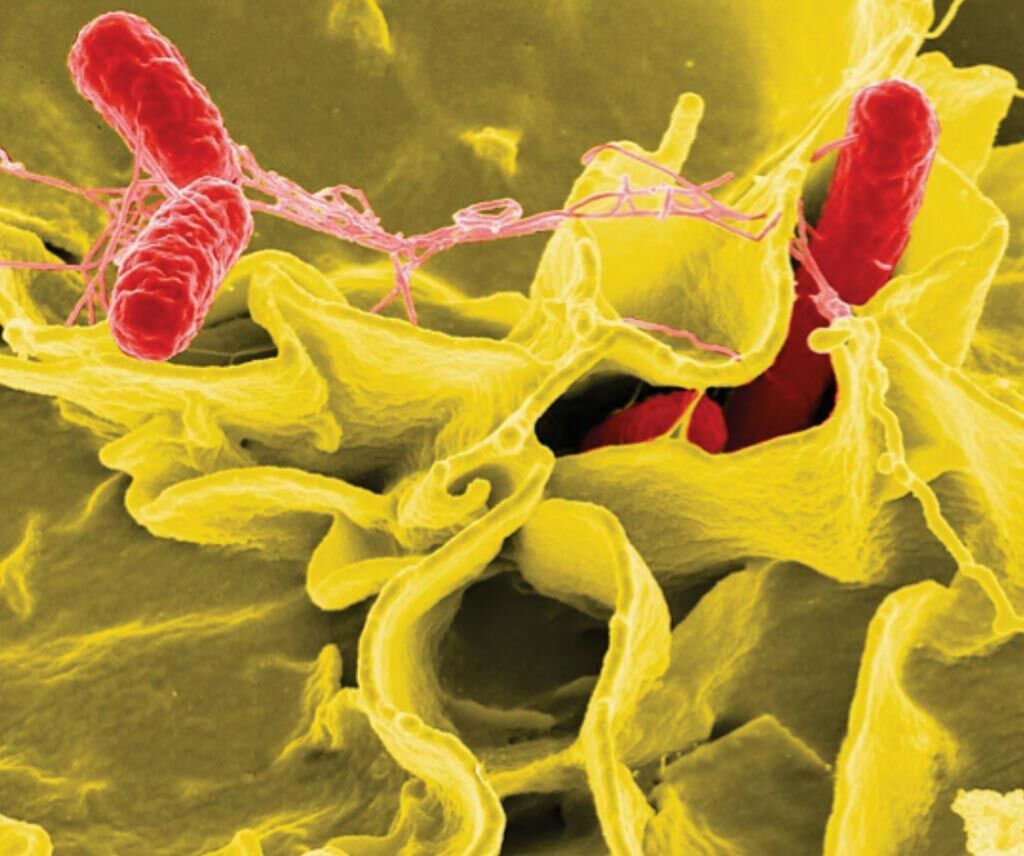
Imagen: Microfotografía electrónica de barrido realzada con color mostrando células humanas cultivadas invadidas por Salmonella typhimurium (rojo) (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los EUA).
Salmonella enterica subespecie enterica serovar Typhimurium (conocida como S. typhimurium) y otras cepas de Salmonella no tifoideas son causas comunes de infecciones gastrointestinales en personas que viven en los países industrializados.
Sin embargo, en el África subsahariana (SSA), las infecciones invasivas del torrente sanguíneo por Salmonella no tifoidea (iNTS) son comunes, con un total de alrededor de 3,4 millones de casos anualmente, siendo S. typhimurium responsable de aproximadamente dos tercios de estos casos. La resistencia a los medicamentos ha aumentado en grupos sucesivos de S. typhimurium con el tiempo y la tasa de mortalidad por iNTS puede ser extremadamente alta.
Los científicos del Instituto de Medicina Tropical (Amberes, Bélgica) y sus colegas recolectaron muestras de sangre de personas con sospecha de infecciones del torrente sanguíneo en hospitales de la República Democrática del Congo. Se incluyeron todas las cepas de S. typhimurium resistentes a azitromicina (AZI) disponibles para este estudio (n = 54). Se seleccionaron muestras de 27 aislados representativos de S. typhimurium no resistentes a AZI como controles para este análisis.
Los aislamientos, confirmados bioquímicamente como Salmonella spp., fueron serotipifcados utilizando antisueros comerciales (Sifin, Berlín, Alemania). El ADN de las 81 cepas se purificó utilizando el kit Gentra PureGene Yeast/Bact (Qiagen, Hilden, Alemania), siguiendo las pautas del fabricante y el ADN se secuenció en una plataforma Illumina HiSeq (Illumina, San Diego, CA, EUA). El contenido del adaptador Illumina se eliminó de las lecturas usando Trimmomatic v.0.33.
Se sabe que las infecciones por iNTS en el África subsahariana están dominadas por un tipo de S. typhimurium conocido como ST313, asociado con la resistencia a los antibióticos. Dos grupos de ST313 (denominados linaje I y II) se separaron de forma independiente y posteriormente se extendieron por el continente africano. El tiempo ha elevado la resistencia a los antibióticos y el linaje II ahora es la causa principal de las infecciones por iNTS. El análisis de estos genomas de S. typhimurium identificó un nuevo subgrupo que se ramifica de ST313, llamado linaje II.1. Se estima que este subgrupo, que surgió en 2004, muestra una extensa resistencia a los medicamentos (XDR).
Sandra Van Puyvelde, PhD, profesora asistente y primera autora del estudio, dijo: “Todos los genes de resistencia a los antibióticos que contribuyen a ‘XDR’ están presentes en el mismo plásmido. Esto es preocupante porque un plásmido es un elemento genético móvil que se podría transferir a otras bacterias. Mientras acumulamos más resistencia a los antibióticos, descubrimos que la nueva línea de Salmonella typhimurium también muestra más cambios genéticos y de comportamiento que sugieren una evolución continua de las bacterias hacia las infecciones del torrente sanguíneo”. El estudio fue publicado el 19 de septiembre de 2019 en la revista Nature Communications.
Enlace relacionado:
Instituto de Medicina Tropical
Sifin
Qiagen
Illumina
Sin embargo, en el África subsahariana (SSA), las infecciones invasivas del torrente sanguíneo por Salmonella no tifoidea (iNTS) son comunes, con un total de alrededor de 3,4 millones de casos anualmente, siendo S. typhimurium responsable de aproximadamente dos tercios de estos casos. La resistencia a los medicamentos ha aumentado en grupos sucesivos de S. typhimurium con el tiempo y la tasa de mortalidad por iNTS puede ser extremadamente alta.
Los científicos del Instituto de Medicina Tropical (Amberes, Bélgica) y sus colegas recolectaron muestras de sangre de personas con sospecha de infecciones del torrente sanguíneo en hospitales de la República Democrática del Congo. Se incluyeron todas las cepas de S. typhimurium resistentes a azitromicina (AZI) disponibles para este estudio (n = 54). Se seleccionaron muestras de 27 aislados representativos de S. typhimurium no resistentes a AZI como controles para este análisis.
Los aislamientos, confirmados bioquímicamente como Salmonella spp., fueron serotipifcados utilizando antisueros comerciales (Sifin, Berlín, Alemania). El ADN de las 81 cepas se purificó utilizando el kit Gentra PureGene Yeast/Bact (Qiagen, Hilden, Alemania), siguiendo las pautas del fabricante y el ADN se secuenció en una plataforma Illumina HiSeq (Illumina, San Diego, CA, EUA). El contenido del adaptador Illumina se eliminó de las lecturas usando Trimmomatic v.0.33.
Se sabe que las infecciones por iNTS en el África subsahariana están dominadas por un tipo de S. typhimurium conocido como ST313, asociado con la resistencia a los antibióticos. Dos grupos de ST313 (denominados linaje I y II) se separaron de forma independiente y posteriormente se extendieron por el continente africano. El tiempo ha elevado la resistencia a los antibióticos y el linaje II ahora es la causa principal de las infecciones por iNTS. El análisis de estos genomas de S. typhimurium identificó un nuevo subgrupo que se ramifica de ST313, llamado linaje II.1. Se estima que este subgrupo, que surgió en 2004, muestra una extensa resistencia a los medicamentos (XDR).
Sandra Van Puyvelde, PhD, profesora asistente y primera autora del estudio, dijo: “Todos los genes de resistencia a los antibióticos que contribuyen a ‘XDR’ están presentes en el mismo plásmido. Esto es preocupante porque un plásmido es un elemento genético móvil que se podría transferir a otras bacterias. Mientras acumulamos más resistencia a los antibióticos, descubrimos que la nueva línea de Salmonella typhimurium también muestra más cambios genéticos y de comportamiento que sugieren una evolución continua de las bacterias hacia las infecciones del torrente sanguíneo”. El estudio fue publicado el 19 de septiembre de 2019 en la revista Nature Communications.
Enlace relacionado:
Instituto de Medicina Tropical
Sifin
Qiagen
Illumina
Últimas Microbiología noticias
- Prueba rápida promete respuestas inmediatas a infecciones resistentes a medicamentos
- Tecnología con CRISPR neutraliza bacterias resistentes a antibióticos
- Nuevo estudio identifica características del microbioma intestinal asociadas con enfermedad de Alzheimer
- Plataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- Método de diagnóstico en sangre identifica infecciones IVRI pediátricas
- Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Prueba rápida no invasiva utiliza huella de azúcar para detectar infecciones por hongos
Canales
Química Clínica
ver canal
Método rápido de análisis sanguíneo permite decisiones más seguras en emergencias por medicamentos
La intoxicación aguda por drogas recreativas es un motivo frecuente de visitas a urgencias; sin embargo, los médicos rara vez tienen acceso a resultados toxicológicos confirmatorios... Más
Modelo pronóstico basado en APE mejora evaluación del riesgo de cáncer
El cáncer de próstata es la segunda causa principal de muerte por cáncer en los hombres estadounidenses, y aproximadamente uno de cada ocho será diagnosticado a lo largo de su vida.... MásDiagnóstico Molecular
ver canal
Ensayo de biopsia líquida de LCR revela información genómica sobre tumores del SNC
Las neoplasias malignas del sistema nervioso central (SNC) plantean desafíos diagnósticos específicos debido a que las pruebas basadas en tejido suelen ser inviables y la barrera ... Más
Biopsia líquida impulsada por IA clasifica tumores cerebrales pediátricos con alta precisión
Las biopsias líquidas ofrecen una forma no invasiva de estudiar el cáncer mediante el análisis del ADN tumoral circulante en fluidos corporales. Sin embargo, en los tumores cerebrales... MásHematología
ver canal
Prueba rápida en cartuchos busca ampliar acceso al diagnóstico de trastornos de hemoglobina
La anemia de células falciformes y la beta talasemia son trastornos de la hemoglobina que a menudo requieren la derivación a laboratorios especializados para un diagnóstico definitivo,... Más
Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
La amiloidosis de cadenas ligeras (AL) es un trastorno poco común y potencialmente mortal de la médula ósea, en el que las proteínas amiloides anormales se acumulan en los órganos. Aproximadamente 3.... MásInmunología
ver canal
Nuevo biomarcador predice respuesta a quimioterapia en cáncer de mama triple negativo
El cáncer de mama triple negativo es una forma agresiva de cáncer de mama en la que las pacientes suelen mostrar respuestas muy variables a la quimioterapia. Predecir quién se ben... MásAnálisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
El cáncer de pulmón de células pequeñas (CPCP) es una enfermedad agresiva con opciones de tratamiento limitadas, e incluso las inmunoterapias recientemente aprobadas no benefician... Más
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... MásPatología
ver canal
Plataforma de IA estandariza flujo de trabajo de microscopía de frotis de tuberculosis
La baciloscopia de esputo sigue siendo fundamental para la monitorización y el seguimiento del tratamiento de la tuberculosis, especialmente en entornos con alta incidencia de tuberculosis, donde... Más
Herramienta de IA predice complicaciones del trasplante mediante biomarcadores sanguíneos antes de síntomas
Los trasplantes de células madre y médula ósea pueden salvar vidas, pero pueden surgir complicaciones graves meses después de que los pacientes sean dados de alta del hospital.... MásTecnología
ver canal
Biomarcador de IA predice riesgo de cáncer de hígado
El cáncer de hígado, o carcinoma hepatocelular, causa más de 800.000 muertes al año en todo el mundo y, a menudo, no se detecta hasta las últimas etapas.... Más
Tecnología robótica para extracciones de sangre diagnósticas automatizadas
La extracción rutinaria de sangre para diagnóstico es una tarea de alto volumen que puede sobrecargar al personal e introducir variabilidad dependiente del factor humano, con implicaciones... MásIndustria
ver canal
QuidelOrtho colabora con Lifotronic para ampliar cartera global de inmunoensayos
QuidelOrtho (San Diego, CA, EUA) ha firmado un acuerdo de suministro estratégico a largo plazo con Lifotronic Technology (Shenzhen, China) para expandir su cartera global de inmunoensayos y acelerar... Más