Análisis rápido CRISPR/Cas para el diagnóstico de fiebres virales en ambientes de baja tecnología
|
Por el equipo editorial de LabMedica en español Actualizado el 07 Sep 2020 |
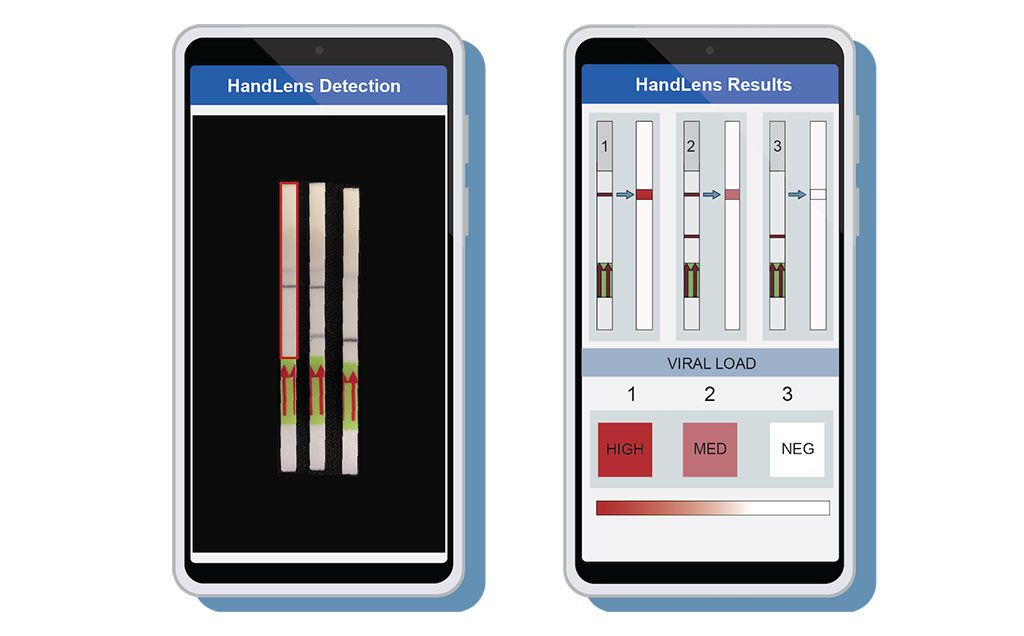
Imagen: Se diseñó una aplicación para teléfonos móviles llamada HandLens para leer e informar los resultados en las tiras de papel SHERLOCK (Fotografía cortesía de Anna Lachenauer, Facultad de Medicina de la Universidad de Stanford)
Las pruebas de diagnóstico rápido para los virus de la fiebre del Ébola y Lassa, basadas en el complejo CRISPR/Cas13a, dirigidas a la detección del ARN, se han probado con éxito en condiciones de campo en varios países africanos.
COVID-19 no es la única enfermedad que existe. Los recientes brotes de fiebres hemorrágicas virales (VHF), incluida la enfermedad por el virus del Ébola (EVE) y la fiebre de Lassa (LF), ponen de relieve la necesidad urgente de realizar pruebas sensibles y desplegables para diagnosticar estas devastadoras enfermedades humanas. En este sentido, los investigadores del Instituto Broad de MIT y Harvard (Cambridge, MA, EUA) y sus colaboradores, desarrollaron diagnósticos basados en CRISPR-Cas13a, dirigidos al virus del Ébola (EBOV) y al virus Lassa (LASV), con lecturas de flujo lateral y fluorescentes.
Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente interespaciadas) son segmentos de ADN procariótico que contienen repeticiones cortas de secuencias de bases. A cada repetición le siguen segmentos cortos de “ADN espaciador” de exposiciones anteriores a un virus bacteriano o plásmido. Desde 2013, se ha utilizado el sistema CRISPR/Cas9 en la investigación para la edición de genes (agregar, interrumpir o cambiar la secuencia de genes específicos) y la regulación de genes. Al administrar la enzima Cas9 y los ARN guía apropiados (ARNsg) a una célula, el genoma del organismo se puede cortar en cualquier ubicación deseada.
Los recientes esfuerzos computacionales para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida al ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación del ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, guiado por ARN, dirigido a la detección del ARN, que tiene un gran potencial para aplicaciones precisas, robustas y escalables, guiadas por ARN dirigidas a la detección de ARN.
En el desarrollo de un método para diagnósticos desplegables y rápidamente adaptables, los investigadores trabajaron con la plataforma SHERLOCK (siglas en inglés para Desbloqueo de Reportero Enzimático Específico de Alta Sensibilidad), basada en CRISPR. SHERLOCK utiliza la proteína Cas13a dirigida al ARN para la detección sensible y específica del ácido nucleico viral. Este método funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también puede ser cuantificada. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple tira de papel, en un equipo de laboratorio o para proporcionar una lectura electroquímica que se puede leer con un teléfono móvil.
SHERLOCK se puede combinar con el método HUDSON (Calentamiento de Muestras de Diagnóstico no Extraídas para Eliminar Nucleasas), que inactiva patógenos y libera ácido nucleico mediante una desnaturalización química y térmica combinada, eliminando la necesidad de una extracción de ácidos nucleicos basada en columna o en perlas. Este proceso hace que las muestras de los pacientes sean más seguras para que el personal clínico las manipule en entornos de baja tecnología y elimina la necesidad de extraer el material genético de un virus de las muestras, antes del análisis.
Para completar el paquete, los investigadores también desarrollaron una aplicación para teléfonos móviles llamada HandLens, que puede leer e informar inmediatamente los resultados de las tiras de papel SHERLOCK. Esta herramienta puede ayudar en situaciones en las que la tira de papel emite una señal débil que es difícil de interpretar para un médico.
Los investigadores utilizaron la técnica SHERLOCK junto con HUDSON y HandLens en Nigeria durante un reciente brote de fiebre de Lassa y en Sierra Leona y la República Democrática del Congo durante los brotes del virus del Ébola. Los resultados revelaron que los ensayos SHERLOCK funcionaron de manera tan consistente o mejor que otras metodologías de diagnóstico, lo que demuestra el potencial de la plataforma para el uso clínico en áreas con recursos limitados.
La técnica SHERLOCK para el diagnóstico rápido de enfermedades virales se describió en la edición en línea del 17 de agosto de 2020 de la revista Nature Communications.
Enlace relacionado:
Instituto Broad de MIT y Harvard
COVID-19 no es la única enfermedad que existe. Los recientes brotes de fiebres hemorrágicas virales (VHF), incluida la enfermedad por el virus del Ébola (EVE) y la fiebre de Lassa (LF), ponen de relieve la necesidad urgente de realizar pruebas sensibles y desplegables para diagnosticar estas devastadoras enfermedades humanas. En este sentido, los investigadores del Instituto Broad de MIT y Harvard (Cambridge, MA, EUA) y sus colaboradores, desarrollaron diagnósticos basados en CRISPR-Cas13a, dirigidos al virus del Ébola (EBOV) y al virus Lassa (LASV), con lecturas de flujo lateral y fluorescentes.
Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente interespaciadas) son segmentos de ADN procariótico que contienen repeticiones cortas de secuencias de bases. A cada repetición le siguen segmentos cortos de “ADN espaciador” de exposiciones anteriores a un virus bacteriano o plásmido. Desde 2013, se ha utilizado el sistema CRISPR/Cas9 en la investigación para la edición de genes (agregar, interrumpir o cambiar la secuencia de genes específicos) y la regulación de genes. Al administrar la enzima Cas9 y los ARN guía apropiados (ARNsg) a una célula, el genoma del organismo se puede cortar en cualquier ubicación deseada.
Los recientes esfuerzos computacionales para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida al ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación del ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, guiado por ARN, dirigido a la detección del ARN, que tiene un gran potencial para aplicaciones precisas, robustas y escalables, guiadas por ARN dirigidas a la detección de ARN.
En el desarrollo de un método para diagnósticos desplegables y rápidamente adaptables, los investigadores trabajaron con la plataforma SHERLOCK (siglas en inglés para Desbloqueo de Reportero Enzimático Específico de Alta Sensibilidad), basada en CRISPR. SHERLOCK utiliza la proteína Cas13a dirigida al ARN para la detección sensible y específica del ácido nucleico viral. Este método funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también puede ser cuantificada. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple tira de papel, en un equipo de laboratorio o para proporcionar una lectura electroquímica que se puede leer con un teléfono móvil.
SHERLOCK se puede combinar con el método HUDSON (Calentamiento de Muestras de Diagnóstico no Extraídas para Eliminar Nucleasas), que inactiva patógenos y libera ácido nucleico mediante una desnaturalización química y térmica combinada, eliminando la necesidad de una extracción de ácidos nucleicos basada en columna o en perlas. Este proceso hace que las muestras de los pacientes sean más seguras para que el personal clínico las manipule en entornos de baja tecnología y elimina la necesidad de extraer el material genético de un virus de las muestras, antes del análisis.
Para completar el paquete, los investigadores también desarrollaron una aplicación para teléfonos móviles llamada HandLens, que puede leer e informar inmediatamente los resultados de las tiras de papel SHERLOCK. Esta herramienta puede ayudar en situaciones en las que la tira de papel emite una señal débil que es difícil de interpretar para un médico.
Los investigadores utilizaron la técnica SHERLOCK junto con HUDSON y HandLens en Nigeria durante un reciente brote de fiebre de Lassa y en Sierra Leona y la República Democrática del Congo durante los brotes del virus del Ébola. Los resultados revelaron que los ensayos SHERLOCK funcionaron de manera tan consistente o mejor que otras metodologías de diagnóstico, lo que demuestra el potencial de la plataforma para el uso clínico en áreas con recursos limitados.
La técnica SHERLOCK para el diagnóstico rápido de enfermedades virales se describió en la edición en línea del 17 de agosto de 2020 de la revista Nature Communications.
Enlace relacionado:
Instituto Broad de MIT y Harvard
Últimas Microbiología noticias
- Plataforma de IA permite detección rápida de patógenos de C. auris resistentes a fármacos
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- Método de diagnóstico en sangre identifica infecciones IVRI pediátricas
- Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Prueba rápida no invasiva utiliza huella de azúcar para detectar infecciones por hongos
- Dispositivo de diagnóstico rápido de sepsis para atención crítica personalizada a pacientes de UCI
- Plataforma microfluídica evalúa función de neutrófilos en pacientes con sepsis
- Nuevo método diagnóstico confirma sepsis de forma más temprana
Canales
Química Clínica
ver canal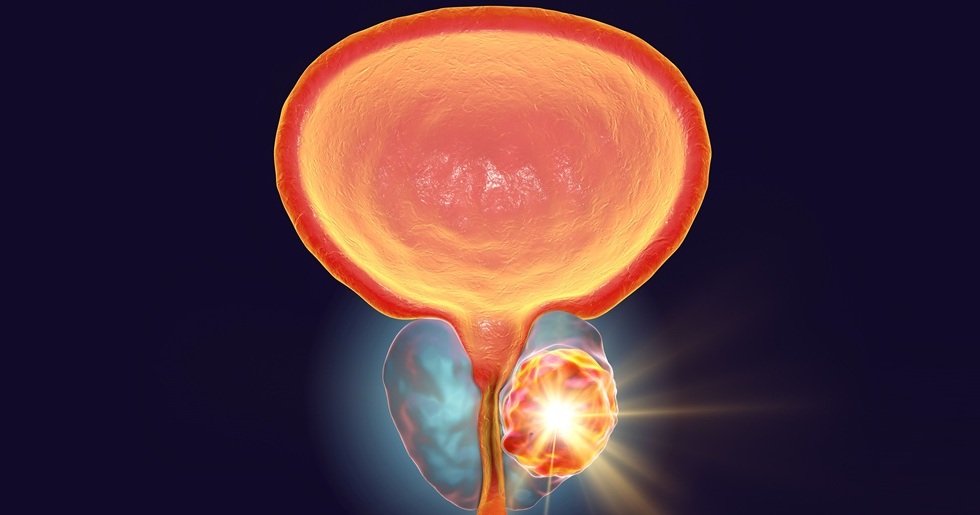
Modelo pronóstico basado en APE mejora evaluación del riesgo de cáncer
El cáncer de próstata es la segunda causa principal de muerte por cáncer en los hombres estadounidenses, y aproximadamente uno de cada ocho será diagnosticado a lo largo de su vida.... Más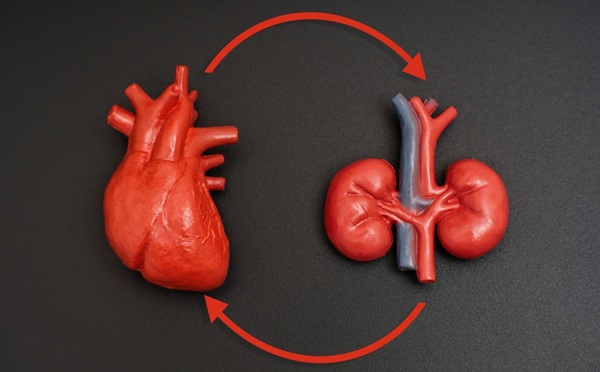
Vesículas extracelulares se relacionan con riesgo de insuficiencia cardíaca en pacientes con ERC
La enfermedad renal crónica (ERC) afecta a más de 1 de cada 7 estadounidenses y está estrechamente asociada con complicaciones cardiovasculares, las cuales representan más de... MásDiagnóstico Molecular
ver canal
Biopsia líquida podría reemplazar a biopsia quirúrgica para diagnosticar linfoma nervioso central primario
El linfoma primario del sistema nervioso central (LPSNC) se diagnostica habitualmente mediante biopsia quirúrgica, que sigue siendo el método de referencia, pero conlleva un riesgo considerable.... Más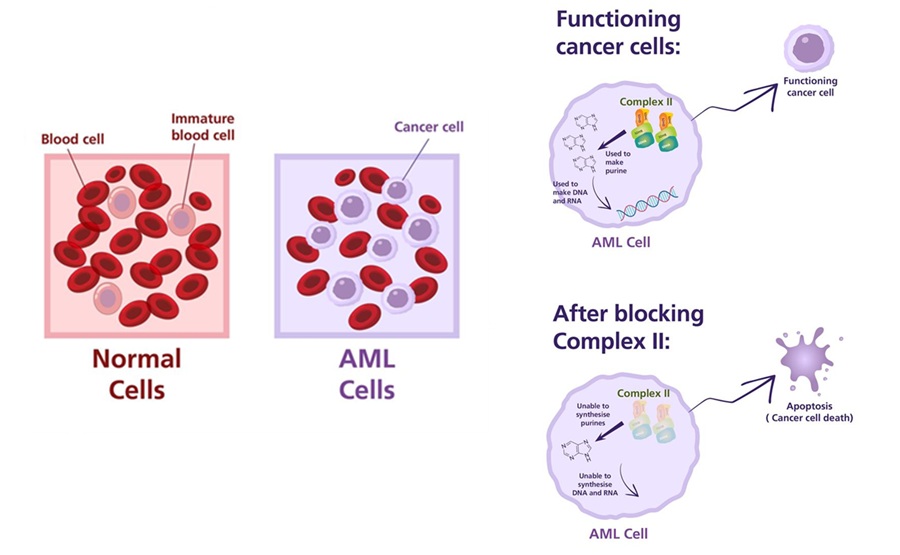
Herramienta revela debilidad metabólica oculta en cánceres de sangre
La leucemia mieloide aguda (LMA) es uno de los cánceres de sangre más agresivos, caracterizado por bajas tasas de supervivencia y limitadas opciones de tratamiento, especialmente en pacientes que no responden... MásHematología
ver canal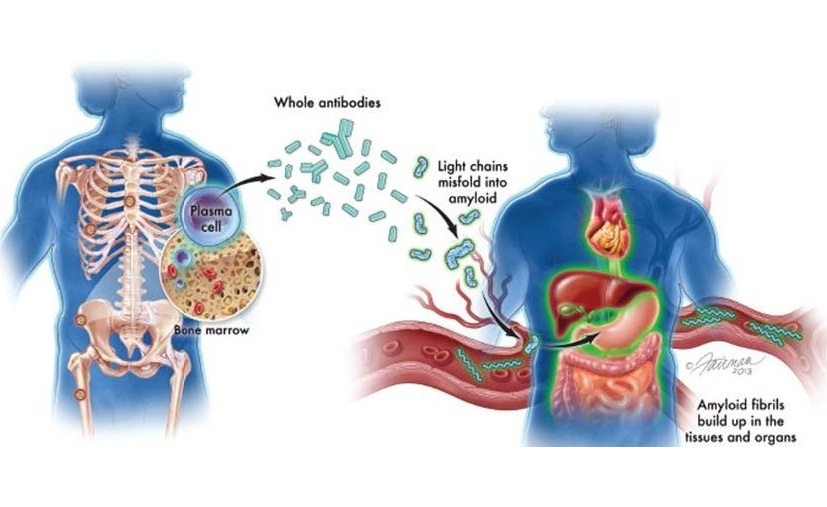
Nuevas directrices buscan mejorar diagnóstico de amiloidosis AL
La amiloidosis de cadenas ligeras (AL) es un trastorno poco común y potencialmente mortal de la médula ósea, en el que las proteínas amiloides anormales se acumulan en los órganos. Aproximadamente 3.... Más
Sistema automatizado de hemostasia ayuda a laboratorios a optimizar flujo de trabajo
Las secciones de hemostasia de alto volumen requieren una rápida respuesta, a la vez que gestionan repeticiones y pruebas reflejas. La manipulación de los tubos y las comprobaciones prea... MásInmunología
ver canalAnálisis de sangre identifica pacientes con cáncer pulmonar beneficiarios de fármaco de inmunoterapia
El cáncer de pulmón de células pequeñas (CPCP) es una enfermedad agresiva con opciones de tratamiento limitadas, e incluso las inmunoterapias recientemente aprobadas no benefician... Más
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... MásPatología
ver canal
IA mejora detección de mutaciones genéticas en diagnóstico del cáncer
La identificación precisa de mutaciones genéticas es fundamental para el diagnóstico del cáncer y la investigación genómica, pero los métodos actuales presentan... Más
Biopsia de piel ofrece nuevo método diagnóstico para enfermedades neurodegenerativas
La amiloidosis por transtiretina (ATTR) es una enfermedad rara, progresiva y muy agresiva causada por el plegamiento incorrecto de una proteína específica que se acumula en forma de filamentos... MásTecnología
ver canal
ADLM lanza programa de ciencia de datos único para profesionales de medicina de laboratorio
Los laboratorios clínicos generan miles de millones de resultados de pruebas cada año, lo que crea un cúmulo de datos con el potencial de facilitar pruebas más personalizadas,... MásTecnología de biosensores de aptámeros transforma detección de virus
La detección rápida y fiable de virus es esencial para controlar brotes, desde la gripe estacional hasta pandemias globales como la de COVID-19. Los métodos de diagnóstico ... MásIndustria
ver canal
Diasorin y Fisher Scientific firman acuerdo de distribución en EUA para plataforma POC molecular
Diasorin (Saluggia, Italia) ha firmado un acuerdo de distribución exclusivo con Fisher Scientific, parte de Thermo Fisher Scientific (Waltham, MA, EUA), para la plataforma molecular de punto de... Más