Sistema de microarrays basados en CRISPR permite pruebas masivas para patógenos virales
|
Por el equipo editorial de LabMedica en español Actualizado el 11 May 2020 |

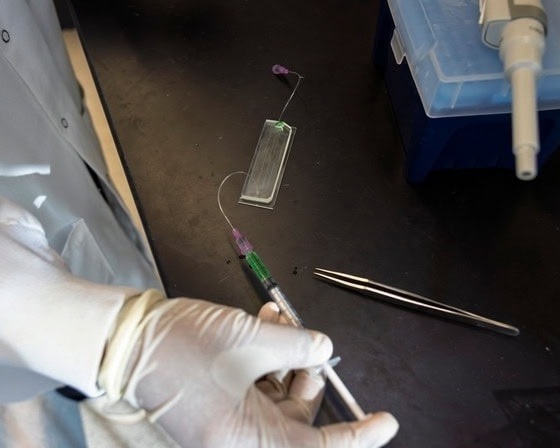
Imagen: Fotografía del chip de micropozos CARMEN (Fotografía cortesía de Michael James Butts)
Un novedoso sistema de diagnóstico microfluídico basado en ácidos nucleicos es capaz de detectar un virus específico de un catálogo de más de 160 patógenos humanos, incluido el coronavirus COVID-19, simultáneamente en más de mil muestras.
Para permitir la vigilancia de rutina y las aplicaciones de diagnóstico integrales, existe la necesidad de tecnologías de detección que se puedan ampliar para analizar muchas muestras y, al mismo tiempo, detectar múltiples patógenos individuales. Para lograr esto, los investigadores del Instituto Broad del MIT y Harvard (Cambridge, MA, EUA) desarrollaron una plataforma de diagnóstico llamada CARMEN (sigla en inglés para Reacciones Arregladas Combinadas para la Evaluación Multiplexada de Ácidos Nucleicos). El sistema CARMEN depende de gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR/Cas 13.
Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente y separadas entre sí) son segmentos de ADN procariótico que contienen repeticiones cortas de secuencias de bases. Cada repetición es seguida por segmentos cortos de “ADN espaciador” de exposiciones previas a un virus o plásmido bacteriano.
Los esfuerzos computacionales recientes para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida al ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación de ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, dirigido por el ARN contra el ARN y que tiene un gran potencial para aplicaciones precisas, robustas y escalables de orientación por el ARN contra el ARN.
La plataforma CARMEN consta de un chip de goma, un poco más grande que un teléfono inteligente, que contiene decenas de miles de micropozos diseñados para contener un par de gotitas del tamaño de nanolitros. Una gota contiene material genético viral de una muestra y la otra contiene reactivos de detección de virus.
La detección de ácidos nucleicos virales se realiza mediante una modificación del protocolo SHERLOCK. Este es un método para la detección de moléculas individuales de objetivos de ácido nucleico y significa desbloqueo específico del reportero enzimático de alta sensibilidad. Funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también se puede cuantificar. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple prueba de tira de papel, en equipos de laboratorio o para proporcionar una lectura electroquímica que se pueda leer con un teléfono móvil.
En la plataforma CARMEN, las gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR se autoorganizan en la matriz de micropozos para emparejarse con las gotas de muestras amplificadas, analizando cada muestra contra cada ARN CRISPR (ARNcr) en replicado. La combinación de detección CARMEN y Cas13 (CARMEN-Cas13) permitió realizar pruebas robustas de más de 4.500 pares de ARNc-objetivo en una sola matriz. El protocolo completo, desde la extracción de ARN hasta los resultados, requirió menos de ocho horas.
Empleando el método CARMEN-Cas13, los investigadores desarrollaron un ensayo multiplexado que simultáneamente diferenciaba 169 virus asociados con humanos con más de 10 secuencias genómicas publicadas e incorporaba rápidamente un ARNcr adicional para detectar el agente causante del coronavirus pandémico COVID-19. CARMEN-Cas13 permitió además el subtipo integral de cepas de influenza A y la identificación multiplexada de docenas de mutaciones de resistencia a los medicamentos contra el VIH.
“Este enfoque miniaturizado para el diagnóstico es eficiente en recursos y fácil de implementar”, dijo el coautor principal, el Dr. Paul Blainey, profesor asociado de ingeniería biológica en el Instituto de Tecnología de Massachusetts. “Las nuevas herramientas requieren creatividad e innovación, y con estos avances en química y microfluídica, estamos entusiasmados con el potencial de CARMEN a medida que la comunidad trabaja para vencer a la COVID-19 y las futuras amenazas de enfermedades infecciosas”.
El método CARMEN-Cas13 se describió en la edición en línea del 29 de abril de 2020 de la revista Nature.
Enlace relacionado:
Instituto Broad del MIT y Harvard
Para permitir la vigilancia de rutina y las aplicaciones de diagnóstico integrales, existe la necesidad de tecnologías de detección que se puedan ampliar para analizar muchas muestras y, al mismo tiempo, detectar múltiples patógenos individuales. Para lograr esto, los investigadores del Instituto Broad del MIT y Harvard (Cambridge, MA, EUA) desarrollaron una plataforma de diagnóstico llamada CARMEN (sigla en inglés para Reacciones Arregladas Combinadas para la Evaluación Multiplexada de Ácidos Nucleicos). El sistema CARMEN depende de gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR/Cas 13.
Los CRISPR (repeticiones palindrómicas cortas agrupadas regularmente y separadas entre sí) son segmentos de ADN procariótico que contienen repeticiones cortas de secuencias de bases. Cada repetición es seguida por segmentos cortos de “ADN espaciador” de exposiciones previas a un virus o plásmido bacteriano.
Los esfuerzos computacionales recientes para identificar nuevos sistemas CRISPR descubrieron un nuevo tipo de enzima dirigida al ARN, Cas13. La diversa familia Cas13 contiene al menos cuatro subtipos conocidos, incluidos Cas13a (anteriormente C2c2), Cas13b, Cas13c y Cas13d. Se demostró que Cas13a se une y escinde el ARN, protegiendo a las bacterias de los fagos de ARN y sirviendo como una plataforma poderosa para la manipulación de ARN. Se sugirió que Cas13a podría funcionar como parte de un sistema CRISPR/Cas versátil, dirigido por el ARN contra el ARN y que tiene un gran potencial para aplicaciones precisas, robustas y escalables de orientación por el ARN contra el ARN.
La plataforma CARMEN consta de un chip de goma, un poco más grande que un teléfono inteligente, que contiene decenas de miles de micropozos diseñados para contener un par de gotitas del tamaño de nanolitros. Una gota contiene material genético viral de una muestra y la otra contiene reactivos de detección de virus.
La detección de ácidos nucleicos virales se realiza mediante una modificación del protocolo SHERLOCK. Este es un método para la detección de moléculas individuales de objetivos de ácido nucleico y significa desbloqueo específico del reportero enzimático de alta sensibilidad. Funciona amplificando secuencias genéticas y programando una molécula CRISPR para detectar la presencia de una firma genética específica en una muestra, que también se puede cuantificar. Cuando encuentra esas firmas, la enzima CRISPR se activa y libera una señal robusta. Esta señal se puede adaptar para trabajar en una simple prueba de tira de papel, en equipos de laboratorio o para proporcionar una lectura electroquímica que se pueda leer con un teléfono móvil.
En la plataforma CARMEN, las gotas de nanolitros que contienen reactivos de detección de ácido nucleico basados en CRISPR se autoorganizan en la matriz de micropozos para emparejarse con las gotas de muestras amplificadas, analizando cada muestra contra cada ARN CRISPR (ARNcr) en replicado. La combinación de detección CARMEN y Cas13 (CARMEN-Cas13) permitió realizar pruebas robustas de más de 4.500 pares de ARNc-objetivo en una sola matriz. El protocolo completo, desde la extracción de ARN hasta los resultados, requirió menos de ocho horas.
Empleando el método CARMEN-Cas13, los investigadores desarrollaron un ensayo multiplexado que simultáneamente diferenciaba 169 virus asociados con humanos con más de 10 secuencias genómicas publicadas e incorporaba rápidamente un ARNcr adicional para detectar el agente causante del coronavirus pandémico COVID-19. CARMEN-Cas13 permitió además el subtipo integral de cepas de influenza A y la identificación multiplexada de docenas de mutaciones de resistencia a los medicamentos contra el VIH.
“Este enfoque miniaturizado para el diagnóstico es eficiente en recursos y fácil de implementar”, dijo el coautor principal, el Dr. Paul Blainey, profesor asociado de ingeniería biológica en el Instituto de Tecnología de Massachusetts. “Las nuevas herramientas requieren creatividad e innovación, y con estos avances en química y microfluídica, estamos entusiasmados con el potencial de CARMEN a medida que la comunidad trabaja para vencer a la COVID-19 y las futuras amenazas de enfermedades infecciosas”.
El método CARMEN-Cas13 se describió en la edición en línea del 29 de abril de 2020 de la revista Nature.
Enlace relacionado:
Instituto Broad del MIT y Harvard
Últimas Microbiología noticias
- Nueva prueba mide eficacia de antibióticos para eliminar bacterias
- Nuevas normas para antimicrobianos atienden tuberculosis para optimizar diagnóstico
- Método de diagnóstico de ITU ofrece resultados de resistencia a antibióticos 24 horas antes
- Avances en análisis microbiano mejora predicción de enfermedades
- Método de diagnóstico en sangre identifica infecciones IVRI pediátricas
- Prueba de diagnóstico rápido es estándar de oro para detección de sepsis
- Prueba rápida de tuberculosis POC proporciona resultados en 15 minutos
- Ensayo rápido identifica patógenos de infecciones sanguíneas directamente en muestras de pacientes
- Firmas moleculares basadas en sangre para permitir un diagnóstico rápido de TBEP
- Análisis sanguíneo rápido diagnostica infecciones infantiles potencialmente mortales
- Paneles entéricos de alto rendimiento detectan múltiples infecciones bacterianas gastrointestinales
- Prueba rápida no invasiva utiliza huella de azúcar para detectar infecciones por hongos
- Dispositivo de diagnóstico rápido de sepsis para atención crítica personalizada a pacientes de UCI
- Plataforma microfluídica evalúa función de neutrófilos en pacientes con sepsis
- Nuevo método diagnóstico confirma sepsis de forma más temprana
- Nuevos marcadores podrían predecir riesgo de infección grave por clamidia
Canales
Química Clínica
ver canal
Análisis sanguíneo podría predecir e identificar recaídas tempranas en pacientes con mieloma
El mieloma múltiple es un cáncer incurable de la médula ósea. Si bien muchos pacientes viven más de una década tras el diagnóstico, una proporción... Más
Sistema compacto de imágenes Raman detecta señales tumorales sutiles
El diagnóstico preciso del cáncer a menudo depende de una tinción tisular laboriosa y de una revisión patológica experta, lo que puede retrasar los resultados y limitar... MásDiagnóstico Molecular
ver canal
Análisis sanguíneo predice enfermedad de Crohn con anterioridad a síntomas
La enfermedad de Crohn es un trastorno inflamatorio crónico del tracto gastrointestinal que causa síntomas digestivos persistentes, dolor y fatiga, lo que a menudo requiere tratamiento de por vida.... Más
Pruebas de ADN de pólipos colorrectales mejoran comprensión de riesgos hereditarios
El cáncer colorrectal es uno de los cánceres más comunes en los países occidentales, y los factores hereditarios intervienen en aproximadamente el 5-10 % de los casos, especialmente... MásHematología
ver canal
Algoritmo de IA distingue eficazmente subtipos de alfa talasemia
La alfa talasemia afecta a millones de personas en todo el mundo y es especialmente común en regiones como el Sudeste Asiático, donde las tasas de portadores pueden alcanzar niveles extremadamente... Más
Pruebas de MRD podrían predecir supervivencia en pacientes con leucemia
La leucemia mieloide aguda es un cáncer sanguíneo agresivo que altera la producción normal de células sanguíneas y suele recaer incluso después de un tratamiento intensivo. Actualmente, los médicos carecen... MásInmunología
ver canal
Enfoque de secuenciación genómica completa identifica pacientes con cáncer beneficiarios con inhibidores de PARP
Las terapias dirigidas contra el cáncer, como los inhibidores de PARP, pueden ser muy eficaces, pero solo en pacientes cuyos tumores presentan defectos específicos en la reparación del ADN.... Más
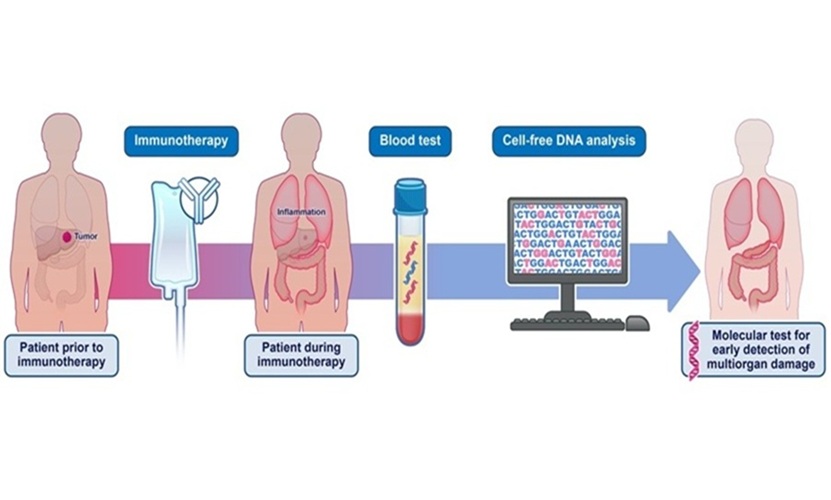
Biopsia líquida ultrasensible demuestra eficacia para predecir respuesta a inmunoterapia
La inmunoterapia ha transformado el tratamiento del cáncer, pero solo una pequeña proporción de pacientes experimenta un beneficio duradero, con tasas de respuesta que a menudo se... MásPatología
ver canal
Imágenes ópticas sin etiquetas impulsadas por IA identifican cáncer de tiroides durante cirugía
El cáncer de tiroides es el cáncer endocrino más común, y su creciente tasa de detección ha incrementado el número de pacientes sometidos a cirugía.... Más
Método de aprendizaje profundo mejora diagnóstico del cáncer
Identificar la invasión vascular es fundamental para determinar la agresividad de un cáncer. Sin embargo, hacerlo de forma fiable puede resultar difícil con los flujos de trabajo ... MásTecnología
ver canal
Sensores generados por IA abren caminos para detección temprana del cáncer
Los cánceres son mucho más fáciles de tratar cuando se detectan a tiempo; sin embargo, muchos tumores permanecen invisibles hasta que están avanzados o han reaparecido tras... Más
Prueba sanguínea pionera detecta cáncer de pulmón mediante imágenes infrarrojas
Detectar el cáncer en sus etapas tempranas y rastrear su respuesta al tratamiento sigue siendo un gran desafío, especialmente cuando la cantidad de células cancerosas en el torrente... MásIndustria
ver canal
WHX Labs Dubai reunirá expertos mundiales en resistencia a antimicrobianos en Cumbre inaugural de líderes de RAM
La World Health Expo (WHX) Labs de Dubai (anteriormente Medlab Middle East), que se llevará a cabo en el Dubai World Trade Centre del 10 al 13 de febrero, abordará la creciente amenaza mundial... Más