Cepa nueva de bacterias causa la escarlatina
|
Por el equipo editorial de LabMedica en español Actualizado el 02 Oct 2019 |
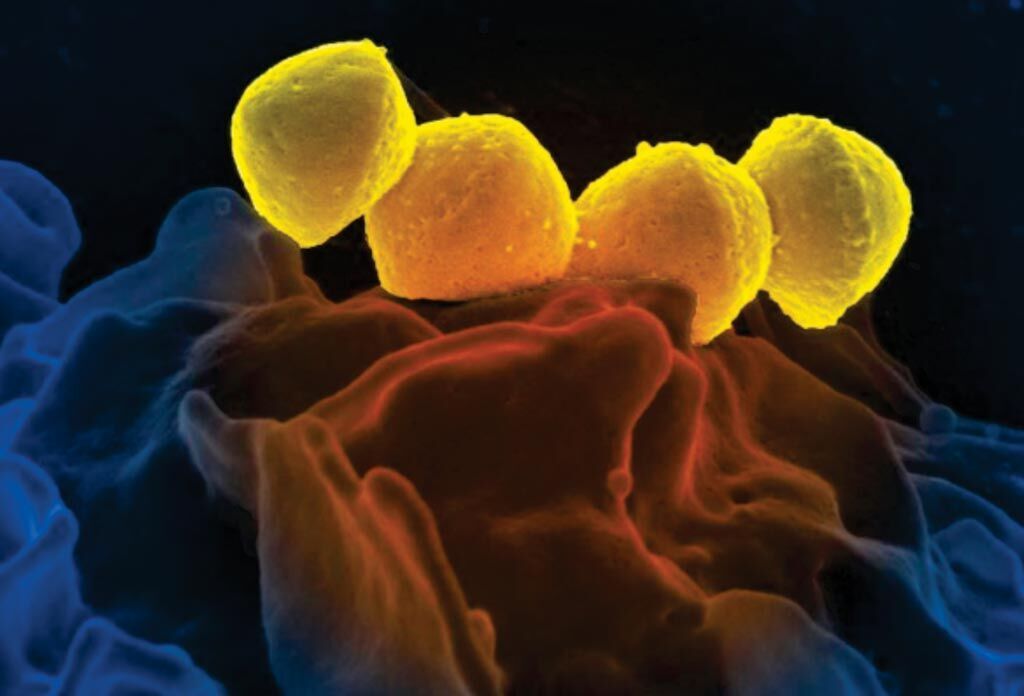
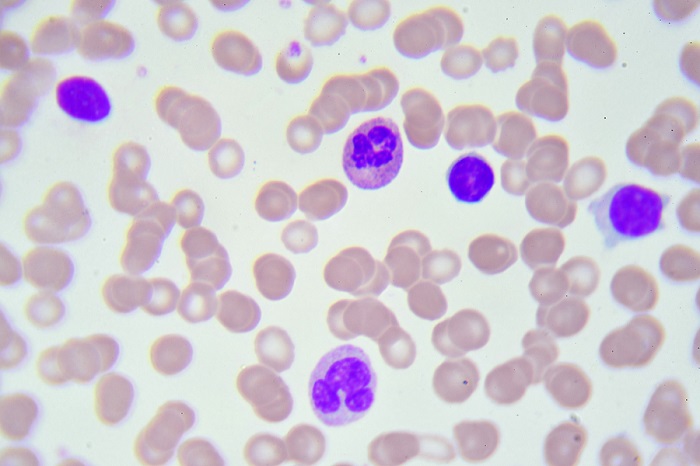
Imagen: Esta imagen microscópica electrónica de barrido, coloreada digitalmente (SEM) muestra cuatro bacterias Streptococcus del grupo A (GAS) de color amarillo, que se encuentran sobre la superficie de un glóbulo blanco humano (WBC), denominado neutrófilo (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los Estados Unidos).
La escarlatina es un exantema clásico de la infancia causado por la bacteria Streptococcus pyogenes (estreptococo del grupo A). Los síntomas, que afectan a los niños pequeños, incluyen temperatura alta, dolor de garganta y una erupción de color rojo rosado que se siente como papel de lija.
La escarlatina es causada por toxinas liberadas por la bacteria Streptococcus pyogenes, también conocida como Strep A, y los casos siguen un patrón estacional que alcanza su punto máximo entre marzo y mayo. La escarlatina se trata fácilmente con antibióticos. En el Reino Unido, los casos de infecciones invasivas causadas por la misma bacteria aumentaron en 2016 en comparación con los cinco años anteriores.
Los especialistas en enfermedades infecciosas del Colegio Imperial de Londres (Londres, Reino Unido) y sus colegas, identificaron las cepas de Strep A que causan infecciones en Londres y más ampliamente en Inglaterra y Gales, según lo definido por la presencia del tipo de gen emm. Descubrieron que el aumento inicial de la escarlatina en 2014 en Londres se asoció con los tipos de cepa Strep A emm3 y emm4. Sin embargo, durante la primavera de 2015 y 2016, las cepas emm1 se volvieron dominantes entre las infecciones de garganta.
En la primavera de 2014, solo el 5% (5/96) de los aislados de la bacteria recolectada en el noroeste de Londres eran cepas emm1, pero para 2015, esta cifra aumentó al 19% (28/147). En 2016, emm1 se convirtió en la cepa más frecuente con un 33% (47/144 aislamientos). Para investigar más a fondo los aislamientos emm1, los científicos secuenciaron los genomas de los 135 aislamientos no invasivos de la bacteria emm1, recolectados en el noroeste de Londres entre 2009 y 2016, y todos los 552 aislamientos invasivos emm1 recolectados en Inglaterra y Gales durante los picos de la enfermedad estacional entre 2013 y 2016, y los compararon entre sí. También evaluaron la cantidad de toxina producida por diferentes cepas de emm1.
Los investigadores descubrieron que la mayoría de las cepas emm1 de 2015 y 2016 eran un clon emm1 distinto y separable, al que se refirieron como M1UK. El clon tenía 27 mutaciones únicas y se asoció con una producción significativamente mayor de la exotoxina pirógena estreptocócica toxina A (SpeA). Esta toxina desencadena la escarlatina y puede contribuir a la faringitis estreptocócica A y a algunas infecciones invasivas. El análisis confirmó que M1UK produce nueve veces más toxina que otras cepas emm1 (190 ng/mL en comparación con 21 ng/mL). En 2010 ya estaba presente en Inglaterra y para 2016, M1UK representaba el 84% de todos los genomas emm1 analizados en Inglaterra y Gales.
Shiranee Sriskandan, FRCP, profesora de enfermedades infecciosas y autora principal del estudio, dijo: “El clon bacteriano definido que descubrimos parece estar limitado en gran medida al Reino Unido, pero el hecho de que se hayan identificado dos ejemplos de estos clones en otros lugares sugiere que el clon tiene el potencial de propagación internacional y que ya puede estar presente en otros países. Sin embargo, también es posible que el linaje no dure. En el pasado, aparecieron algunos linajes que luego desaparecieron rápidamente”. El estudio fue publicado el 10 de septiembre de 2019 en la revista The Lancet Infectious Diseases.
Enlace relacionado:
Colegio Imperial de Londres
La escarlatina es causada por toxinas liberadas por la bacteria Streptococcus pyogenes, también conocida como Strep A, y los casos siguen un patrón estacional que alcanza su punto máximo entre marzo y mayo. La escarlatina se trata fácilmente con antibióticos. En el Reino Unido, los casos de infecciones invasivas causadas por la misma bacteria aumentaron en 2016 en comparación con los cinco años anteriores.
Los especialistas en enfermedades infecciosas del Colegio Imperial de Londres (Londres, Reino Unido) y sus colegas, identificaron las cepas de Strep A que causan infecciones en Londres y más ampliamente en Inglaterra y Gales, según lo definido por la presencia del tipo de gen emm. Descubrieron que el aumento inicial de la escarlatina en 2014 en Londres se asoció con los tipos de cepa Strep A emm3 y emm4. Sin embargo, durante la primavera de 2015 y 2016, las cepas emm1 se volvieron dominantes entre las infecciones de garganta.
En la primavera de 2014, solo el 5% (5/96) de los aislados de la bacteria recolectada en el noroeste de Londres eran cepas emm1, pero para 2015, esta cifra aumentó al 19% (28/147). En 2016, emm1 se convirtió en la cepa más frecuente con un 33% (47/144 aislamientos). Para investigar más a fondo los aislamientos emm1, los científicos secuenciaron los genomas de los 135 aislamientos no invasivos de la bacteria emm1, recolectados en el noroeste de Londres entre 2009 y 2016, y todos los 552 aislamientos invasivos emm1 recolectados en Inglaterra y Gales durante los picos de la enfermedad estacional entre 2013 y 2016, y los compararon entre sí. También evaluaron la cantidad de toxina producida por diferentes cepas de emm1.
Los investigadores descubrieron que la mayoría de las cepas emm1 de 2015 y 2016 eran un clon emm1 distinto y separable, al que se refirieron como M1UK. El clon tenía 27 mutaciones únicas y se asoció con una producción significativamente mayor de la exotoxina pirógena estreptocócica toxina A (SpeA). Esta toxina desencadena la escarlatina y puede contribuir a la faringitis estreptocócica A y a algunas infecciones invasivas. El análisis confirmó que M1UK produce nueve veces más toxina que otras cepas emm1 (190 ng/mL en comparación con 21 ng/mL). En 2010 ya estaba presente en Inglaterra y para 2016, M1UK representaba el 84% de todos los genomas emm1 analizados en Inglaterra y Gales.
Shiranee Sriskandan, FRCP, profesora de enfermedades infecciosas y autora principal del estudio, dijo: “El clon bacteriano definido que descubrimos parece estar limitado en gran medida al Reino Unido, pero el hecho de que se hayan identificado dos ejemplos de estos clones en otros lugares sugiere que el clon tiene el potencial de propagación internacional y que ya puede estar presente en otros países. Sin embargo, también es posible que el linaje no dure. En el pasado, aparecieron algunos linajes que luego desaparecieron rápidamente”. El estudio fue publicado el 10 de septiembre de 2019 en la revista The Lancet Infectious Diseases.
Enlace relacionado:
Colegio Imperial de Londres
Últimas Diagnóstico Molecular noticias
- Firma genética en recién nacidos predice sepsis antes de que aparezcan síntomas
- Integración de marcadores proteicos predice resultados de salud en pacientes con enfermedad renal crónica
- Prueba rápida de sangre mediante punción digital detecta sífilis activa en el punto de atención
- Análisis de orina podrían revelar signos tempranos de cáncer y otras enfermedades
- Sistema de PCR inteligente con IA revolucionará diagnóstico clínico
- Análisis de sangre identifica mujeres en trabajo de parto con riesgo de preeclampsia
- Prueba en papel POC podría diagnosticar cáncer en la cabecera
- Prueba de sepsis demuestra utilidad clínica en poblaciones heterogéneas de pacientes gravemente enfermos
- Prueba para múltiples cánceres mide respuesta del huésped al desarrollo del tumor
- Prueba basada en microARN identifica 12 cánceres en cualquier etapa
- Nuevo análisis de sangre mide simultáneamente más de 100 biomarcadores de enfermedad de Alzheimer

- Análisis de sangre diagnostica autismo relacionado con autoanticuerpos maternos
- Prueba de sangre de medicina de precisión predice la enfermedad cardíaca
- Prueba de PCR guía terapia para Alzheimer
- Análisis de sangre predice supervivencia en pacientes con cáncer de próstata metastásico
- Prueba de sepsis en POC ofrece resultados rápidos y precisos en entornos de cuidados intensivos
Canales
Química Clínica
ver canal
Dispositivo de pruebas de saliva predice la insuficiencia cardíaca en 15 minutos
La insuficiencia cardíaca es una enfermedad grave en la que el músculo cardíaco no puede bombear suficiente sangre rica en oxígeno a todo el cuerpo. Se considera una de las... Más
Herramienta de diagnóstico identifica múltiples condiciones de salud a partir de una sola gota de sangre
La espectroscopía infrarroja, un método que utiliza luz infrarroja para estudiar la composición molecular de las sustancias, ha sido una herramienta fundamental en la química durante décadas.... Más
Un analizador integrado de química e inmunoensayo con extenso menú de ensayos ofrece flexibilidad, escalabilidad y conmutabilidad de datos
A medida que los sistemas sanitarios mundiales cambian cada vez más hacia modelos operativos de laboratorios en red para mejorar la eficiencia y el acceso de los pacientes, existe una mayor necesidad... MásDiagnóstico Molecular
ver canal
Firma genética en recién nacidos predice sepsis antes de que aparezcan síntomas
La sepsis neonatal, que se produce debido a la respuesta anormal del organismo a una infección grave durante los primeros 28 días de vida, provoca aproximadamente 200.000 muertes en todo... Más.jpeg)
Integración de marcadores proteicos predice resultados de salud en pacientes con enfermedad renal crónica
Los intentos anteriores de descubrir nuevos biomarcadores renales como factores de riesgo para la progresión de la enfermedad renal crónica (ERC) se han centrado generalmente en la evaluación... MásHematología
ver canal.jpg)
Sistema de diagnóstico de hemograma completo y sepsis busca resultados más rápidos, tempranos y fáciles
Cada hora es crucial para proteger a los pacientes de las infecciones, pero actualmente existen herramientas limitadas para ayudar a un diagnóstico temprano antes de que los pacientes lleguen al hospital.... Más
Nuevo grupo sanguíneo descubierto ayudará a identificar y tratar a pacientes
El antígeno del grupo sanguíneo AnWj, un marcador de superficie descubierto en 1972, ha sido un misterio en cuanto a su origen genético, hasta ahora. La causa más común... MásInmunología
ver canal
Biomarcador podría predecir respuesta a inmunoterapia en cáncer de hígado
Hasta hace poco, los pacientes con diagnóstico de carcinoma hepatocelular contaban con opciones de tratamiento limitadas, ya que las terapias existentes prolongaban la vida solo unos pocos meses.... Más
Prueba epigenética podría determinar eficacia de nuevos tratamientos de inmunoterapia contra mieloma múltiple
El mieloma múltiple es un cáncer de la sangre que afecta principalmente a personas mayores de sesenta años y su incidencia aumenta a medida que la población envejece.... MásPatología
ver canal.jpg)
Método de diagnóstico por imágenes facilita diagnósticos precisos para cánceres de cabeza y cuello
Los cánceres de cabeza y cuello, aunque se consideran poco frecuentes, representan una parte importante de los casos de cáncer y han experimentado un notable aumento en los últimos 30 años.... Más
Medición más rápida de huella vibratoria de moléculas avanza en el diagnóstico biomédico
Identificar diferentes tipos de moléculas y células es un proceso vital tanto en la ciencia básica como en la aplicada. La espectroscopia Raman es una técnica de medición... MásPrueba clínica de anticuerpos detecta rápidamente incluso niveles bajos de infecciones parasitarias comunes
Las enfermedades tropicales desatendidas abarcan una variedad de afecciones que afectan a millones de personas en todo el mundo, principalmente en regiones empobrecidas, pero a menudo carecen del enfoque... MásTecnología
ver canal
Métodos no invasivos detectan exposición al plomo de forma más rápida, sencilla y precisa en POC
La exposición al plomo puede afectar negativamente la salud de múltiples maneras, provocando daños en el cerebro y el sistema nervioso central, retrasos en el desarrollo y el crecimiento,... Más
Prueba no invasiva detecta malaria sin muestra sanguínea
La malaria sigue siendo un problema de salud mundial importante, con aproximadamente 250 millones de casos y más de 600.000 muertes registradas anualmente. Casi la mitad de la población mundial... Más
Dispositivo portátil detecta cáncer colorrectal y de próstata en una hora
La detección temprana de biomarcadores de cáncer antes de que la enfermedad progrese mejora significativamente las posibilidades de supervivencia del paciente. Las demoras en las pruebas,... Más
Tecnología Light-AI para diagnóstico del cáncer eliminaría necesidad de extracciones de sangre y biopsias tradicionales
Existen numerosos casos de perros que ladran tanto a sus dueños que estos sospechan que algo anda mal, lo que los lleva a visitar a un médico y, finalmente, a recibir un diagnóstico... MásIndustria
ver canal
Beckman Coulter se asocia con BioPorto para distribución global de pruebas NGAL para lesiones renales agudas
La lesión renal aguda (LRA) es un episodio repentino de insuficiencia o daño renal que puede ocurrir en cuestión de horas o días. Esta afección provoca la acumulación... Más_1.jpg)







.jpg)