Método molecular eficiente detecta las leptospiras patógenas
Por el equipo editorial de LabMedica en español
Actualizado el 27 Jul 2015
El diagnóstico de la leptospirosis, que se basa principalmente en los métodos anticuados de serotipificación, es particularmente difícil debido a que presenta síntomas no específicos que son compartidos con otras enfermedades febriles, lo cual conduce a menudo a un diagnóstico erróneo.Actualizado el 27 Jul 2015
Las especies patógenas de Leptospira producen una enfermedad zoonótica prevalente pero aún descuidada, con complicaciones desde leves hasta potencialmente mortales en una variedad de animales susceptibles y en los seres humanos. Con frecuencia, no se inicia el tratamiento antimicrobiano durante la infección temprana buscando evitar complicaciones más graves de la infección diseminada, debido en gran parte a la falta de pruebas de diagnóstico eficientes.
Unos científicos de la Universidad de Maryland, (College Park, MD, EUA) y sus colegas recogieron muestras de sangre de 25 pacientes que participaron durante un programa de vigilancia activa de la leptospirosis en un hospital estatal de Salvador (Brasil), de junio a septiembre de 2013 y se recogieron las muestras durante su hospitalización inicial. Todos los pacientes fueron examinados para buscar leptospirosis usando los métodos descritos previamente con hemocultivo y una prueba de aglutinación microscópica (MAT), así como una reacción en cadena de polimerasa cuantitativa (qPCR) del ADN. Los pacientes confirmados fueron positivos para al menos una de las tres pruebas mencionadas. Se determinó que 22 de los 25 pacientes tenían leptospirosis confirmada por el laboratorio.
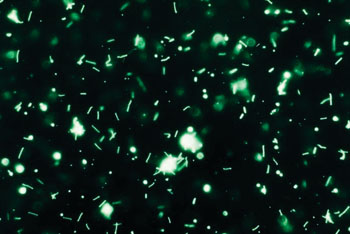
Hay regiones específicas de las moléculas del ácido ribonucleico de los ribosomas 16S (rARN) de las leptospiras que constituyen un objetivo diagnóstico novedoso y eficiente para la detección basada en PCR de serovares patógenas de la Leptospira. Todas las reacciones de qPCR se realizaron con el sistema CFX96 para detección por PCR en tiempo real (Bio-Rad Laboratories, Inc.; Hércules, CA, EUA). La prueba para diagnóstico con sangre humana adicionada fue al menos 100 veces más sensible que las correspondientes pruebas de PCR cuantitativa basadas en el ADN de la leptospira, dirigidas a la misma secuencia de nucleótidos 16S de las moléculas de ARN y ADN. La sensibilidad y la especificidad de la prueba de ARN comparada con la confirmación del laboratorio de leptospirosis humana en las muestras clínicas fueron de 64 % y 100 %, respectivamente, superiores a los de una prueba para detección del ADN establecida en paralelo.
El equipo descubrió que las transcripciones de 16S siguen siendo muy estables ex vivo, incluyendo muestras de sangre humana almacenadas sin tratar, lo cual resalta aún más su uso para la detección clínica de la L. interrogans. Juntos, estos estudios ponen de relieve una utilidad novedosa de los objetivos de ARN y específicamente de los rARN 16S para el desarrollo de modalidades basadas en PCR para el diagnóstico de la leptospirosis humana y que también pueden servir como paradigma para la detección de otros patógenos bacterianos para los cuales sería conveniente contar con un diagnóstico precoz. El estudio fue publicado el 19 de junio de 2015 en la revista Public Library of Science ONE.
Enlaces relacionados:
University of Maryland
Bio-Rad Laboratories