Bacterias como supermicrobios resistentes a los antibióticos
Por el equipo editorial de LabMedica en español
Actualizado el 23 Apr 2015
Las enterobacterias patógenas, incluyendo Escherichia coli y Klebsiella pneumoniae, son las principales causas de infecciones resistente a múltiples antibióticos (MDR) en los hospitales de todo el mundo y éstos patógenos han demostrado recientemente que han adquirido resistencia a los carbapenémicos.Actualizado el 23 Apr 2015
Se han identificado dos genes que confieren resistencia contra una clase particularmente fuerte de antibióticos y que pueden ser compartidos fácilmente entre una familia de bacterias responsables de una parte importante de infecciones respiratorias y urinarias adquiridas en los hospitales.
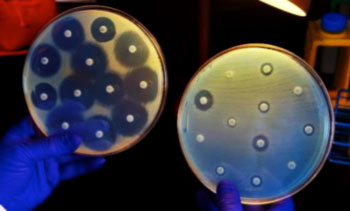
Imagen: La placa de la derecha fue inoculada con una Enterobacteria resistente al Carbapenem (CRE) que resultó ser resistente a todos los antibióticos ensayados; las bacterias en la placa izquierda son susceptibles a los antibióticos en los discos (Fotografía cortesía de James Gathany).
Los científicos de la Facultad de Medicina de la Universidad de Washington (St. Louis, MO, EUA) recolectaron 450 aislados bacterianos, incluyendo 195 Enterobacterias en Pakistán, durante febrero de 2012 hasta marzo de 2013. Los investigadores seleccionaron al azar 55 aislados de Enterobacterias para hacerles la secuenciación de todo el genoma. Luego seleccionaron 23 aislamientos de muestras obtenidas de pacientes en los EUA entre enero de 2010 y junio de 2013, en el Hospital Judío Barnes (St. Louis, MO, EUA), que tenían una proporción similar de susceptibilidad y resistencia a los β-lactámicos a los recolectados en Pakistán para la secuenciación.
Los investigadores también secuenciaron una porción especial de material genético de las bacterias, llamados plásmidos. La mayor parte del ADN de una bacteria se encuentra en su cromosoma, pero las bacterias también tienen muchos pedazos adicionales, más pequeños y circulares de ADN conocidos como plásmidos que fácilmente pueden pasar de una cepa bacteriana a otra. Un plásmido es como un camión de reparto de genes bacterianos; es la forma principal como los genes de resistencia a los antibióticos se difunden entre las bacterias.
La variedad de cepas que descubrieron que codificaban la carbpenemasa de Klebsiella pneumoniae (KPC) y la metalo-β-Lacatamasa-1 de Nueva Delhi (NDM-1) fue consistente con la evidencia existente de que la transferencia horizontal de genes (HGT) es un factor importante en su propagación. Ellos identificaron algunos casos claves en los que el plásmido que llevaba NDM-1 o KPC eran casi idénticos, lo que significa que esto fácilmente podría facilitar la propagación de la resistencia a los antibióticos entre las bacterias que causan enfermedades que se encuentran en los EUA y en el sur de Asia.
Gautam Dantas, PhD, profesor asociado de patología e inmunología y autor principal del estudio, dijo: “Nuestros resultados también sugieren que va a ser más fácil para las cepas de estas bacterias, que aún no son resistentes, captar un gen que les permite sobrevivir el tratamiento con carbapenem. Los carbapenémicos son uno de nuestros últimos recursos para el tratamiento de infecciones bacterianas, los que utilizamos cuando nada más funciona. A medida que las formas resistentes a los medicamentos de Enterobacterias se vuelvan más generalizadas, las probabilidades aumentarán de transmitir una de estas súperbacterias a un amigo con un sistema inmunológico debilitado que realmente puede ser afectado por ellas”. El estudio fue publicado en línea en marzo de 2015 para la edición de junio de 2015 de la revista Emerging Infectious Diseases.
Enlaces relacionados:
Escuela de Medicina de la Universidad Washington
Hospital Barnes Jewish