Herramienta detecta patógenos bacterianos más comunes
Por el equipo editorial de LabMedica en español
Actualizado el 12 Jan 2015
Se desarrolló un análisis con microarrays para detectar más de 60 de las especies bacterianas más comunes de bacterias Gram-positivas y Gram-negativas, incluyendo la detección de los genes de resistencia, mecA, vanA y vanB, directamente a partir de muestras osteoarticulares.Actualizado el 12 Jan 2015
Se hizo una evaluación del desempeño de la plataforma de diagnóstico en muestras principalmente positivas en los cultivos mediante la comparación de sus resultados con el cultivo bacteriano y una reacción de cadena de la polimerasa bacteriana (PCR), casera, de amplio alcance. La evaluación extensa incluía no sólo muestras de origen osteoarticular, sino también muestras de otros sitios estériles principales del cuerpo.
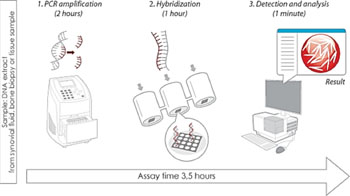
Imagen: El flujo de trabajo del análisis mediante microarrays Prove-it Bone&Joint (Fotografía cortesía de Mobidiag).
Microbiólogos clínicos en el Hospital Universitario de Basilea (Suiza) recogieron muestras clínicas entre enero y mayo de 2012 de los pacientes hospitalizados. De 200 muestras, 120 fueron obtenidas usando un aparato osteoarticular (65 biopsias, 42 implantes, y 13 aspirados), y otras 80 muestras de sitios del cuerpo como pleura, ascitis, líquido cefalorraquídeo (LCR), pulmón, páncreas, vesícula biliar, válvulas cardiacas así como prótesis intracardíacas o de mama (40 biopsias, 27 aspirados, 6 implantes, 5 muestras de LCR y otras 2).
Se realizaron cultivos aeróbicos y de bacterias anaerobias siguiendo los protocolos estándar incluyendo cultivo de enriquecimiento mediante medio de tioglicolato. Se realizó una PCR bacteriana de amplio alcance y las muestras que mostraron productos de PCR visibles, fueron secuenciados en el Secuenciador Capilar ABI 3130 (Applied Biosystems, Foster City, EUA). Los extractos de ADN fueron analizados con el ensayo Prove-it Bone & Joint (Mobidiag; Helsinki, Finlandia).
En 129/176 (73.3%) se aisló un microorganismo (muestras monomicrobianas), y en 47/176 (26,7%), se aisló más de un microorganismo (muestras polimicrobianas). La sensibilidad del análisis Prove-it Bone&Joint en muestras osteoarticulares monomicrobianas fue de 62,6% y en no-osteoarticulares, fue del 52,2%. Cuando se incluyeron solamente las muestras que contenían especies cubiertas por el panel en el cálculo, las sensibilidades de ambos tipos de muestras fueron 72,2% y 68,6%, respectivamente. El ensayo Prove-it predijo correctamente la resistencia/susceptibilidad a la meticilina en todas las 66 muestras en las que se realizó la prueba de susceptibilidad fenotípica. En dos muestras, se detectó una señal positiva para Staphylococcus aureus y se detectó el gen mecA. Una muestra contenía una cepa verdaderamente resistente a la meticilina de S. aureus (SARM), mientras que la otra muestra contenía S. aureus sensible a la meticilina y Staphylococcus epidermidis resistente a la meticilina.
Los autores concluyeron que el ensayo Prove-it Bone&Joint es una herramienta de diagnóstico muy rápida que proporciona información precoz sobre la identidad de los patógenos más comunes presentes no sólo en las muestras osteoarticulares, sino también en otras muestras estériles primarias a la espera de los resultados definitivos del cultivo. Aunque este ensayo sólo puede detectar un número limitado de especies bacterianas presentes en el panel de Prove-it, puede servir como una alternativa para la PCR bacteriana de alcance para los laboratorios que no son capaces de realizar este análisis molecular basado en secuencias. El estudio fue publicado en la edición de diciembre de 2014 de la revista Journal of Microbiological Methods.
Enlaces relacionados:
University Hospital Basel
Applied Biosystems
Mobidiag