Análisis diferencia especies coendémicas de Leishmania
Por el equipo editorial de LabMedica en español
Actualizado el 02 Oct 2014
En un estudio de muestras clínicas de pacientes del sureste de México, un análisis con PCR-RFLP fue eficaz para la diferenciación de las especies co-endémicas de Leishmania, lo cual permite tener diagnósticos más refinados y tratamientos más adecuados para los pacientes de una población dada, con diferentes formas de leishmaniosis del Nuevo Mundo.Actualizado el 02 Oct 2014
La leishmaniosis cutánea (LC) americana incluye: LC localizada (LLC) causada por la L. (L.) mexicana; LC difusa (DLC) causada por la L. (L.) amazonensis, la Leishmania (L.) venezuelensis y la Leishmania (L.) pifanoi y la LC de mucosas (MLC) causada por miembros del complejo de la L. viannia. En las regiones endémicas, pueden coexistir varias especies de Leishmania. La identificación de las especies infectantes basándose sólo en los síntomas clínicos es difícil, sobre todo porque varias especies pueden producir tanto LLC como MLC. La confirmación del diagnóstico y la identificación correcta son importantes para un tratamiento apropiado y específico para cada especie y para los estudios epidemiológicos.
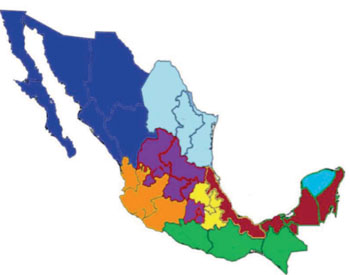
Imagen: Un mapa de México donde se muestran en color marrón las regiones endémicas de Leishmania estudiadas: de izquierda a derecha los estados de Veracruz, Tabasco, Campeche y Quintana Roo (Fotografía cortesía de la Prof. Monroy-Ostria A. et al, el Instituto Politécnico Nacional y la revista Interdisciplinary Perspectives on Infectious Diseases).
En un trabajo conjunto internacional dirigido por Amalia Monroy-Ostria, profesora de la Escuela Nacional de Ciencias Biológicas del Instituto Politécnico Nacional (IPN; Ciudad de México, México), se evaluó un análisis por PCR-RFLP (polimorfismo de la longitud de los fragmentos de restricción) basado en los genes del ITS1 (espaciador 1 de transcripción interna) conservado, para el diagnóstico directo de la leishmaniosis y la identificación de las especies del parásito que, en medida pequeña pero significativa, coexisten en las regiones endémicas de Leishmania del sureste de México. La mayoría de las muestras clínicas examinadas, 109 de 116 (94%), mostraron patrones similares a los de la L. mexicana, 2 mostraron patrones similares a los de la L. viannia y 5 mostraron patrones que sugieren una co-infección con 2 cepas: co-infección con L. (L.) mexicana y L. (V.) viannia o con L. (L.) mexicana y L. (L.) amazonensis. De 21 aislamientos de Leishmania, el 52% mostró un patrón similar al de la cepa L. (L.) mexicana, 5% mostró un patrón mixto compatible con el de la L. (L.) mexicana y L. (V.) viannia, 8 con L. (L.) amazonensis y L. (L.) mexicana y 1 con el de la L. (V.) viannia.
El análisis del ITS1 con PCR-RFLP permite el diagnóstico de la leishmaniosis directamente (sin necesidad de aislamiento del parásito a partir de muestras clínicas) y la determinación simultánea de la mayoría de especies infectantes de Leishmania del Nuevo Mundo, en un tiempo relativamente corto y a bajo costo. Se pueden hacer mejoras, por ejemplo, mediante la adaptación más delimitada a las secuencias que se puedan encontrar para caracterizar más específicamente las especies locales de Leishmania para una región dada (por ejemplo, con respecto a las secuencias de genes amplificados en la PCR o a las enzimas de restricción utilizadas para el RFLP).
El estudio, realizado por Monroy-Ostria A. et al., fue publicado en la revista Interdisciplinary Perspectives on Infectious Diseases, en julio de 2014.
Enlace relacionado:
Instituto Politecnico Nacional