Software de reconocimiento de imágenes aumenta exactitud del diagnóstico de la malaria
Por el equipo editorial de LabMedica en español
Actualizado el 01 Oct 2014
Se ha adaptado un software de reconocimiento facial para ayudar en la identificación del parásito de la malaria mediante el examen microscópico de los frotis sanguíneos.Actualizado el 01 Oct 2014
Con el fin de desarrollar un método visual sencillo, más eficaz para diagnosticar la malaria, un equipo de investigadores escandinavos cooptó algoritmos de visión informáticos similares a los utilizados en los sistemas de reconocimiento facial. El programa funciona en una imagen digitalizada de los frotis delgados de sangre que había sido extendidos en un portaobjetos de microscopio. El algoritmo analiza más de 50.000 glóbulos rojos de la sangre por muestra y los clasifica de acuerdo con la probabilidad de que la célula esté infectada. Entonces, el programa genera un panel de imágenes de un centenar de células con más probabilidades de contener el parásito. Este panel luego es evaluado por un microscopista experimentado que hace el diagnóstico final.
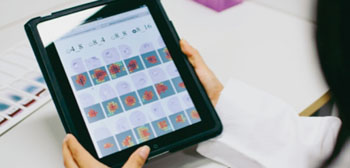
Imagen: El método de detección de parásitos se basa en algoritmos de visión de computadora similares a los usados en los sistemas de reconocimiento facial, combinado con la visualización de solo las áreas diagnósticamente más relevantes. Se pueden usar computadores tabletas para ver las imágenes (Fotografía cortesía del Instituto para Medicina Molecular).
Para comprobar el método, los frotis delgados de sangre teñidos con Giemsa que contenían trofozoitos anulares de Plasmodium falciparum (n = 27) y controles no infectados (n = 20) fueron escaneados digitalmente con un objetivo de inmersión en aceite para capturar unos 50.000 eritrocitos por muestra. Se identificaron regiones candidatas de parásitos con base en el color y tamaño del objeto, seguido por la extracción de características de la imagen (patrones binarios locales, contraste local, y descriptores, de la transformación de la característica en Escala invariante) utilizados como entrada a un clasificador de máquinas de vectores soporte. El clasificador fue entrenado con las láminas digitales de diez pacientes y validado en seis muestras.
De cada área digitalizada de un frotis de sangre, se generó un panel con las 128 regiones candidatas más probables con parásitos. Se pidió a dos microscopistas expertos inspeccionar visualmente el panel en un equipo Tablet PC y juzgar si el paciente estaba infectado con P. falciparum. El método consigue una sensibilidad diagnóstica y especificidad de 95% y 100%, así como 90% y 100% para los dos lectores, respectivamente, utilizando la herramienta de diagnóstico. La parasitemia se calculó por separado por un sistema automatizado y el coeficiente de correlación entre los recuentos de parasitemia manuales y automatizados fue de 0.97.
“No estamos sugiriendo que todo el proceso de diagnóstico de la malaria podría o debería ser automatizado. Más bien, nuestro objetivo es desarrollar métodos que requieren significativamente menos mano de obra que los tradicionales y que tienen un potencial para aumentar considerablemente el rendimiento en el diagnóstico de la malaria”, dijo el autor principal, el Dr. Johan Lundin, director de investigación en el Instituto de Medicina Molecular (Helsinki, Finlandia).
El estudio con la descripción completa de la nueva aproximación diagnóstica fue publicado en la edición digital del 21 de agosto de 2014, de la revista PLOS ONE.
Enlace relacionado:
Institute for Molecular Medicine