Detección sensible y exacta del cáncer a través del análisis de los patrones de metilación del ADN libre circulante
Por el equipo editorial de LabMedica en español
Actualizado el 01 Jul 2020
Un método avanzado de biopsia líquida para la detección y diagnóstico de varios tipos de cáncer se basa en el análisis de los perfiles de metilación del ADN tumoral circulante.Actualizado el 01 Jul 2020
El diagnóstico definitivo del tumor se basa en muestras de tejido obtenidas mediante cirugía invasiva. Los métodos de diagnóstico no invasivos podrían brindar una oportunidad para evitar la cirugía y mitigar los riesgos innecesarios para los pacientes.
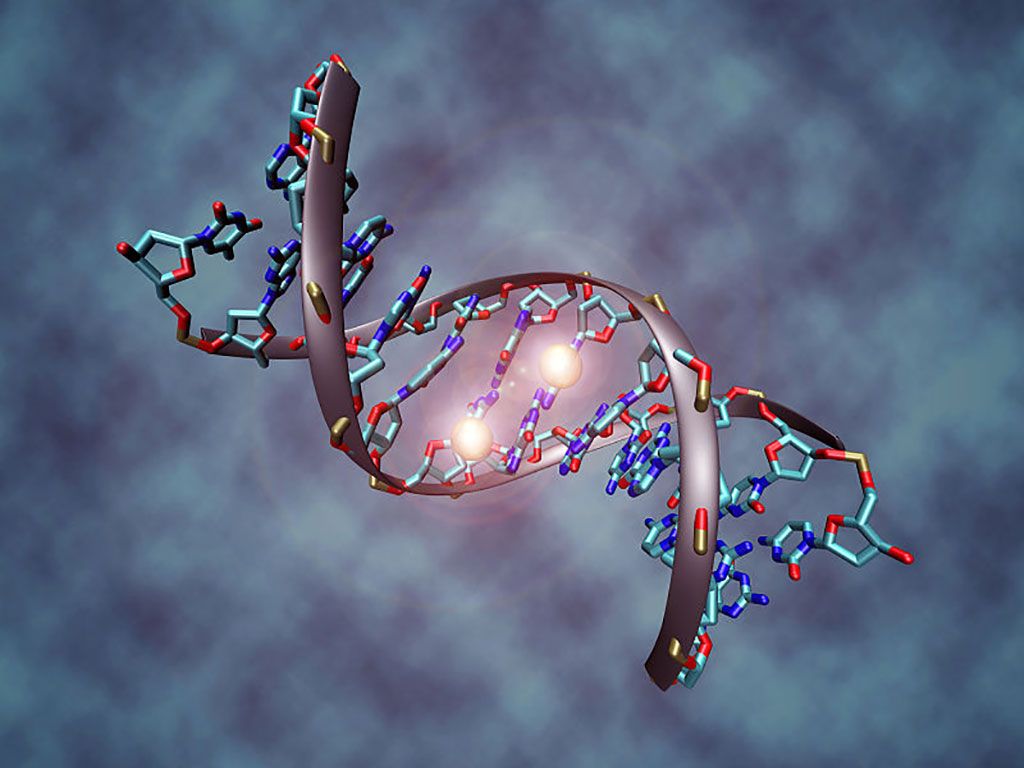
Imagen: Esta imagen muestra una molécula de ADN que ha sido metilada en ambas cadenas en la citosina del centro. Las dos esferas blancas representan grupos metilo (Fotografía cortesía de Wikimedia Commons)
A este respecto, se ha encontrado que el ADN tumoral circula en la sangre de los pacientes con cáncer junto con el ADN de las células no cancerosas. La metilación del ADN es un determinante importante de la variación fenotípica humana, pero su especificidad inherente de tipo celular ha impedido el progreso en esta área de investigación. En regiones genómicas excepcionales, la variación en la metilación del ADN entre individuos ocurre sistémicamente. Al igual que las variantes genéticas, estas variantes epigenéticas sistémicas “interindividuales” son estables, pueden influir en el fenotipo y pueden ser evaluadas en cualquier muestra de ADN obtenida fácilmente. El conjunto de modificaciones de metilación de ácido nucleico en el genoma de un organismo o en una célula particular, se llama un metiloma.
Un equipo de investigadores canadienses, miembros de la Red de Salud de la Universidad (Toronto, Canadá), empleó un método de inmunoprecipitación de ADN metilado, libre de células, y secuenciación de alto rendimiento (cfMeDIP–seq), que es un ensayo altamente sensible capaz de detectar tumores en etapa temprana. En dos artículos publicados en la edición en línea del 22 de junio de 2020 de la revista Nature Medicine, los investigadores describieron el uso de este método para diagnosticar cánceres de cerebro y riñón.
En el estudio de cáncer cerebral, los investigadores rastrearon el origen y el tipo de cáncer comparando muestras de biopsia tumoral de pacientes con el análisis de ADN libre de células (ADNc) en muestras de plasma sanguíneo de 221 pacientes. Con este método, pudieron unir el ADNc del plasma circulante con el ADN del tumor, confirmando la capacidad para identificar el ADN del tumor cerebral que circula en la sangre. Luego, utilizando un método de aprendizaje automático, los investigadores desarrollaron un programa de computadora con el fin de clasificar el tipo de tumor cerebral basado únicamente en el ADN tumoral circulante.
Se demostró que el mismo análisis de sangre identifica con exactitud el cáncer de riñón a partir del ADN libre de células circulantes obtenido del plasma o de la orina. El método se probó en muestras de 99 pacientes con cáncer de riñón temprano y avanzado, 15 pacientes con cáncer de vejiga urotelial en estadio IV y 28 pacientes de control sanos, libres de cáncer. Después del análisis de muestras de suero para determinar los patrones de metilación del ADNc, los investigadores informaron una clasificación exacta de los pacientes en todas las etapas del carcinoma de células renales (CCR) en plasma y demostraron la validez del ensayo para identificar a pacientes con CCR utilizando la determinación del ADN libre de células de la orina, aunque con algo menos de exactitud.
El autor principal (en el artículo sobre cáncer cerebral), el Dr. Daniel De Carvalho, profesor asociado de biofísica médica en la Universidad de Toronto (Canadá), dijo: “La posibilidad de mapear modificaciones epigenéticas en todo el genoma, combinado con métodos computacionales poderosos nos han llevado a este punto de inflexión. La caracterización molecular de los tumores mediante el análisis de las alteraciones epigenéticas además de las mutaciones genéticas nos da una comprensión más completa de las características alteradas de un tumor, y abre las posibilidades para pruebas tumorales más específicas, sensibles y agnósticas”.
Enlace relacionado:
Red de Salud de la Universidad
Universidad de Toronto