Desarrollan un análisis con una plantilla doble de ácidos nucleicos para el cáncer
Por el equipo editorial de LabMedica en español
Actualizado el 22 Jan 2020
La secuenciación paralela masiva o la secuenciación paralela masivamente es cualquiera de varios enfoques de alto rendimiento para la secuenciación de ADN utilizando el concepto de procesamiento masivamente paralelo; también se llama secuenciación de próxima generación o secuenciación de segunda generación.Actualizado el 22 Jan 2020
La secuenciación dirigida de próxima generación es un método poderoso para identificar de manera integral los biomarcadores para el cáncer. El material de partida es actualmente ADN o ARN para diferentes variaciones, pero dividirlo en dos ensayos es oneroso y, a veces, poco práctico, lo que provoca un retraso o una falta total de detección de eventos críticos, en particular, eventos de fusión potentes y detectables.
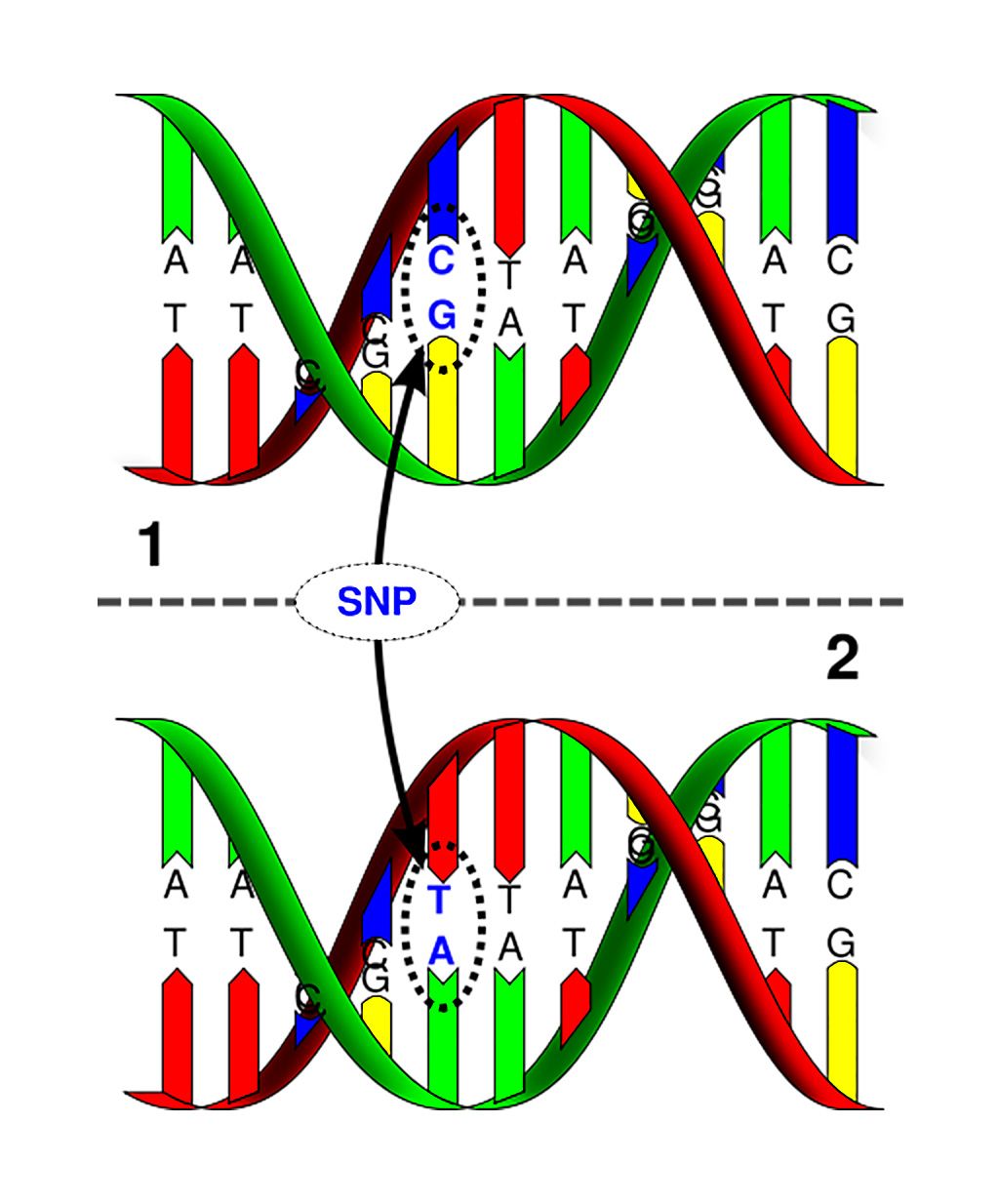
Imagen: Polimorfismo de nucleótido único: la molécula de ADN superior difiere de la molécula de ADN inferior en la ubicación de un solo par de bases (Fotografía cortesía de la Universidad Marshall).
Los científicos del Hospital de Cáncer de Zhejiang (Hangzhou, China) y sus asociados, desarrollaron un ensayo de un solo tubo, de doble plantilla y una tubería de bioinformática integrada, para la detección de variantes relevantes. El ARN se usó para la detección de fusión, mientras que el ADN se usó para variaciones de un solo nucleótido (SNV) e inserción y deleciones (indeles). La química de reacción presentaba ligadura de adaptador con código de barras, amplificación lineal multiplexada y reacción en cadena de polimerasa multiplexada (PCR) para reducción de ruido y detección de fusión novedosa. También se desarrolló un ensayo de control de calidad auxiliar.
Los investigadores informaron que en una cohorte de cáncer de pulmón de 1.000 muestras, identificaron todos los puntos calientes y fusiones SNV/indels principales, así como la omisión del exón 14 MET y varias fusiones nuevas o raras. Las frecuencias de ocurrencia estuvieron en línea con los informes anteriores y fueron verificadas por secuenciación de Sanger. Un evento de fusión notable fue HLA-DRB1-MET, que constituyó la segunda fusión MET intergénica que se detectó en el cáncer de pulmón.
Los autores concluyeron que su método debería beneficiar no solo a la mayoría de los pacientes que portan objetivos centrales accionables, sino también a aquellos con variaciones raras. La extensión futura de este ensayo a la expresión de ARN y el perfil del número de copias de ADN de genes diana, tales como el ligando de muerte programado 1, pueden proporcionar biomarcadores adicionales para terapias de punto de control inmune. El estudio fue publicado en la edición de diciembre de 2019 de la revista Clinical Chemistry.
Enlace relacionado:
Hospital de Cáncer de Zhejiang







 Analyzer.jpg)