Microscopía no invasiva detecta la activación y diferencia los tipos celulares
Por el equipo editorial de LabMedica en español
Actualizado el 09 Dec 2019
Las técnicas de medición que permiten el análisis global de las respuestas celulares al tiempo que conservan la sensibilidad unicelular son cada vez más necesarias para comprender procesos biológicos complejos y dinámicos.Actualizado el 09 Dec 2019
Las células tienen numerosos rasgos que se pueden usar para identificarlas: se pueden usar las características genéticas, epigenéticas, conductuales, compositivas o morfológicas para definir el fenotipo de una célula dada. A medida que mejoran las capacidades de discriminación de los sistemas de medición, pueden surgir subtipos de células recientemente definidos.
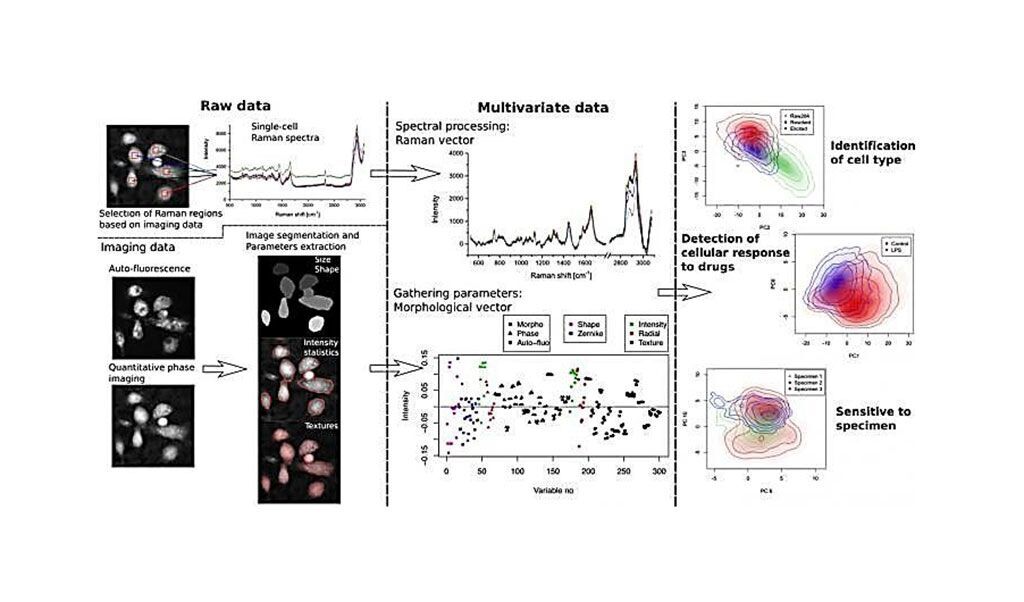
Imagen: Métodos para extraer características del análisis de células inmunes sin usar etiquetas. Los datos multivariados sin etiquetas, compuestos de parámetros tanto morfológicos como espectrales, se utilizan para identificar características de alto nivel a nivel de una sola célula, como el tipo celular, la respuesta a los medicamentos y las diferencias de respuesta entre las muestras (Fotografía cortesía de la Universidad de Osaka)
Los científicos de la Universidad de Osaka (Osaka, Japón) desarrollaron una plataforma de imagen multimodal sin etiquetas que permite el estudio de cultivos celulares de forma no invasiva sin la necesidad de ningún agente de contraste. El par mostró cómo se pueden emplear las señales libres de etiquetas para crear modelos que puedan detectar el estado de activación de las células de macrófagos y distinguir entre diferentes tipos de células incluso en el caso de poblaciones de células primarias altamente heterogéneas.
Las pruebas compuestas por tres tipos de células y dos condiciones (Control y LPS) producen seis conjuntos principales de condiciones controladas donde las mediciones se han distribuido aproximadamente de manera uniforme entre ellas. Para comprender cómo la variabilidad incontrolable podría afectar los resultados, las mediciones se realizan durante varios días, con células enchapadas en diferentes placas para tener en cuenta las diferencias en el cultivo celular, donde se miden 200-400 células por placa. El equipo combinó la información de imagen de las imágenes de fase cuantitativa (QPI) grabadas con holografía fuera del eje y autofluorescencia (AF), junto con la espectroscopía Raman, para recuperar indicadores de nivel de células individuales que permiten el análisis de respuestas y condiciones celulares.
Luego, el equipo desarrolló herramientas estadísticas generalizadas para evaluar la influencia de los parámetros experimentales controlados (subpoblaciones celulares, estimulación inmunológica) y no controlados (condiciones de cultivo, variaciones animales, etc.) en los biomarcadores sin etiqueta. Estos indicadores pueden detectar diferentes subpoblaciones de células de macrófagos que se originan de diferentes progenitores, así como su estado de activación, y cómo estos cambios están relacionados con diferencias específicas en la morfología y el contenido molecular. Los indicadores moleculares también muestran una mayor sensibilidad que permite la identificación de otras condiciones de investigación, como las diferencias entre las células que se originan en diferentes animales, lo que permite la detección de un comportamiento atípico de subpoblaciones celulares determinadas. El estudio fue publicado el 19 de noviembre de 2019 en la revista Scientific Reports.
Enlace relacionado:
Universidad de Osaka