Validan los datos analíticos completos para los análisis de selección de cADN
Por el equipo editorial de LabMedica en español
Actualizado el 13 Nov 2019
Tradicionalmente se ha usado el tejido tumoral, tanto para el diagnóstico del cáncer como para la prueba de biomarcadores moleculares. Con demasiada frecuencia, el tejido obtenido se agota durante el diagnóstico inicial, dejando tejido insuficiente para la posterior prueba de biomarcadores.Actualizado el 13 Nov 2019
El uso de biopsias líquidas en la medicina de precisión para la estratificación de pacientes que usan biomarcadores asociados con terapias dirigidas, es una tendencia emergente en oncología que cada vez tiene más adeptos para guiar las decisiones terapéuticas en el manejo del cáncer.
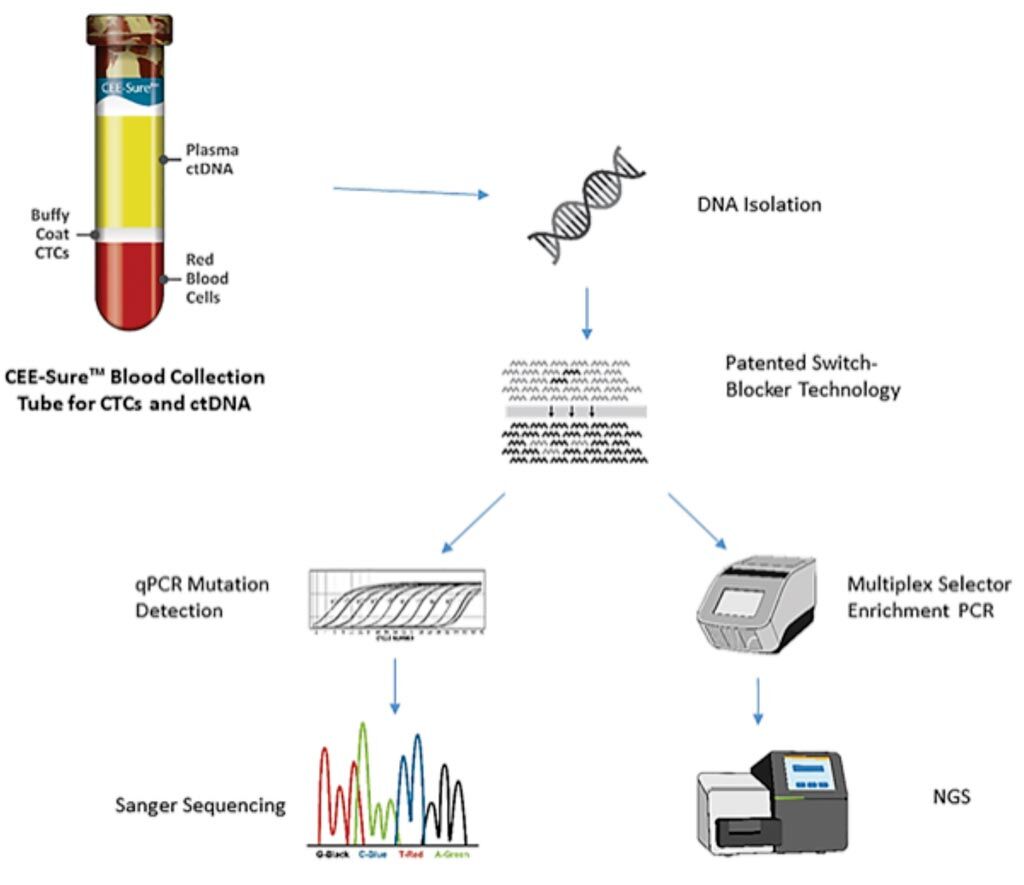
Imagen: Diagrama de la plataforma Selector de Objetivos para el cADN con sensibilidad de detección de copia única para mutaciones de EGFR, BRAF y KRAS clínicamente accionables (Fotografía cortesía de Biocept Inc).
Un equipo de científicos que trabaja para la empresa comercial Biocept Inc (San Diego, CA, EUA) presentó una descripción detallada del enfoque de Selector de Objetivos de la empresa, incluido un método de supresión de tipo salvaje que la empresa ha llamado “bloqueador de interruptor”. También informaron una fuerte reproducibilidad, y una sensibilidad y especificidad analítica en cinco genes, que coinciden estrechamente con algunos de los otros métodos de reacción en cadena de polimerasa (PCR) de alta sensibilidad que también se utilizan en la clínica y/o se comercializan como kits.
Las líneas celulares modelo se propagaron en medio de cultivo con suero fetal bovino al 10% y las células se recolectaron y procesaron para extraer ADN usando el QIAamp DNA Mini Kit (QIAGEN, Germantown, MD, EUA). El ADN libre de células se extrajo de 4 mL de muestras de plasma usando el kit de ADN circulante Qiasymphony DSP de Qiagen en el instrumento QIAsymphony SP. El ADN libre de células se eluyó en 100 μL de solución tampón. Las muestras de ADN se cuantificaron luego con el kit de ensayo Qubit dsDNA HS (Thermo Fisher Scientific, Waltham, MA, EUA). Típicamente, se usaron 10 μL de muestra de ADN para cada reacción de ensayo del Seleccionador de Objetivos.
Se usaron más de 600 muestras para demostrar la sensibilidad analítica, y cada uno de los cinco objetivos estudiados: deleciones del exón 19 de EGFR, mutaciones L858 y T790, BRAF V60 y KRAS G12/G13 y demostraron una sensibilidad analítica de 0,02% o mejor y se observó un solo falso negativo, de un ensayo BRAF, en 667 pruebas realizadas. Se realizaron otras 560 pruebas para calcular la especificidad analítica, que Biocept informó fue superior al 99% para todas las alteraciones objetivo. La compañía tuvo dos falsos positivos en los ensayos después de realizar su protocolo de prueba clínica completo de reacción en cadena de polimerasa cuantitativa (qPCR), seguido de secuenciación de Sanger y límites de temperatura de la curva de fusión, que utiliza para eliminar cualquier producto de amplificación no específico.
Los autores señalaron que, “aunque la ddPCR es una metodología extremadamente sensible, necesita diseño y pruebas para cada mutación específica dentro de una región de punto caliente. Por ejemplo, evaluar las siete mutaciones más comunes en el exón 2 de KRAS (codones 12 y 13) requiere ejecutar al menos siete ensayos separados. Se requiere un ADN de entrada para cada variante de un solo nucleótido (SNV), lo que puede agotar la muestra disponible. Por el contrario, un único ensayo de selección de objetivos cubre el mismo punto de acceso de dos codones con sensibilidad para todos los SNV”. El estudio fue publicado el 3 de octubre de 2019 en la revista PLOS ONE.
Enlace relacionado:
Biocept Inc
QIAGEN
Thermo Fisher Scientific