Cepa nueva de bacterias causa la escarlatina
Por el equipo editorial de LabMedica en español
Actualizado el 02 Oct 2019
La escarlatina es un exantema clásico de la infancia causado por la bacteria Streptococcus pyogenes (estreptococo del grupo A). Los síntomas, que afectan a los niños pequeños, incluyen temperatura alta, dolor de garganta y una erupción de color rojo rosado que se siente como papel de lija.Actualizado el 02 Oct 2019
La escarlatina es causada por toxinas liberadas por la bacteria Streptococcus pyogenes, también conocida como Strep A, y los casos siguen un patrón estacional que alcanza su punto máximo entre marzo y mayo. La escarlatina se trata fácilmente con antibióticos. En el Reino Unido, los casos de infecciones invasivas causadas por la misma bacteria aumentaron en 2016 en comparación con los cinco años anteriores.
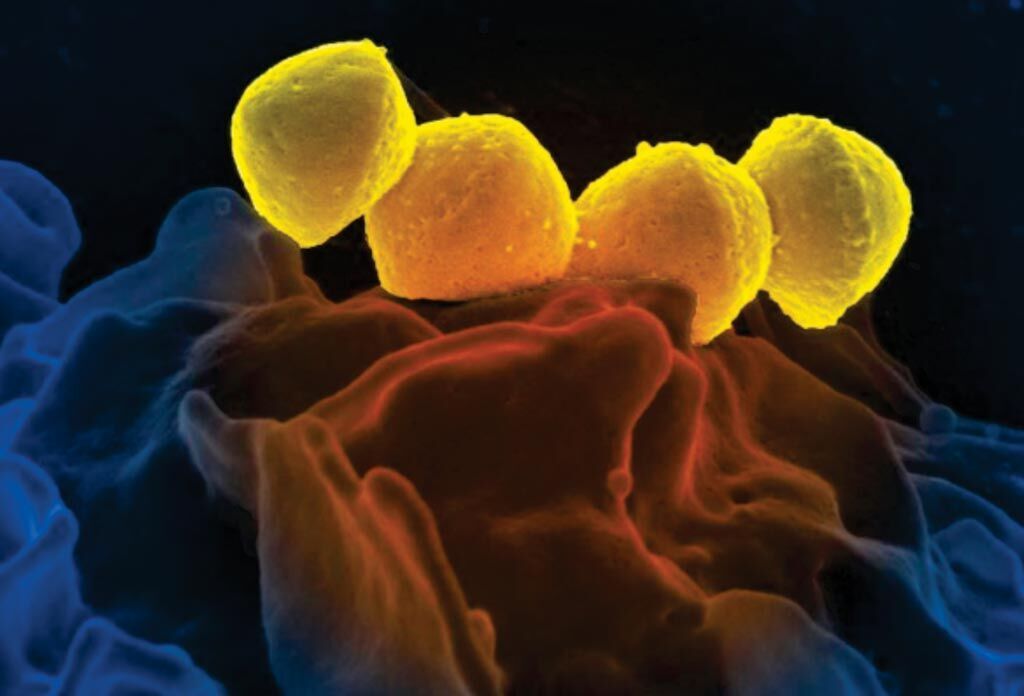
Imagen: Esta imagen microscópica electrónica de barrido, coloreada digitalmente (SEM) muestra cuatro bacterias Streptococcus del grupo A (GAS) de color amarillo, que se encuentran sobre la superficie de un glóbulo blanco humano (WBC), denominado neutrófilo (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los Estados Unidos).
Los especialistas en enfermedades infecciosas del Colegio Imperial de Londres (Londres, Reino Unido) y sus colegas, identificaron las cepas de Strep A que causan infecciones en Londres y más ampliamente en Inglaterra y Gales, según lo definido por la presencia del tipo de gen emm. Descubrieron que el aumento inicial de la escarlatina en 2014 en Londres se asoció con los tipos de cepa Strep A emm3 y emm4. Sin embargo, durante la primavera de 2015 y 2016, las cepas emm1 se volvieron dominantes entre las infecciones de garganta.
En la primavera de 2014, solo el 5% (5/96) de los aislados de la bacteria recolectada en el noroeste de Londres eran cepas emm1, pero para 2015, esta cifra aumentó al 19% (28/147). En 2016, emm1 se convirtió en la cepa más frecuente con un 33% (47/144 aislamientos). Para investigar más a fondo los aislamientos emm1, los científicos secuenciaron los genomas de los 135 aislamientos no invasivos de la bacteria emm1, recolectados en el noroeste de Londres entre 2009 y 2016, y todos los 552 aislamientos invasivos emm1 recolectados en Inglaterra y Gales durante los picos de la enfermedad estacional entre 2013 y 2016, y los compararon entre sí. También evaluaron la cantidad de toxina producida por diferentes cepas de emm1.
Los investigadores descubrieron que la mayoría de las cepas emm1 de 2015 y 2016 eran un clon emm1 distinto y separable, al que se refirieron como M1UK. El clon tenía 27 mutaciones únicas y se asoció con una producción significativamente mayor de la exotoxina pirógena estreptocócica toxina A (SpeA). Esta toxina desencadena la escarlatina y puede contribuir a la faringitis estreptocócica A y a algunas infecciones invasivas. El análisis confirmó que M1UK produce nueve veces más toxina que otras cepas emm1 (190 ng/mL en comparación con 21 ng/mL). En 2010 ya estaba presente en Inglaterra y para 2016, M1UK representaba el 84% de todos los genomas emm1 analizados en Inglaterra y Gales.
Shiranee Sriskandan, FRCP, profesora de enfermedades infecciosas y autora principal del estudio, dijo: “El clon bacteriano definido que descubrimos parece estar limitado en gran medida al Reino Unido, pero el hecho de que se hayan identificado dos ejemplos de estos clones en otros lugares sugiere que el clon tiene el potencial de propagación internacional y que ya puede estar presente en otros países. Sin embargo, también es posible que el linaje no dure. En el pasado, aparecieron algunos linajes que luego desaparecieron rápidamente”. El estudio fue publicado el 10 de septiembre de 2019 en la revista The Lancet Infectious Diseases.
Enlace relacionado:
Colegio Imperial de Londres