Encuentran mutación que determina la naturaleza de la respuesta del huésped a la infección por SARM
Por el equipo editorial de LabMedica en español
Actualizado el 01 Oct 2019
Un artículo reciente describió los factores genéticos que determinan por qué algunas personas desarrollan infecciones crónicas por Staphylococcus aureus resistente a la meticilina (SARM), mientras que otras desarrollan infecciones que se resuelven relativamente rápido.Actualizado el 01 Oct 2019
La gravedad y la duración de la infección por SARM varían ampliamente entre los individuos. Los factores del huésped que predisponen a la infección persistente por SARM son poco conocidos, aunque los estudios de asociación genética ya pudieron identificar variantes potencialmente influyentes.
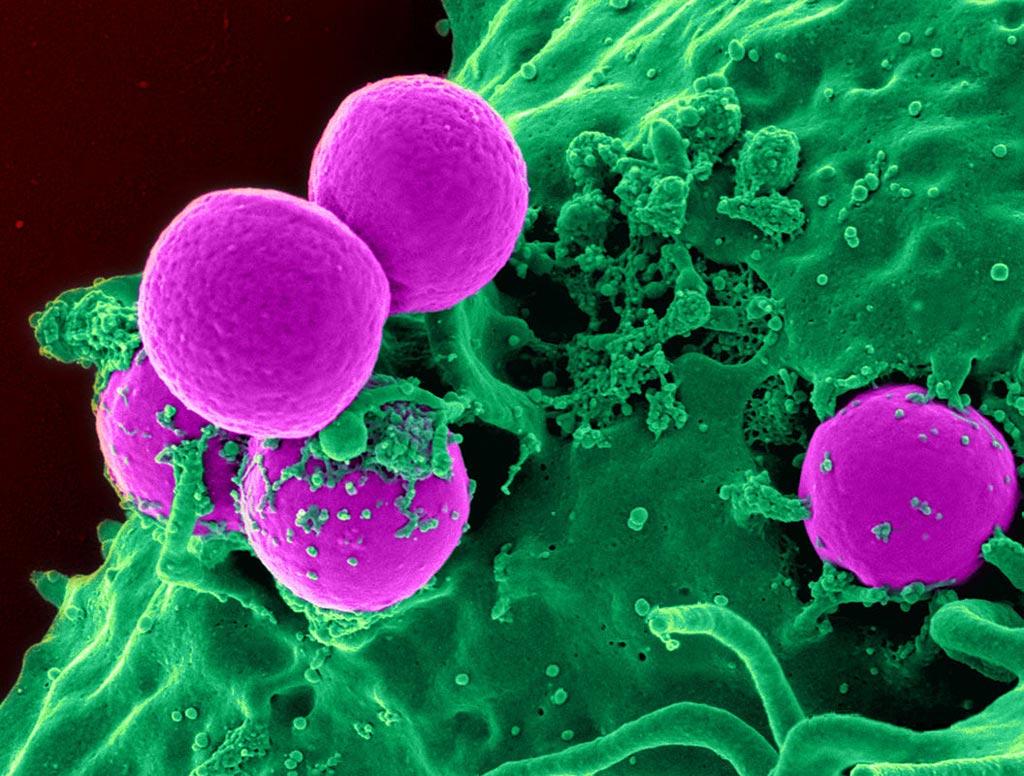
Imagen: Una microfotografía electrónica de barrido (SEM) de neutrófilos humanos ingiriendo un SARM (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas de los EUA).
Los investigadores de la Universidad de Duke (Durham, NC, EUA) buscaron dichos factores del huésped analizando dos conjuntos de pacientes que habían sido relacionados estrechamente por edad, género, condiciones de salud y otros factores de riesgo, para las infecciones del torrente sanguíneo por SARM. Sesenta y ocho pacientes fueron incluidos en el estudio; la mitad con una infección persistente por SARM y la otra mitad que pudo eliminar la infección del torrente sanguíneo. Se utilizó la secuenciación del exoma completo para identificar las diferencias genómicas entre los dos conjuntos de pacientes.
Los resultados revelaron que una mutación ubicada en el ADN de la región DNMT3A del cromosoma 2p se expresó en aproximadamente el 62% de los pacientes que eliminaron su infección por SARM, mientras que se expresó en solo el 9% en aquellos que tenían infecciones persistentes.
En otra serie de experimentos, los investigadores demostraron que las variantes de DNMT3A podían alterar la respuesta del huésped a la infección a través del aumento de la metilación de genes reguladores clave, lo que resultó en una producción reducida de interleuquina-10 y, a su vez, permitió una respuesta inmune más protectora que eliminó la infección.
“La creciente prevalencia de infecciones por estafilococos resistentes a los antibióticos ha creado una necesidad urgente de comprender mejor quién es más susceptible a estas infecciones por S. aureus difíciles de tratar y por qué”, dijo el autor principal, el Dr. Vance Fowler, profesor de medicina, genética molecular y microbiología en la Universidad de Duke. “Nuestro estudio identifica una mutación particular de DNMT3A que contribuye a una mayor capacidad para resolver las infecciones por SARM. El mecanismo para esto parece ser una mayor metilación de las regiones reguladoras de genes y niveles más bajos de la citoquina antiinflamatoria IL-10”.
El artículo sobre el SARM se publicó en la edición en línea del 16 de septiembre de 2019 de la revista Proceedings of the [U.S.] National Academy of Sciences.
Enlace relacionado:
Universidad de Duke