Asocian infección de C. difficile con el bacterioma vinculado a los hongos
Por el equipo editorial de LabMedica en español
Actualizado el 17 Sep 2019
La infección por Clostridioides difficile (ICD), la etiología más común de la diarrea infecciosa nosocomial, es causada por una disbiosis intestinal que casi siempre se atribuye a los antibióticos. Esto causa una de las infecciones adquiridas en el hospital más comunes en los Estados Unidos.Actualizado el 17 Sep 2019
Cada vez hay más datos que muestran que distintas estructuras comunitarias bacterianas y fúngicas permiten diferenciar el estado disbiótico de la ICD de la diarrea asociada a antibióticos debido a otras causas. Los datos sugieren que las interacciones entre especies fúngicas y bacterianas juegan un papel importante en las infecciones nosocomiales.
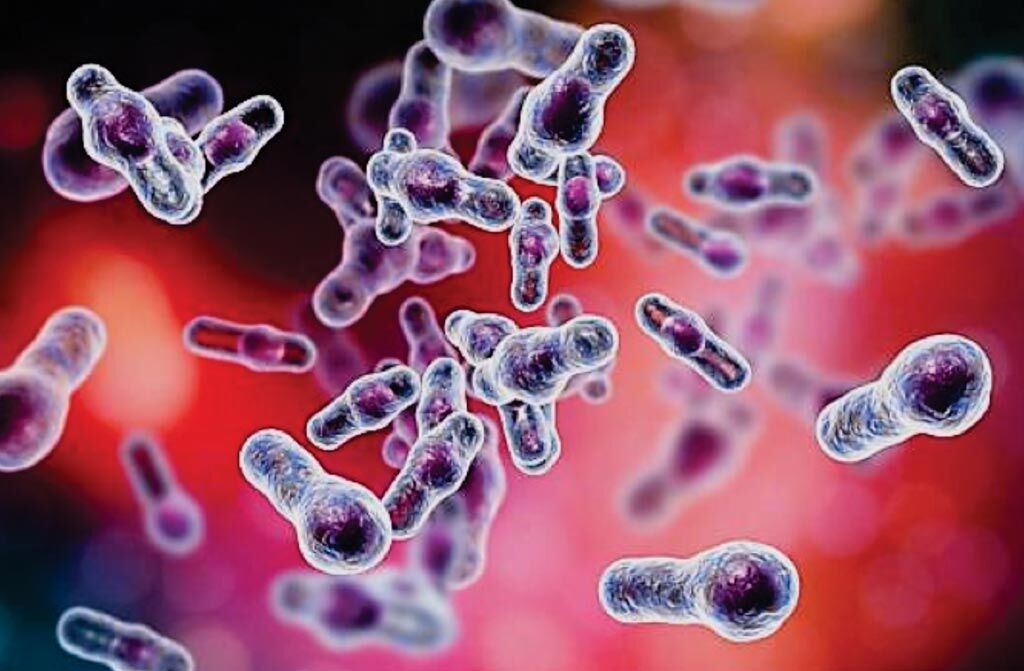
Imagen: Bacilos de Clostridioides difficile en forma de palillos de tambor tal como aparecen en la microscopía electrónica de barrido (Fotografía cortesía de Kateryna Kon, PhD).
Un equipo de científicos que colabora con la Universidad de Arizona (Tucson, AZ, EUA) recolectó heces diarreicas de 49 pacientes hospitalizados (18 de los cuales dieron positivo para ICD) bajo estrictos criterios de inclusión. La prueba de C. difficile se realizó usando una prueba de amplificación de ácido nucleico, disponible comercialmente, diseñada para detectar una secuencia altamente conservada dentro del gen tcdA. Se conservaron las muestras de heces positivas y negativas para C. difficile.
Los extractos de ADN fecal fueron analizadas con la reacción en cadena de la polimerasa (PCR) (Illumina, San Diego, CA, EUA) para los genes 16S rARN e ITS2, agrupados en proporciones equimolares, purificados en gel y secuenciados en el Illumina MiSeq (Bibliotecas 16S rARN) y plataformas NextSeq (bibliotecas ITS). Las secuencias bacterianas (gen 16S rARN) y fúngicas (ITS) se filtraron de calidad, se agruparon en unidades taxonómicas operativas (OTU) y se normalizaron utilizando las líneas de producción USEARCH y QIIME. El equipo utilizó un enfoque de secuenciación escalonada para identificar taxones de bacterias y hongos enriquecidos, utilizando la secuenciación de amplicon del gen 16S rARN y el espaciador transcrito interno (ITS), con metagenómica y metatranscriptómica coincidentes realizadas en un subconjunto de la población.
Los científicos informaron que distintas composiciones bacterianas y fúngicas permitían diferenciar a los pacientes positivos y negativos para la ICD, con la mayor diferenciación entre las cohortes observadas en función de la metatranscriptómica bacteriana. Los análisis de redes bipartitas demostraron que los taxones Aspergillus y Penicillium compartían una fuerte relación positiva en pacientes con ICD y juntos formaban relaciones negativas coexistentes con varios taxones bacterianos, incluidos Oscillospira, Comamonadaceae, Microbacteriaceae y Cytophagaceae. Los investigadores identificaron nuevas vías asociadas con infecciones por C. difficile. Esas vías apuntaban a otras especies bacterianas, incluida Escherichia coli, que pueden contribuir a la disbiosis o al desequilibrio entre las especies bacterianas en el intestino, asociadas con C. difficile.
David B. Stewart, MD, cirujano y primer autor del estudio, dijo: “El desarrollo de la infección por C. difficile aparentemente es influenciado tanto por patógenos bacterianos como por hongos particulares a esta enfermedad. Hasta ahora, los hongos han sido poco estudiados y subestimados en el microbioma intestinal”. El estudio fue publicado el 28 de agosto de 2019 en la revista mSphere.
Enlace relacionado:
Universidad de Arizona
Illumina