Comparan análisis moleculares para los patógenos gastrointestinales
Por el equipo editorial de LabMedica en español
Actualizado el 02 Sep 2019
La gastroenteritis infecciosa es causada por una infección con bacterias, virus o parásitos. Es un problema de salud pública importante, siendo una de las principales causas de morbilidad y mortalidad en todo el mundo. A pesar de los esfuerzos de salud pública en educación sobre seguridad alimentaria y sistemas de tratamiento de agua, la enfermedad diarreica aguda inducida por patógenos sigue siendo una causa importante de morbilidad y mortalidad a nivel mundial.Actualizado el 02 Sep 2019
Varias pruebas de laboratorio, que incluyen cultivo bacteriano de rutina, inmunoensayos enzimáticos serológicos (EIA) para virus, análisis molecular utilizando la reacción en cadena de la polimerasa (PCR) para bacterias y virus y microscopía para parásitos, se realizan de forma rutinaria para pacientes con vómitos, diarrea y dolor abdominal con el fin de descartar patógenos gastrointestinales comunes. El tiempo de respuesta rápido es importante para el diagnóstico, el manejo clínico y el control de infecciones.

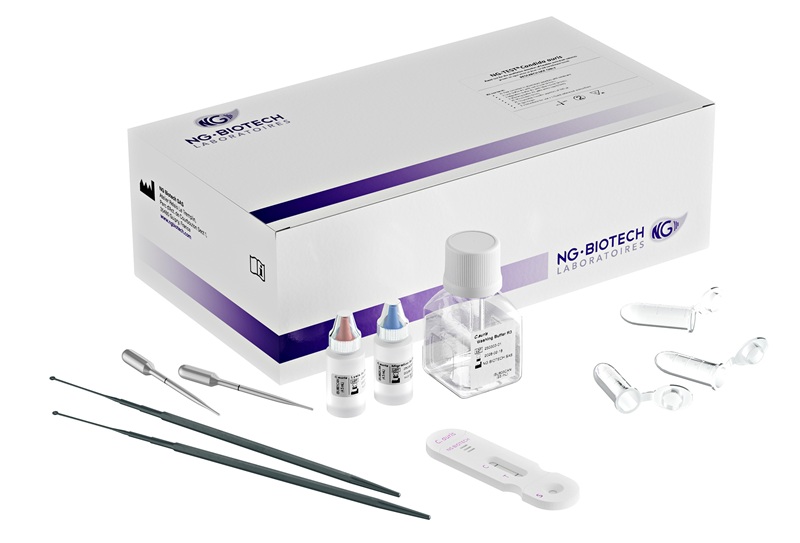
Imagen: Ensayos de panel completo gastrointestinal Allplex, para la detección e identificación de 25 patógenos gastrointestinales (seis virus, 13 bacterias y seis parásitos) utilizando PCR, en tiempo real, de un solo paso (Fotografía cortesía de Seegene).
Los científicos del laboratorio médico de la Universidad Católica de Corea (Seúl, Corea) recolectaron un total de 858 muestras de heces, enviadas al departamento de medicina de laboratorio en el hospital entre enero y octubre de 2016. Cada muestra fue enviada para una de cuatro pruebas microbiológicas diferentes: 197 muestras (23%) para ensayos de reacción en cadena de polimerasa multiplex (PCR) desarrolladas en el laboratorio para Escherichia coli y Salmonella spp.; 182 muestras (21%) para cultivo bacteriano de rutina, 175 muestras (20%) para cultivo de Clostridium difficile toxigénico y con la prueba Xpert C difficile/Epi (Cepheid, Sunnyvale, CA, EUA) y 304 muestras (36% ) para pruebas de antígeno de norovirus y rotavirus. Cada uno de los tres ensayos permitía el uso de muestras de heces crudas.
Otros métodos utilizados en el estudio incluyeron el ensayo de panel completo gastrointestinal Allplex con 24 objetivos: 13 bacterias, cinco virus y seis parásitos en cuatro paneles (Seegene, Seúl, Corea del Sur); el Panel de Patógenos Gastrointestinales xTAG con 15 objetivos: nueve bacterias, tres virus y tres parásitos (Luminex Corporation, Austin, TX, EUA) y el panel BD MAX Enteric con cinco bacterias y tres parásitos (Becton Dickinson, Franklin Lakes, NJ, EUA). Se usaron otras pruebas para la confirmación.
Los científicos informaron que las concordancias de porcentaje positivo general de Seegene, Luminex y BD MAX fueron 94% (258/275), 92% (254/275) y 78% (46/59), respectivamente; para Salmonella, Luminex mostró un porcentaje bajo de concordancia negativa debido a los falsos positivos frecuentes (n = 31) que muestran una intensidad fluorescente mediana baja. Para los virus, las concordancias de porcentaje positivo/negativo de Seegene y Luminex fueron de 99% / 96% y 93% / 99%, respectivamente. En comparación con las pruebas de rutina de microbiología, Seegene, Luminex y BD MAX identificaron adicionalmente 39, 40 y 12 patógenos, respectivamente. Sesenta y un casos, 16 casos con Seegene, 51 casos con Luminex y un caso con BD MAX, mostraron resultados positivos para múltiples patógenos, pero solo tres fueron positivos por consenso.
Los autores concluyeron que los tres ensayos moleculares multiplex mostraron una concordancia sustancial o casi perfecta, y permitieron la identificación adicional de patógenos gastrointestinales. Estos ensayos moleculares multiplex parecen ser una herramienta prometedora para la detección simultánea de múltiples patógenos gastrointestinales. Para decidir cuál de estos ensayos usar como procedimiento de diagnóstico de rutina, se deben tener en cuenta varios factores, tales como el rango de patógenos detectados, el costo, el rendimiento, el tiempo práctico y las habilidades técnicas requeridas. El estudio fue publicado en la edición de agosto de 2019 de la revista Archives of Pathology & Laboratory Medicine.
Enlace relacionado:
Universidad Católica de Corea
Cepheid
Seegene
Luminex Corporation
Becton Dickinson









 Analyzer.jpg)