Características del microbioma humano estratifican a los niños con SII
Por el equipo editorial de LabMedica en español
Actualizado el 07 May 2019
El síndrome del intestino irritable (SII) es una afección gastrointestinal disruptiva caracterizada por hinchazón, cambios en los hábitos intestinales y dolor que afecta hasta al 20% de la población mundial (niños y adultos). La evidencia creciente indica que el inicio y los síntomas del SII están relacionados con el microbioma intestinal.Actualizado el 07 May 2019
Para mejorar el tratamiento de los niños con SII, los investigadores han desarrollado una forma sofisticada de analizar los contenidos microbianos y metabólicos del intestino. Un informe describe cómo una nueva batería de pruebas permite a los científicos diferenciar a los pacientes con SII de los niños sanos e identifica las correlaciones entre ciertos microbios y metabolitos con el dolor abdominal.

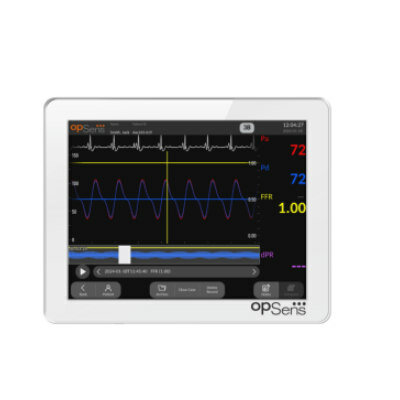
Imagen: El fluorómetro Qubit 4 diseñado para medir con exactitud el ADN, el ARN y la cantidad de proteínas, y ahora también la integridad y calidad del ARN, utilizando los análisis Qubit, altamente sensibles (Fotografía cortesía de Thermo Fisher Scientific).
Los científicos de la facultad de medicina Baylor (Houston, TX, EUA) y sus colegas, obtuvieron muestras de 23 niños preadolescentes con SII (de 7 a 12 años) y 22 controles sanos. Se pidió a los participantes que hicieran anotaciones diarias en sus diarios sobre el dolor y las heces durante dos semanas y que proporcionaran muestras de heces (fecales).
El ADN de las heces se extrajo utilizando el kit de aislamiento de ADN PowerSoil (MO BIO Laboratories, Carlsbad, CA, EUA). La calidad y el rendimiento del ADN se evaluaron mediante geles de agarosa NanoDrop 1000 (NanoDrop, Wilmington, DE, EUA) y el fluorómetro Qubit (Life Technologies, Carlsbad, CA, EUA). Se generaron bibliotecas de WGS utilizando bibliotecas pareadas de 100 pb y la plataforma HiSeq 2000 (Illumina, San Diego, CA, EUA).
Los investigadores descubrieron que había diferencias en la composición bacteriana, los genes bacterianos y los metabolitos fecales en niños con SII en comparación con los controles sanos. Además de identificar las correlaciones de estos factores con el dolor abdominal, generaron un clasificador de gran exactitud utilizando marcadores metagenómicos y metabólicos que permiten diferenciar a los niños con SII de los controles sanos con una exactitud del 80% o más. Este clasificador evalúa metabolitos específicos, tipos de bacterias, vías funcionales y otros factores.
Este clasificador basado en microbiomas puede ayudar potencialmente a identificar subpoblaciones de niños con SII que tienen más probabilidades de beneficiarse de terapias relacionadas con microbiomas, incluida la modificación de la dieta, mientras que guían a otros a planes de tratamiento alternativos apropiados. Los investigadores también proporcionan información sobre cómo los hallazgos específicos relacionados con los microbiomas pueden estar relacionados con el dolor abdominal, lo que abre nuevos enfoques potenciales para el tratamiento.
James Versalovic, MD, PhD, patólogo y coautor del estudio, dijo: “Este estudio destaca la importancia del eje microbioma-intestino-cerebro y nuestra comprensión del dolor abdominal crónico. El desarrollo de nuevos clasificadores de enfermedades basados en datos de microbiomas permite que se desarrollen diagnósticos de precisión para el SII y trastornos similares. Aunque otros estudios han encontrado diferencias en los microbiomas intestinales de los pacientes con SII, este estudio es el primero en combinar el análisis profundo de microbiomas con el desarrollo de nuevas estrategias de diagnóstico”. El estudio se publicó el 17 de abril de 2019 en la revista Journal of Molecular Diagnostics.
Enlace relacionado:
Facultad de Medicina Baylor
MO BIO Laboratories
NanoDrop
Life Technologies
Illumina