Un brote de tuberculosis multirresistente no fue detectado usando las pruebas estándar
Por el equipo editorial de LabMedica en español
Actualizado el 06 Nov 2018
El diagnóstico rápido de Mycobacterium tuberculosis resistente a los medicamentos antituberculosos es esencial para evitar una mayor adquisición de la resistencia a los medicamentos, una alta morbilidad y mortalidad, y una transmisión ininterrumpida de las cepas resistentes.Actualizado el 06 Nov 2018
La detección rápida de resistencia a la rifampicina, el medicamento más importante contra la tuberculosis, es especialmente crucial porque predice la resistencia a múltiples fármacos, definida como resistencia a al menos isoniazida y rifampicina. El lanzamiento global de ensayos moleculares rápidos revoluciona el diagnóstico de resistencia a la rifampicina, predictiva de resistencia a múltiples fármacos, en la tuberculosis.

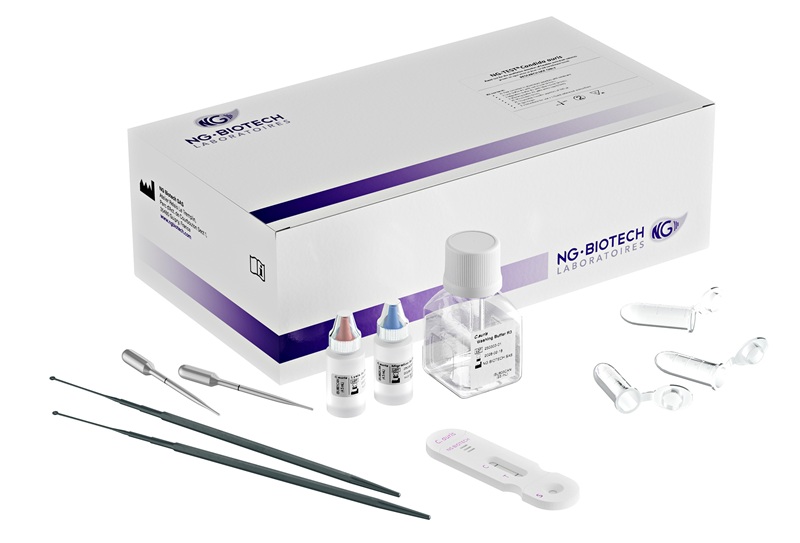
Imagen: La prueba de secuenciación profunda Deeplex-MycTB analiza un amplio panel de genes diana en las bacterias y puede identificar resistencia a más de una docena de antibióticos simultáneamente (Fotografía cortesía de Genoscreen).
Un gran equipo internacional de científicos que trabajan con la Katholieke Universiteit Leuven (Lovaina, Bélgica) examinó los registros de 37.644 cultivos positivos a Mycobacterium tuberculosis de cuatro provincias sudafricanas, para identificar aislamientos con sensibilidad a la rifampicina y resistencia a la isoniazida según las pruebas Xpert MTB/RIF (Cepheid, Sunnyvale, CA, EUA), GenoType MTBDRplus (Hain-Life Science, Nehren, Alemania;) y BACTEC MGIT 960 (Becton, Dickinson and Company, Franklin Lakes, NJ, EUA).
De 1.823 aislamientos que cumplieron con estos criterios, 277 se seleccionaron al azar y se examinaron para la detección de Ile491Phe con PCR multiplex específica de alelo y secuenciación de Sanger de rpoB. Las cepas positivas para Ile491Phe (así como 17 aislamientos portadores Ile491Phe de otro estudio) se analizaron mediante la secuenciación profunda Deeplex-MycTB (Genoscreen, Lille, Francia) y la secuenciación de todo el genoma para evaluar sus patrones de resistencia extensa de transmisión y evolución. La prueba Genoscreen analiza un amplio panel de genes objetivo en la bacteria y puede identificar resistencia a más de una docena de antibióticos simultáneamente.
El equipo identificó Ile491Phe en 37 (15%) de 249 muestras con PCR y resultados de secuenciación múltiplex válidos para alelos, reclasificándolos como resistentes a múltiples medicamentos (MDR). Deeplex-MycTB identificó además los 37 aislamientos como genotípicamente resistentes a todos los medicamentos de primera línea. Seis de los aislamientos sudafricanos albergaron cuatro mutaciones distintas potencialmente asociadas con una disminución de la sensibilidad a la bedaquilina. Consistente con los perfiles genotípicos de Deeplex-MycTB, la secuenciación del genoma completo reveló la propagación silenciosa concurrente en Sudáfrica de un linaje de cepa de tuberculosis MDR que se extiende desde el brote original y al menos otro linaje portador de Ile491Phe independiente que emergió de forma independiente.
Los autores concluyeron que las estrategias diagnósticas actuales no detectan un número importante de casos de tuberculosis MDR que albergan la mutación Ile491Phe en el gen rpoB, lo que resulta en un tratamiento ineficaz de primera línea, una amplificación continua de la resistencia a los medicamentos y una propagación silenciosa concurrente en la comunidad. El estudio fue publicado el 17 de octubre de 2018 en la revista The Lancet Infectious Diseases.
Enlace relacionado:
Katholieke Universiteit Leuven
Cepheid
Hain-Life Science
Becton, Dickinson and Company
Genoscreen