Descubren biomarcador para patógeno hipervirulento
Por el equipo editorial de LabMedica en español
Actualizado el 07 Aug 2018
No existe un método preciso para diferenciar entre la cepa hipervirulenta y la cepa clásica de Klebsiella pneumoniae (cKp), que se observa con mayor frecuencia en el hemisferio occidental, es menos virulenta y generalmente causa infecciones en el entorno hospitalario.Actualizado el 07 Aug 2018
Se han descubierto varios biomarcadores que pueden identificar con exactitud la K. pneumoniae hipervirulenta (hvKp), un patógeno que infecta a personas completamente sanas y puede causar ceguera en un día e infecciones carnívoras, abscesos cerebrales y la muerte en pocos días.
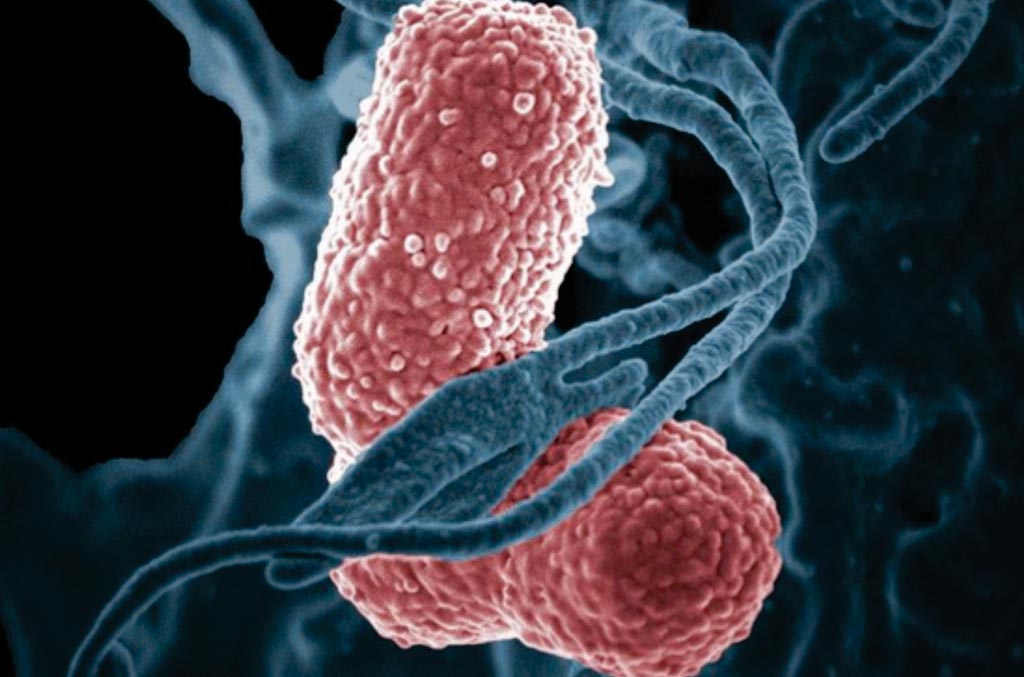
Imagen: Un neutrófilo humano (gris) interactuando con Klebsiella pneumoniae (rosa). Una versión hipervirulenta de este patógeno suscita una preocupación creciente debido a los informes recientes que describen cepas hipervirulentas que son resistentes a los antibióticos (Fotografía cortesía del Instituto Nacional de Alergias y Enfermedades Infecciosas).
Un equipo internacional de científicos liderado por investigadores de la Universidad de Buffalo (Buffalo, NY, EUA) identificó un biomarcador para diferenciar las cepas de hvKp de las cepas de cKp. Escogieron utilizar datos clínicos para desarrollar cohortes de cepas para evaluación, dado que la inclusión de cualquier información genotípica o fenotípica bacteriana en la definición de cohortes de cepas podría introducir sesgo. La cohorte rica en hvKp consistió en 85 cepas aisladas de pacientes no identificados de Taiwán y EUA Dado que la mayoría de las infecciones por K. pneumoniae en Norteamérica y el Reino Unido presumiblemente se deben a cepas de cKp, la cohorte de cepas rica en 90 cKp se generó a partir de aislados de sangre no identificados elegidos al azar de diferentes países.
Los aislados del estudio se evaluaron para la presencia de 10 genes y marcadores para tipos de cápsulas. Estos incluyen varios genes ubicados en el plásmido de virulencia, que se ha demostrado experimentalmente que contribuyen a la hipervirulencia en los modelos de infección in vivo. Se evaluaron dos rasgos fenotípicos hipotéticamente discriminatorios. La detección basada en reacciones de cadena de polimerasa (PCR) de varios tipos de cápsulas se realizó usando el instrumento Applied Biosystems GeneAmp PCR system 9700 (Foster City, CA, EUA).
Los científicos descubrieron que los genes peg-344, iroB, iucA, prmpA y prmpA2 demostraron una exactitud diagnóstica> 0,95 para identificar cepas en la cohorte rica en hvKp. La producción cuantitativa de sideróforos de ≥ 30 μg/mL también predijo fuertemente las cepas como miembros de la cohorte rica en hvKp (exactitud 0,96) y exhibió una relación de riesgo de 31,7 para enfermedad grave o muerte. La prueba de cuerda, un marcador ampliamente utilizado para las cepas hvKp, tuvo un desempeño inferior, logrando una exactitud de solo 0,90.
Thomas A. Russo, MD, profesor de medicina y autor principal del estudio dijo: “La ventaja de identificar estos biomarcadores genéticos es que se pueden convertir en pruebas rápidas de ácidos nucleicos y, si la Administración de Medicamentos y Alimentos de los EUA los aprueba, podrían proporcionar a los médicos un método exacto para determinar rápidamente si un paciente padece una infección debido a la cepa clásica o a la hipervirulenta”. El estudio fue publicado el 20 de junio de 2018 en la revista Journal of Clinical Microbiology.










 Analyzer.jpg)