Espectrometría de masas identifica bacterias resistentes a la colistina
Por el equipo editorial de LabMedica en español
Actualizado el 18 May 2017
Se ha desarrollado un método que utiliza espectrometría de masas para diagnosticar con rapidez y exactitud las infecciones bacterianas resistentes a los fármacos, así como para diferenciar entre los dos mecanismos principales de resistencia.Actualizado el 18 May 2017
La investigación fue una colaboración dirigida por el Dr. Laurent Dortet de la Universidad del Sur de París (París, Francia) y el Dr. Gerald Larrouy-Maumus y el Prof. Alain Filloux, del Colegio Imperial de Londres (Londres, Reino Unido). El equipo hizo los ensayos en Escherichia coli y Klebsiella pneumoniae, ambos miembros de la familia Enterobacteriacae. Algunas cepas se han vuelto resistentes a casi todos los antibióticos disponibles. Con frecuencia, la colistina es la única opción de tratamiento para estas bacterias multirresistentes, pero algunas cepas también han desarrollado resistencia a la colistina.
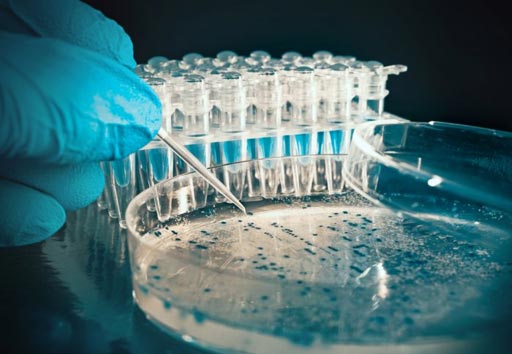
Imagen: Los investigadores ensayaron colonias bacterianas con el método que desarrollaron usando un espectrómetro de masas para diferenciar rápidamente entre las bacterias resistentes a la colistina y las no resistentes, así como para identificar qué bacterias tienen resistencia a través de los plásmidos (Fotografía cortesía del Colegio Imperial de Londres).
La investigación previa había demostrado que había surgido una resistencia a la colistina, codificada por los cromosomas y codificada por los plásmidos. La resistencia codificada por plásmidos se considera más peligrosa porque puede transmitirse de un tipo de bacteria a otro.
“Esta resistencia codificada por el plásmido es particularmente preocupante porque tiene el potencial de propagarse rápida y fácilmente y, si eso ocurre, los medicamentos de último recurso, como la colistina también podrían quedar obsoletos”, dijo el Dr. Dortet, “Si, por otro lado, somos capaces de identificar rápidamente las bacterias que tienen este tipo de resistencia, podemos tomar medidas para detener su propagación. Esto podría incluir el aislamiento del paciente en una sala separada donde son tratados por personal médico dedicado”.
Los investigadores probaron 134 colonias diferentes de bacterias usando un espectrómetro de masas. Encontraron que era posible diferenciar, no sólo entre las bacterias resistentes y no resistentes a colistina, sino también cuáles bacterias tienen resistencia a través de los plásmidos. Además, la prueba se puede realizar en unos 15 minutos y costaría menos de un dólar por muestra.
“Lo interesante de esta técnica es que se basa en una tecnología que ya está disponible en la mayoría de los hospitales. Esto significa que podría ser lanzada de forma rápida y barata, y tener un impacto, potencialmente rápido, en la lucha contra la resistencia a los medicamentos”, dijo el Dr. Larrouy-Maumus.
Los investigadores trabajan ahora con Imperial Innovations, la oficina de transferencia de tecnología del Colegio Imperial de Londres, para patentar la técnica y desarrollarla para uso clínico en los laboratorios hospitalarios.
La prueba también podría ser útil para el cribado de muestras veterinarias, donde se sabe que los niveles de resistencia a la colistina son altos. También podría utilizarse para probar si los nuevos medicamentos son capaces de restaurar la vulnerabilidad de las bacterias a la colistina.
“La detección rápida de la resistencia a la colistina será de enorme valor en la asistencia sanitaria. Permitirá intervenciones tempranas para controlar la transmisión, proteger a los pacientes y mejorar el manejo individual de estos”, dijo la profesora Alison Holmes, del Imperial, “Por fuera del ámbito de la salud, también tendrá un enorme potencial en la agricultura y la producción de carne. La capacidad de discriminar los diferentes mecanismos de resistencia a la colistina también generará una mayor comprensión “.
El estudio fue presentado en el 27º Congreso Europeo de Microbiología Clínica y Enfermedades Infecciosas (22-25 de abril de 2017, Viena, Austria).