Patógenos entéricos se expanden post tratamiento con antibióticos
Por el equipo editorial de LabMedica en español
Actualizado el 24 Oct 2013
El intestino humano es un objetivo frecuente de las bacterias patógenas, pero la comunidad de microbios residentes suministra protección contra las infecciones bacterianas.Actualizado el 24 Oct 2013
La afectación de esta microbiota con antibióticos orales precede, frecuentemente, la emergencia de patógenos entéricos severos, pero estos últimos como aprovechan la falla en la protección ofrecida por la microbiota, es bastante desconocido.
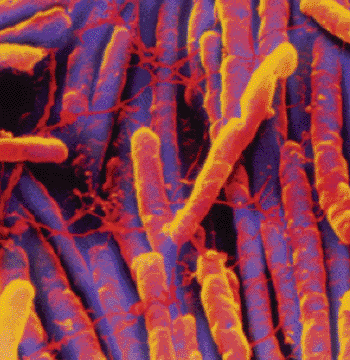
Imagen: Microfotografía electrónica de barrido (SEM) del patógeno Clostridium difficile (Fotografía cortesía de Bioquell).
Científicos de la Universidad de Stanford (CA, EUA) y otros equipos de colaboradores, examinaron la manera de identificar formas de contrarrestar los efectos de la disminución de las bacterias comensales que viven en el intestino después del tratamiento antibiótico. Ellos investigaron por qué dos patógenos potencialmente mortales pueden conseguir un equilibrio en el medio ambiente prohibitivo del intestino después de un tratamiento con antibióticos.
Dos agentes patógenos asociados a los antibióticos, Salmonella enterica serovar Typhimurium (S. typhimurium) y Clostridium difficile utilizan una estrategia común para catabolisar los carbohidratos liberados por la microbiota de las mucosas, durante su expansión en el intestino. S. typhimurium accede a la fucosa y el ácido siálico dentro del lumen intestinal de una manera dependiente de la microbiota, y la ablación genética de las respectivas vías catabólicas reduce su competitividad in vivo. Del mismo modo, la expansión de C. difficile es ayudada por la elevación inducida por la microbiota de los niveles de ácido siálico in vivo.
Tan pronto como esta pareja de invasores parásitos se multiplican a un número suficiente, inducen la inflamación. Aunque la inflamación no es un buen ambiente para la restauración de las bacterias buenas, C. difficile y S. typhimurium se desarrollan en él. Los científicos introdujeron una única cepa bacteriana Bacteroides thetaiotaomicron en animales libres de gérmenes. Esta cepa bacteriana amigable reside en el intestino de los ratones y los seres humanos normales. B. thetaiotaomicron tiene enzimas que liberan moléculas de azúcar de las cadenas de moco que cuelgan de la pared intestinal, pero carece de las enzimas para descomponer las moléculas que componen el ácido siálico. Sin embargo, en un intestino normal, hay varios otros microorganismos que pueden descomponer las moléculas de ácido siálico y fucosa.
Justin Sonnenburg, PhD, el autor principal del estudio, dijo: “Los chicos malos en el intestino están aprovechando la basura de los nutrientes que son liberados por los chicos buenos, que son víctimas de los daños colaterales debido a los antibióticos. Los antibióticos hacen que nuestras bacterias intestinales amigables ayuden inconscientemente a estos patógenos. Nuestro trabajo muestra cómo van tras ellos después de una dosis de antibióticos. Se aprovechan de un alza temporal en los azúcares disponibles liberados del mucus intestinal dejados por los microbios comensales muertos”.
El equipo llegó a la conclusión de que se podría crear algún día un medicamento que inhiba las enzimas utilizadas por las bacterias intestinales amigables para liberar el ácido siálico de la mucosidad, privando así a los patógenos de los alimentos. El medicamento podría entonces darse junto con antibióticos. Añadieron que los probióticos en forma de cepas bacterianas que digieren el ácido siálico rápidamente también podrían lograr un efecto similar. El estudio fue publicado el 1 de septiembre de 2013, en la revista Nature.
Enlace relacionado:
Stanford University